Viren
| Virus ⓘ | |
|---|---|

| |
| SARS-CoV-2, ein Mitglied der Unterfamilie Coronavirinae | |
| Klassifizierung des Virus | |
| (ohne Rangfolge): | Virus |
| Reiche | |
| |
Ein Virus ist ein submikroskopisches infektiöses Agens, das sich nur innerhalb der lebenden Zellen eines Organismus vermehrt. Viren infizieren alle Lebensformen, von Tieren und Pflanzen bis hin zu Mikroorganismen, einschließlich Bakterien und Archaeen. Seit Dmitri Iwanowskis Artikel aus dem Jahr 1892, in dem er einen nichtbakteriellen Erreger beschrieb, der Tabakpflanzen infiziert, und der Entdeckung des Tabakmosaikvirus durch Martinus Beijerinck im Jahr 1898 wurden von den Millionen von Virustypen in der Umwelt mehr als 9.000 Virusarten detailliert beschrieben. Viren kommen in fast allen Ökosystemen der Erde vor und sind die zahlreichste Art biologischer Einheiten. Das Studium der Viren wird als Virologie bezeichnet, ein Teilgebiet der Mikrobiologie. ⓘ
Wenn eine Wirtszelle infiziert wird, ist sie oft gezwungen, schnell Tausende von Kopien des ursprünglichen Virus zu produzieren. Wenn sie sich nicht in einer infizierten Zelle befinden oder gerade dabei sind, eine Zelle zu infizieren, existieren Viren in Form unabhängiger Partikel oder Virionen, bestehend aus (i) dem genetischen Material, d. h. langen DNA- oder RNA-Molekülen, die die Struktur der Proteine kodieren, durch die das Virus wirkt; (ii) einer Proteinhülle, dem Kapsid, das das genetische Material umgibt und schützt; und in einigen Fällen (iii) einer äußeren Hülle aus Lipiden. Die Formen dieser Viruspartikel reichen von einfachen helikalen und ikosaedrischen Formen bis hin zu komplexeren Strukturen. Die meisten Virusarten haben Virionen, die zu klein sind, um mit einem Lichtmikroskop gesehen zu werden, und die nur ein Hundertstel so groß sind wie die meisten Bakterien. ⓘ
Die Ursprünge der Viren in der Evolutionsgeschichte des Lebens sind unklar: Einige könnten sich aus Plasmiden entwickelt haben - DNA-Stücke, die sich zwischen Zellen bewegen können -, während andere sich aus Bakterien entwickelt haben könnten. In der Evolution sind Viren ein wichtiges Mittel für den horizontalen Gentransfer, der die genetische Vielfalt in ähnlicher Weise wie die sexuelle Fortpflanzung erhöht. Einige Biologen betrachten Viren als eine Lebensform, da sie genetisches Material tragen, sich fortpflanzen und sich durch natürliche Auslese weiterentwickeln, obwohl ihnen die Schlüsselmerkmale, wie z. B. die Zellstruktur, fehlen, die allgemein als notwendige Kriterien für die Definition von Leben angesehen werden. Da sie einige, aber nicht alle dieser Eigenschaften besitzen, wurden Viren als "Organismen am Rande des Lebens" und als Replikatoren bezeichnet. ⓘ
Viren verbreiten sich auf viele Arten. Ein Übertragungsweg führt über krankheitstragende Organismen, die als Vektoren bekannt sind: So werden Viren häufig von Insekten, die sich von Pflanzensaft ernähren, wie z. B. Blattläusen, von Pflanze zu Pflanze übertragen; und Viren bei Tieren können durch blutsaugende Insekten übertragen werden. Viele Viren, darunter Influenzaviren, SARS-CoV-2, Windpocken, Pocken und Masern, werden durch Husten und Niesen in der Luft verbreitet. Norovirus und Rotavirus, häufige Erreger viraler Gastroenteritis, werden über den fäkal-oralen Weg durch Hand-zu-Mund-Kontakt oder über Lebensmittel oder Wasser übertragen. Die infektiöse Dosis des Norovirus, die erforderlich ist, um eine Infektion beim Menschen auszulösen, beträgt weniger als 100 Partikel. HIV ist eines von mehreren Viren, die durch sexuelle Kontakte und durch den Kontakt mit infiziertem Blut übertragen werden. Die Vielfalt der Wirtszellen, die ein Virus infizieren kann, wird als sein "Wirtsspektrum" bezeichnet. Sie kann eng sein, was bedeutet, dass ein Virus nur wenige Arten infizieren kann, oder weit, was bedeutet, dass es viele Arten infizieren kann. ⓘ
Virusinfektionen bei Tieren lösen eine Immunreaktion aus, die in der Regel das infizierende Virus eliminiert. Immunreaktionen können auch durch Impfstoffe hervorgerufen werden, die eine künstlich erworbene Immunität gegen die spezifische Virusinfektion verleihen. Einige Viren, darunter die Erreger von HIV/AIDS, HPV-Infektionen und viraler Hepatitis, entziehen sich diesen Immunreaktionen und führen zu chronischen Infektionen. Es wurden mehrere Klassen von antiviralen Medikamenten entwickelt. ⓘ


2011 waren etwa 1,8 Millionen verschiedene rezente Arten von Lebewesen bekannt, die als Wirte für Viren fungieren, jedoch lediglich etwa 9110 Virenarten. Modellrechnungen zeigen jedoch, dass die Anzahl von Virenarten wahrscheinlich noch viel größer ist. So wurde 2013 berichtet, dass die Säugetiere alleine mindestens 320.000 noch unentdeckte Virenarten beherbergen. Da die Anzahl der Säugetierarten im Vergleich zu anderen Taxa winzig klein ist (lediglich rund 6500 Säugerarten, aber eine Million bekannte Arten von Insekten), kann von einer noch viel größeren Anzahl Virenarten ausgegangen werden. Da das Augenmerk der Virologie auf den Arten liegt, welche für die Humanmedizin, die Nutztiermedizin sowie für die Landwirtschaft bedeutsam sind, macht die offizielle Beschreibung und Benennung neuer Virenarten allerdings nur langsam Fortschritte. ⓘ
Viren befallen Zellen von Eukaryoten (Pflanzen, Pilze und Tiere einschließlich des Menschen) sowie von Prokaryoten (Bakterien und Archaeen). Viren, die Prokaryoten als Wirte nutzen, werden Bakteriophagen genannt; für Viren, die speziell Archaeen befallen, wird teilweise aber auch die Bezeichnung Archaeophagen verwendet. ⓘ
Die Wissenschaft, die sich mit Viren und Virusinfektionen beschäftigt, wird als Virologie bezeichnet. ⓘ
Etymologie
Das Wort stammt aus dem lateinischen Neutrum vīrus, das sich auf Gift und andere schädliche Flüssigkeiten bezieht, von der gleichen indoeuropäischen Basis wie Sanskrit viṣa, Avestan vīša und Altgriechisch ἰός (alle mit der Bedeutung "Gift"), das erstmals 1398 in John Trevisas Übersetzung von Bartholomeus Anglicus' De Proprietatibus Rerum im Englischen belegt ist. Virulent, von lateinisch virulentus ('giftig'), stammt aus der Zeit um 1400. Die Bedeutung "Erreger von Infektionskrankheiten" wird erstmals 1728 erwähnt, also lange vor der Entdeckung von Viren durch Dmitri Iwanowsky im Jahr 1892. Der englische Plural lautet viruses (manchmal auch vira), während das lateinische Wort ein Massensubstantiv ist, das keinen klassisch belegten Plural hat (vīra wird im Neulateinischen verwendet). Das Adjektiv viral stammt aus dem Jahr 1948. Der Begriff Virion (Plural Virionen) stammt aus dem Jahr 1959 und bezieht sich auf ein einzelnes Viruspartikel, das aus einer Zelle freigesetzt wird und andere Zellen desselben Typs infizieren kann. ⓘ
Ursprünge
Viren kommen überall dort vor, wo es Leben gibt, und existieren wahrscheinlich schon seit der Entstehung lebender Zellen. Der Ursprung von Viren ist unklar, da sie keine Fossilien bilden. Daher wird mit Hilfe molekularer Techniken untersucht, wie sie entstanden sind. Darüber hinaus integriert sich virales Erbgut gelegentlich in die Keimbahn der Wirtsorganismen, wodurch es über viele Generationen hinweg vertikal an die Nachkommen des Wirts weitergegeben werden kann. Dies stellt eine unschätzbare Informationsquelle für Paläovirologen dar, um alte Viren zurückzuverfolgen, die bis zu Millionen von Jahren zurückliegen. Es gibt drei Haupthypothesen, die den Ursprung der Viren erklären sollen:
- Regressionshypothese
- Viren könnten einst kleine Zellen gewesen sein, die in größeren Zellen parasitiert haben. Im Laufe der Zeit gingen Gene verloren, die sie für ihren Parasitismus nicht benötigten. Die Bakterien Rickettsien und Chlamydien sind lebende Zellen, die sich wie Viren nur innerhalb von Wirtszellen vermehren können. Sie unterstützen diese Hypothese, da ihre Abhängigkeit vom Parasitismus wahrscheinlich den Verlust von Genen verursacht hat, die ihnen das Überleben außerhalb einer Zelle ermöglichten. Dies wird auch als "Entartungshypothese" oder "Reduktionshypothese" bezeichnet.
- Hypothese des zellulären Ursprungs
- Einige Viren könnten sich aus DNA- oder RNA-Abschnitten entwickelt haben, die aus den Genen eines größeren Organismus "entkommen" sind. Die entkommene DNA könnte von Plasmiden (nackte DNA-Stücke, die zwischen Zellen wandern können) oder Transposons (DNA-Moleküle, die sich replizieren und an verschiedene Positionen innerhalb der Gene der Zelle wandern) stammen. Transposons, die früher als "springende Gene" bezeichnet wurden, sind Beispiele für mobile genetische Elemente und könnten der Ursprung einiger Viren sein. Sie wurden 1950 von Barbara McClintock in Mais entdeckt. Diese Hypothese wird manchmal auch als "Vagrancy-Hypothese" oder "Escape-Hypothese" bezeichnet.
- Ko-Evolutions-Hypothese
- Diese Hypothese wird auch als "Virus-First-Hypothese" bezeichnet und besagt, dass sich Viren aus komplexen Protein- und Nukleinsäuremolekülen zur gleichen Zeit entwickelt haben könnten, als die ersten Zellen auf der Erde auftauchten, und dass sie über Milliarden von Jahren vom zellulären Leben abhängig waren. Viroide sind RNA-Moleküle, die nicht als Viren eingestuft werden, weil ihnen eine Proteinhülle fehlt. Sie weisen Merkmale auf, die mehreren Viren gemeinsam sind, und werden oft als subvirale Erreger bezeichnet. Viroide sind wichtige Krankheitserreger von Pflanzen. Sie kodieren nicht für Proteine, sondern interagieren mit der Wirtszelle und nutzen die Wirtsmaschinerie für ihre Replikation. Das Hepatitis-Delta-Virus des Menschen hat ein den Viroiden ähnliches RNA-Genom, besitzt aber eine vom Hepatitis-B-Virus abgeleitete Proteinhülle und kann keine eigene produzieren. Es handelt sich also um ein defektes Virus. Obwohl sich das Genom des Hepatitis-Delta-Virus in einer Wirtszelle eigenständig replizieren kann, benötigt es die Hilfe des Hepatitis-B-Virus, um eine Proteinhülle zu bilden, damit es auf neue Zellen übertragen werden kann. In ähnlicher Weise ist der Sputnik-Virophage auf das Mimivirus angewiesen, das den Protozoen Acanthamoeba castellanii infiziert. Diese Viren, die auf das Vorhandensein anderer Virusarten in der Wirtszelle angewiesen sind, werden als "Satelliten" bezeichnet und stellen möglicherweise evolutionäre Zwischenstufen von Viroiden und Viren dar. ⓘ
In der Vergangenheit gab es Probleme mit all diesen Hypothesen: Die Regressionshypothese erklärte nicht, warum selbst die kleinsten zellulären Parasiten den Viren in keiner Weise ähneln. Die Escape-Hypothese erklärte nicht die komplexen Kapside und andere Strukturen auf Viruspartikeln. Die Virus-First-Hypothese widersprach der Definition von Viren, da diese Wirtszellen benötigen. Heute weiß man, dass Viren sehr alt sind und dass ihre Ursprünge vor der Aufspaltung des Lebens in die drei Domänen liegen. Diese Entdeckung hat die modernen Virologen dazu veranlasst, diese drei klassischen Hypothesen zu überdenken und neu zu bewerten. ⓘ
Die Beweise für eine Urwelt von RNA-Zellen und die Computeranalyse von Virus- und Wirts-DNA-Sequenzen führen zu einem besseren Verständnis der evolutionären Beziehungen zwischen den verschiedenen Viren und können dazu beitragen, die Vorfahren der modernen Viren zu identifizieren. Bislang haben solche Analysen nicht bewiesen, welche dieser Hypothesen richtig ist. Es scheint unwahrscheinlich, dass alle derzeit bekannten Viren einen gemeinsamen Vorfahren haben, und die Viren sind in der Vergangenheit wahrscheinlich durch einen oder mehrere Mechanismen mehrfach entstanden. ⓘ
Mikrobiologie
Für eine Evolution eines Virus (bzw. irgendeines Gens) ist seine Variabilität und Selektion von Bedeutung. Die Variabilität ist (wie bei allen Organismen) durch Kopierfehler bei der Replikation des Erbgutes gegeben und dient unter anderem der Immunevasion und der Änderung des Wirtsspektrums, während die Selektion oft durch die (Immun)-Antwort des Wirtes durchgeführt wird. ⓘ
Höher organisierte Lebewesen haben per Rekombination und Crossing-over bei der geschlechtlichen Fortpflanzung eine sehr effektive Möglichkeit der genetischen Variabilität besonders in Richtung einer Umweltanpassung und damit Weiterentwicklung ihrer jeweiligen Art entwickelt. Virionen beziehungsweise Viren zeigen als überdauerungsfähige Strukturen, die für ihre Vermehrung und damit auch Ausbreitung auf lebende Wirte angewiesen sind, ohne geschlechtliche Fortpflanzung allein mit ihrer Mutationsfähigkeit eine mindestens ebenbürtige Möglichkeit für eine genetische Variabilität. ⓘ
Dabei ist es dann letztlich unerheblich, dass diese Mutationen im Genom der Viren im Grunde zuerst auf Kopierfehlern während der Replikation innerhalb der Wirtszellen beruhen. Was zählt, ist allein der daraus für die Arterhaltung resultierende positive Effekt der extremen Steigerung der Anpassungsfähigkeit. Während Fehler dieser Art zum Beispiel bei einer hochentwickelten Säugetierzelle zum Zelltod führen können, beinhalten sie für Viren sogar einen großen Selektionsvorteil. ⓘ
Kopierfehler bei der Replikation drücken sich in Punktmutationen, also im Einbau von falschen Basen an zufälligen Genorten, aus. Da Viren im Gegensatz zu den höherentwickelten Zellen nur über wenige oder keine Reparaturmechanismen verfügen, werden diese Fehler nicht korrigiert. ⓘ
Sonderformen der genetischen Veränderung bei Viren werden beispielsweise bei den Influenza-Viren mit den Begriffen Antigendrift und Antigenshift (genetische Reassortierung) dort genau beschrieben. ⓘ
Eigenschaften des Lebens
In der Wissenschaft gehen die Meinungen darüber auseinander, ob Viren eine Form von Leben oder organische Strukturen sind, die mit lebenden Organismen interagieren. Sie wurden als "Organismen am Rande des Lebens" bezeichnet, da sie Organismen insofern ähneln, als sie Gene besitzen, sich durch natürliche Selektion weiterentwickeln und sich reproduzieren, indem sie mehrere Kopien von sich selbst durch Selbstorganisation erstellen. Obwohl sie Gene besitzen, haben sie keine zelluläre Struktur, die oft als Grundeinheit des Lebens angesehen wird. Viren haben keinen eigenen Stoffwechsel und benötigen eine Wirtszelle, um neue Produkte herzustellen. Daher können sie sich außerhalb einer Wirtszelle nicht natürlich vermehren - obwohl einige Bakterien wie Rickettsien und Chlamydien trotz dieser Einschränkung als lebende Organismen gelten. Anerkannte Lebensformen reproduzieren sich durch Zellteilung, während Viren sich spontan in Zellen zusammensetzen. Sie unterscheiden sich vom autonomen Wachstum von Kristallen, da sie genetische Mutationen erben und gleichzeitig der natürlichen Selektion unterliegen. Die Selbstorganisation von Viren innerhalb von Wirtszellen hat Auswirkungen auf die Erforschung des Ursprungs des Lebens, da sie die Hypothese untermauert, dass das Leben als selbstorganisierende organische Moleküle entstanden sein könnte. ⓘ
Struktur
Viren weisen eine große Vielfalt an Größen und Formen auf, die als "Morphologien" bezeichnet werden. Im Allgemeinen sind Viren viel kleiner als Bakterien, und mehr als tausend Bakteriophagenviren würden in die Zelle eines Escherichia coli-Bakteriums passen. Viele untersuchte Viren sind kugelförmig und haben einen Durchmesser zwischen 20 und 300 Nanometern. Einige Filoviren, bei denen es sich um Fäden handelt, haben eine Gesamtlänge von bis zu 1400 nm; ihr Durchmesser beträgt nur etwa 80 nm. Die meisten Viren lassen sich mit einem Lichtmikroskop nicht erkennen, daher werden sie mit einem Raster- und Transmissionselektronenmikroskop sichtbar gemacht. Um den Kontrast zwischen den Viren und dem Hintergrund zu erhöhen, werden elektronendichte "Färbungen" verwendet. Dabei handelt es sich um Lösungen von Schwermetallsalzen, wie z. B. Wolfram, die die Elektronen aus den mit dem Farbstoff bedeckten Bereichen streuen. Wenn die Virionen mit dem Farbstoff bedeckt sind (positive Färbung), werden feine Details verdeckt. Bei der Negativfärbung wird dieses Problem umgangen, indem nur der Hintergrund gefärbt wird. ⓘ
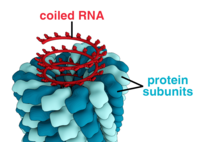
Ein komplettes Viruspartikel, ein so genanntes Virion, besteht aus Nukleinsäure, die von einer schützenden Proteinhülle, dem so genannten Kapsid, umgeben ist. Diese werden aus Proteinuntereinheiten, den sogenannten Kapsomeren, gebildet. Viren können eine von der Wirtszellmembran abgeleitete Lipid-"Hülle" haben. Das Kapsid wird aus Proteinen gebildet, die vom viralen Genom kodiert werden, und seine Form dient als Grundlage für die morphologische Unterscheidung. Die vom Virus kodierten Proteinuntereinheiten fügen sich selbst zu einem Kapsid zusammen, wofür im Allgemeinen das Vorhandensein des Virusgenoms erforderlich ist. Komplexe Viren kodieren für Proteine, die bei der Konstruktion ihres Kapsids helfen. Proteine, die mit Nukleinsäure assoziiert sind, werden als Nukleoproteine bezeichnet, und die Verbindung von viralen Kapsidproteinen mit viraler Nukleinsäure wird als Nukleokapsid bezeichnet. Das Kapsid und die gesamte Virusstruktur können mit Hilfe der Rasterkraftmikroskopie mechanisch (physikalisch) untersucht werden. Im Allgemeinen gibt es fünf morphologische Haupttypen von Viren:
- Helikale
- Diese Viren bestehen aus einer einzigen Art von Kapsomeren, die um eine zentrale Achse herum gestapelt sind und eine spiralförmige Struktur bilden, die einen zentralen Hohlraum oder eine Röhre haben kann. Diese Anordnung führt zu Virionen, die kurze und sehr starre Stäbchen oder lange und sehr flexible Filamente sein können. Das genetische Material (in der Regel einzelsträngige RNA, in einigen Fällen aber auch einzelsträngige DNA) wird durch Wechselwirkungen zwischen der negativ geladenen Nukleinsäure und den positiven Ladungen des Proteins in die Proteinhelix eingebunden. Insgesamt hängt die Länge eines schraubenförmigen Kapsids von der Länge der darin enthaltenen Nukleinsäure ab, und der Durchmesser ist abhängig von der Größe und Anordnung der Kapsomere. Das gut untersuchte Tabakmosaikvirus und das Inovirus sind Beispiele für schraubenförmige Viren. ⓘ
- Ikosaedrisch
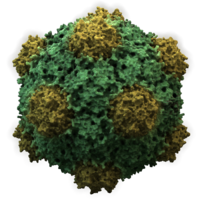
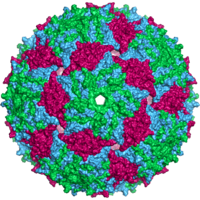
- Die meisten tierischen Viren sind ikosaedrisch oder nahezu kugelförmig mit chiraler ikosaedrischer Symmetrie. Ein regelmäßiges Ikosaeder ist der optimale Weg, um eine geschlossene Hülle aus identischen Untereinheiten zu bilden. Für jede Dreiecksfläche sind mindestens 3 Kapsomere erforderlich, was für das Ikosaeder 60 ergibt. Viele Viren, wie z. B. Rotaviren, haben mehr als 60 Capsomere und erscheinen kugelförmig, behalten aber diese Symmetrie bei. Um dies zu erreichen, sind die Capsomere an den Spitzen von fünf anderen Capsomeren umgeben und werden Pentone genannt. Die Kapsomere an den Dreiecksflächen sind von sechs weiteren Kapsomeren umgeben und werden als Hexon bezeichnet. Hexons sind im Wesentlichen flach, während Pentons, die die 12 Scheitelpunkte bilden, gekrümmt sind. Ein und dasselbe Protein kann als Untereinheit sowohl der Pentamere als auch der Hexamere fungieren, oder sie können aus verschiedenen Proteinen zusammengesetzt sein. ⓘ
- Prolate
- Hierbei handelt es sich um ein Ikosaeder, das entlang der fünffachen Achse verlängert ist, und eine häufige Anordnung der Köpfe von Bakteriophagen. Diese Struktur besteht aus einem Zylinder mit einer Kappe an beiden Enden. ⓘ
- Umhüllte
- Einige Virusarten hüllen sich in eine modifizierte Form einer der Zellmembranen ein, entweder in die äußere Membran, die eine infizierte Wirtszelle umgibt, oder in interne Membranen wie die Kernmembran oder das endoplasmatische Retikulum, wodurch sie eine äußere Lipiddoppelschicht erhalten, die als Virushülle bezeichnet wird. Diese Membran ist mit Proteinen besetzt, für die das virale Genom und das Wirtsgenom kodieren; die Lipidmembran selbst und alle vorhandenen Kohlenhydrate stammen ausschließlich vom Wirt. Das Influenzavirus, HIV (AIDS-Erreger) und das Coronavirus 2 des schweren akuten Respirationssyndroms (Erreger von COVID-19) verwenden diese Strategie. Die meisten behüllten Viren sind für ihre Infektiosität von der Hülle abhängig. ⓘ
- Komplexe
- Diese Viren besitzen ein Kapsid, das weder rein helikal noch rein ikosaedrisch ist und das zusätzliche Strukturen wie Proteinschwänze oder eine komplexe Außenwand aufweisen kann. Einige Bakteriophagen, wie z. B. der Enterobakterien-Phagen T4, haben eine komplexe Struktur, die aus einem ikosaedrischen Kopf besteht, der an einen spiralförmigen Schwanz gebunden ist, der eine sechseckige Grundplatte mit abstehenden Proteinschwanzfasern haben kann. Diese Schwanzstruktur wirkt wie eine molekulare Spritze, die sich an den bakteriellen Wirt anheftet und dann das virale Genom in die Zelle injiziert. ⓘ
Die Pockenviren sind große, komplexe Viren, die eine ungewöhnliche Morphologie aufweisen. Das virale Genom ist mit Proteinen innerhalb einer zentralen Scheibenstruktur, dem so genannten Nukleoid, verbunden. Das Nukleoid ist von einer Membran und zwei Seitenkörpern mit unbekannter Funktion umgeben. Das Virus hat eine äußere Hülle mit einer dicken Proteinschicht, die die Oberfläche bedeckt. Das gesamte Virion ist leicht pleomorph und reicht von eiförmig bis ziegelsteinförmig. ⓘ

Das Proteinkapsid kann unterschiedliche Formen haben, zum Beispiel ikosaederförmig, isometrisch, helikal oder geschossförmig. ⓘ
Serologisch unterscheidbare Variationen eines Virus nennt man Serotypen. ⓘ
Virionen dienen der Verbreitung der Viren. Sie dringen ganz oder teilweise (mindestens ihre Nukleinsäure) in die Wirtszellen ein (infizieren sie). Danach startet durch den Stoffwechsel des Wirts die Vermehrung der Virus-Nukleinsäure und die Produktion der anderen Virionen-Bestandteile. ⓘ
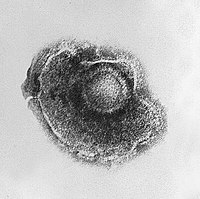
3D-Grafik des SARS-CoV-2-Virions (eingefärbtes Modell) ⓘ
Riesige Viren
Das Mimivirus ist eines der größten charakterisierten Viren mit einem Kapsiddurchmesser von 400 nm. Aus der Oberfläche ragen Proteinfilamente mit einer Größe von 100 nm heraus. Das Kapsid erscheint unter dem Elektronenmikroskop sechseckig, daher ist das Kapsid wahrscheinlich ikosaedrisch. Im Jahr 2011 entdeckten Forscher in Wasserproben, die vom Meeresboden vor der Küste von Las Cruces, Chile, gesammelt wurden, das größte damals bekannte Virus. Es wurde vorläufig Megavirus chilensis genannt und kann mit einem einfachen Lichtmikroskop erkannt werden. Im Jahr 2013 wurde in Chile und Australien die Gattung Pandoravirus entdeckt, deren Genom etwa doppelt so groß ist wie das von Megavirus und Mimivirus. Alle Riesenviren haben dsDNA-Genome und werden in mehrere Familien eingeteilt: Mimiviridae, Pithoviridae, Pandoraviridae, Phycodnaviridae und die Gattung Mollivirus. ⓘ
Einige Viren, die Archaeen infizieren, weisen komplexe Strukturen auf, die mit keiner anderen Form von Viren vergleichbar sind, mit einer Vielzahl von ungewöhnlichen Formen, die von spindelförmigen Strukturen bis hin zu Viren reichen, die hakenförmigen Stäben, Tränen oder sogar Flaschen ähneln. Andere Archaeenviren ähneln den schwanzförmigen Bakteriophagen und können mehrere Schwanzstrukturen aufweisen. ⓘ
Genom
| Eigenschaft | Parameter ⓘ |
|---|---|
| Nukleinsäure |
|
| Form |
|
| Verseiltheit |
|
| Sense |
|
Viren weisen eine enorme Vielfalt an genomischen Strukturen auf; als Gruppe enthalten sie mehr strukturelle genomische Vielfalt als Pflanzen, Tiere, Archaeen oder Bakterien. Es gibt Millionen verschiedener Virustypen, obwohl weniger als 7.000 Typen im Detail beschrieben sind. Mit Stand vom Januar 2021 enthält die Virusgenom-Datenbank des NCBI mehr als 193.000 vollständige Genomsequenzen, aber es gibt zweifellos noch viele weitere zu entdecken. ⓘ
Ein Virus hat entweder ein DNA- oder ein RNA-Genom und wird als DNA-Virus bzw. als RNA-Virus bezeichnet. Die überwiegende Mehrheit der Viren hat ein RNA-Genom. Pflanzenviren haben in der Regel einzelsträngige RNA-Genome und Bakteriophagen haben meist doppelsträngige DNA-Genome. ⓘ
Virale Genome sind zirkulär, wie bei den Polyomaviren, oder linear, wie bei den Adenoviren. Die Art der Nukleinsäure ist für die Form des Genoms unerheblich. Bei RNA-Viren und bestimmten DNA-Viren ist das Genom oft in einzelne Teile aufgeteilt und wird in diesem Fall als segmentiert bezeichnet. Bei RNA-Viren kodiert jedes Segment oft nur für ein einziges Protein, und sie befinden sich in der Regel zusammen in einem Kapsid. Es müssen nicht alle Segmente im selben Virion vorhanden sein, damit das Virus infektiös ist, wie das Brommosaikvirus und verschiedene andere Pflanzenviren zeigen. ⓘ
Ein virales Genom ist, unabhängig vom Nukleinsäuretyp, fast immer entweder einzelsträngig (ss) oder doppelsträngig (ds). Einzelsträngige Genome bestehen aus einer ungepaarten Nukleinsäure, analog zu einer in der Mitte gespaltenen Leiterhälfte. Doppelstrangige Genome bestehen aus zwei komplementären gepaarten Nukleinsäuren, analog zu einer Leiter. Die Viruspartikel einiger Virusfamilien, z. B. die der Hepadnaviridae, enthalten ein Genom, das teilweise doppelsträngig und teilweise einzelsträngig ist. ⓘ
Bei den meisten Viren mit RNA-Genomen und einigen mit einzelsträngigen DNA (ssDNA)-Genomen werden die Einzelstränge entweder als positiv-sinnig (als "Plus-Strang" bezeichnet) oder negativ-sinnig (als "Minus-Strang" bezeichnet) bezeichnet, je nachdem, ob sie komplementär zur viralen Boten-RNA (mRNA) sind. Die virale RNA mit positivem Sinn ist im gleichen Sinne wie die virale mRNA, so dass zumindest ein Teil von ihr von der Wirtszelle sofort übersetzt werden kann. Die virale RNA mit negativem Sinn ist komplementär zur mRNA und muss daher vor der Translation durch eine RNA-abhängige RNA-Polymerase in RNA mit positivem Sinn umgewandelt werden. Die DNA-Nomenklatur für Viren mit genomischer ssDNA ähnelt der RNA-Nomenklatur, da die virale ssDNA mit positivem Sinn in ihrer Sequenz mit der viralen mRNA identisch und somit ein kodierender Strang ist, während die virale ssDNA mit negativem Sinn komplementär zur viralen mRNA ist und somit ein Vorlagenstrang. Mehrere Arten von ssDNA- und ssRNA-Viren haben Genome, die ambisense sind, d. h. die Transkription kann von beiden Strängen in einem doppelsträngigen replikativen Zwischenprodukt erfolgen. Beispiele hierfür sind Geminiviren, ssDNA-Pflanzenviren, und Arenaviren, ssRNA-Viren von Tieren. ⓘ
Genomgröße
Die Genomgröße variiert stark zwischen den Arten. Die kleinsten - die ssDNA-Circoviren, Familie Circoviridae - kodieren für nur zwei Proteine und haben eine Genomgröße von nur zwei Kilobasen; die größten - die Pandoraviren - haben eine Genomgröße von etwa zwei Megabasen und kodieren für etwa 2500 Proteine. Virusgene haben selten Introns und sind oft so im Genom angeordnet, dass sie sich überlappen. ⓘ
Im Allgemeinen haben RNA-Viren aufgrund einer höheren Fehlerquote bei der Replikation eine geringere Genomgröße als DNA-Viren und haben eine maximale Obergrenze für die Größe. Darüber hinaus machen Fehler bei der Replikation das Virus unbrauchbar oder nicht konkurrenzfähig. Um dies zu kompensieren, haben RNA-Viren oft segmentierte Genome - das Genom ist in kleinere Moleküle aufgeteilt - und verringern so die Wahrscheinlichkeit, dass ein Fehler in einem einzelnen Genombestandteil das gesamte Genom unbrauchbar macht. Im Gegensatz dazu haben DNA-Viren aufgrund der hohen Zuverlässigkeit ihrer Replikationsenzyme im Allgemeinen größere Genome. Einzelstrang-DNA-Viren bilden eine Ausnahme von dieser Regel, da die Mutationsraten für diese Genome dem Extremfall der ssRNA-Viren nahe kommen können. ⓘ
Genetische Mutation
Viren unterliegen genetischen Veränderungen durch verschiedene Mechanismen. Dazu gehört ein Prozess, der als antigene Drift bezeichnet wird und bei dem einzelne Basen in der DNA oder RNA zu anderen Basen mutieren. Die meisten dieser Punktmutationen sind "stumm" - sie verändern nicht das Protein, für das das Gen kodiert -, aber andere können evolutionäre Vorteile wie Resistenz gegen antivirale Medikamente mit sich bringen. Eine Antigenverschiebung liegt vor, wenn das Genom des Virus stark verändert wird. Dies kann das Ergebnis einer Rekombination oder eines Reassortiments sein. Wenn dies bei Influenzaviren geschieht, kann es zu Pandemien kommen. RNA-Viren treten häufig als Quasispezies oder Schwärme von Viren derselben Art, aber mit leicht unterschiedlichen Genom-Nukleosidsequenzen auf. Solche Quasispezies sind ein bevorzugtes Ziel für die natürliche Selektion. ⓘ
Segmentierte Genome bringen evolutionäre Vorteile mit sich; verschiedene Stämme eines Virus mit segmentiertem Genom können Gene mischen und kombinieren und so Viren (oder Nachkommen) mit einzigartigen Eigenschaften hervorbringen. Dies wird als Reassortment oder "viraler Sex" bezeichnet. ⓘ
Genetische Rekombination ist der Prozess, bei dem ein DNA- (oder RNA-) Strang gebrochen und dann mit dem Ende eines anderen DNA- (oder RNA-) Moleküls verbunden wird. Dies kann vorkommen, wenn Viren Zellen gleichzeitig infizieren, und Untersuchungen der viralen Evolution haben gezeigt, dass die Rekombination bei den untersuchten Arten weit verbreitet ist. Die Rekombination ist sowohl bei RNA- als auch bei DNA-Viren üblich. ⓘ
Replikationszyklus
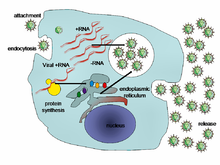
Viruspopulationen wachsen nicht durch Zellteilung, da sie azellulär sind. Stattdessen nutzen sie die Maschinerie und den Stoffwechsel einer Wirtszelle, um mehrere Kopien von sich selbst zu produzieren, die sich dann in der Zelle ansammeln. Bei einer Infektion ist die Wirtszelle gezwungen, schnell Tausende von Kopien des ursprünglichen Virus zu produzieren. ⓘ
Ihr Lebenszyklus ist von Art zu Art sehr unterschiedlich, aber es gibt sechs grundlegende Phasen in ihrem Lebenszyklus: Die Anheftung ist eine spezifische Bindung zwischen viralen Kapsidproteinen und spezifischen Rezeptoren auf der Oberfläche der Wirtszelle. Diese Spezifität bestimmt den Wirtsbereich und die Art der Wirtszelle eines Virus. So infiziert HIV beispielsweise nur eine begrenzte Anzahl menschlicher Leukozyten. Der Grund dafür ist, dass sein Oberflächenprotein gp120 spezifisch mit dem CD4-Molekül - einem Chemokinrezeptor - interagiert, der sich am häufigsten auf der Oberfläche von CD4+ T-Zellen befindet. Dieser Mechanismus hat sich so entwickelt, dass er diejenigen Viren begünstigt, die nur Zellen infizieren, in denen sie replikationsfähig sind. Die Anheftung an den Rezeptor kann dazu führen, dass das virale Hüllprotein Veränderungen erfährt, die zur Verschmelzung von viraler und zellulärer Membran führen, oder zu Veränderungen von nicht umhüllten Virusoberflächenproteinen, die das Eindringen des Virus ermöglichen. ⓘ
Die Penetration oder der Eintritt des Virus erfolgt nach der Anheftung: Die Viren dringen durch rezeptorvermittelte Endozytose oder Membranfusion in die Wirtszelle ein. Die Infektion von Pflanzen- und Pilzzellen unterscheidet sich von der von tierischen Zellen. Pflanzen haben eine starre Zellwand aus Zellulose und Pilze eine aus Chitin, so dass die meisten Viren nur nach einer Verletzung der Zellwand in diese Zellen gelangen können. Fast alle Pflanzenviren (z. B. das Tabakmosaikvirus) können sich auch direkt von Zelle zu Zelle bewegen, und zwar in Form von einzelsträngigen Nukleoproteinkomplexen durch Poren, die Plasmodesmata genannt werden. Bakterien haben wie Pflanzen starke Zellwände, die ein Virus durchbrechen muss, um die Zelle zu infizieren. Da die Zellwände von Bakterien aufgrund ihrer viel geringeren Größe viel dünner sind als die von Pflanzen, haben einige Viren Mechanismen entwickelt, die ihr Genom durch die Zellwand in die Bakterienzelle einschleusen, während das virale Kapsid außerhalb der Zelle bleibt. ⓘ
Uncoating ist ein Prozess, bei dem das virale Kapsid entfernt wird: Dies kann durch Abbau durch virale Enzyme oder Wirtsenzyme oder durch einfache Dissoziation geschehen; das Endergebnis ist die Freisetzung der viralen genomischen Nukleinsäure. ⓘ
Die Replikation von Viren beinhaltet in erster Linie die Vermehrung des Genoms. Die Replikation umfasst die Synthese viraler Boten-RNA (mRNA) aus "frühen" Genen (mit Ausnahmen von Viren mit Positiv-Sense-RNA), die virale Proteinsynthese, den möglichen Zusammenbau viraler Proteine und dann die virale Genomreplikation, die durch die Expression früher oder regulatorischer Proteine vermittelt wird. Bei komplexen Viren mit größeren Genomen können sich eine oder mehrere weitere Runden der mRNA-Synthese anschließen: Die "späte" Genexpression erfolgt im Allgemeinen für Struktur- oder Virionproteine. ⓘ
Zusammenbau - Nach dem strukturvermittelten Selbstzusammenbau der Viruspartikel kommt es häufig zu einer Modifizierung der Proteine. Bei Viren wie HIV erfolgt diese Modifikation (manchmal auch Reifung genannt), nachdem das Virus aus der Wirtszelle freigesetzt wurde. ⓘ
Freisetzung - Viren können durch Lyse aus der Wirtszelle freigesetzt werden, ein Prozess, bei dem die Zelle durch das Zerplatzen ihrer Membran und, falls vorhanden, ihrer Zellwand abgetötet wird: Dies ist ein Merkmal vieler bakterieller und einiger tierischer Viren. Einige Viren durchlaufen einen lysogenen Zyklus, bei dem das virale Genom durch genetische Rekombination an einer bestimmten Stelle in das Chromosom des Wirts eingebaut wird. Das virale Genom wird dann als "Provirus" oder, im Falle von Bakteriophagen, als "Prophage" bezeichnet. Immer wenn sich der Wirt teilt, wird auch das virale Genom repliziert. Das virale Genom ist im Wirt meist stumm. Irgendwann kann aus dem Provirus oder Prophagen das aktive Virus entstehen, das die Wirtszellen lysieren kann. Umhüllte Viren (z. B. HIV) werden normalerweise durch Knospung aus der Wirtszelle freigesetzt. Während dieses Prozesses erwirbt das Virus seine Hülle, die ein modifizierter Teil des Plasmas oder einer anderen internen Membran des Wirts ist. ⓘ
Replikation des Genoms
Das genetische Material in den Viruspartikeln und die Methode, mit der das Material repliziert wird, unterscheiden sich erheblich zwischen den verschiedenen Virustypen. ⓘ
- DNA-Viren
- Die Genomreplikation der meisten DNA-Viren findet im Zellkern statt. Wenn die Zelle den entsprechenden Rezeptor auf ihrer Oberfläche hat, gelangen diese Viren entweder durch direkte Fusion mit der Zellmembran (z. B. Herpesviren) oder - was häufiger der Fall ist - durch rezeptorvermittelte Endozytose in die Zelle. Die meisten DNA-Viren sind vollständig von der DNA- und RNA-Synthesemaschinerie und der RNA-Verarbeitungsmaschinerie der Wirtszelle abhängig. Viren mit größeren Genomen können einen Großteil dieser Maschinen selbst kodieren. Bei Eukaryonten muss das virale Genom die Kernmembran der Zelle durchqueren, um Zugang zu dieser Maschinerie zu erhalten, während es bei Bakterien nur in die Zelle eindringen muss. ⓘ
- RNA-Viren
- Die Replikation von RNA-Viren findet normalerweise im Zytoplasma statt. RNA-Viren können je nach Art ihrer Replikation in vier verschiedene Gruppen eingeteilt werden. Bei einzelsträngigen RNA-Viren bestimmt die Polarität (ob sie direkt von Ribosomen zur Herstellung von Proteinen verwendet werden kann oder nicht) weitgehend den Replikationsmechanismus; das andere wichtige Kriterium ist, ob das genetische Material einzelsträngig oder doppelsträngig ist. Alle RNA-Viren verwenden ihre eigenen RNA-Replikase-Enzyme, um Kopien ihrer Genome zu erstellen. ⓘ
- Revers transkribierende Viren
- Reverse transkribierende Viren haben ssRNA (Retroviridae, Metaviridae, Pseudoviridae) oder dsDNA (Caulimoviridae und Hepadnaviridae) in ihren Partikeln. Revers transkribierende Viren mit RNA-Genom (Retroviren) verwenden ein DNA-Zwischenprodukt zur Replikation, während Viren mit DNA-Genom (Pararetroviren) ein RNA-Zwischenprodukt während der Genomreplikation verwenden. Beide Typen verwenden eine reverse Transkriptase oder ein RNA-abhängiges DNA-Polymerase-Enzym, um die Nukleinsäureumwandlung durchzuführen. Retroviren integrieren die durch reverse Transkription erzeugte DNA als Provirus in das Wirtsgenom als Teil des Replikationsprozesses; Pararetroviren tun dies nicht, obwohl integrierte Genomkopien insbesondere pflanzlicher Pararetroviren zu infektiösen Viren führen können. Sie sind empfindlich gegenüber antiviralen Medikamenten, die das Enzym der reversen Transkriptase hemmen, z. B. Zidovudin und Lamivudin. Ein Beispiel für den ersten Typ ist HIV, ein Retrovirus. Beispiele für den zweiten Typ sind die Hepadnaviridae, zu denen das Hepatitis-B-Virus gehört. ⓘ
Zytopathische Wirkungen auf die Wirtszelle
Das Spektrum der strukturellen und biochemischen Wirkungen, die Viren auf die Wirtszelle haben, ist umfangreich. Diese werden als "zytopathische Wirkungen" bezeichnet. Die meisten Virusinfektionen führen schließlich zum Tod der Wirtszelle. Zu den Todesursachen gehören Zelllyse, Veränderungen der Zelloberflächenmembran und Apoptose. Häufig wird der Zelltod durch die Einstellung der normalen Zellaktivitäten aufgrund der Unterdrückung durch virusspezifische Proteine verursacht, von denen nicht alle Bestandteile des Viruspartikels sind. Die Unterscheidung zwischen zytopathisch und harmlos ist fließend. Einige Viren, wie z. B. das Epstein-Barr-Virus, können eine Zellvermehrung verursachen, ohne bösartig zu werden, während andere, wie z. B. Papillomviren, nachweislich Krebs verursachen. ⓘ
Schlummernde und latente Infektionen
Einige Viren verursachen keine offensichtlichen Veränderungen an der infizierten Zelle. Zellen, in denen das Virus latent und inaktiv ist, zeigen kaum Anzeichen einer Infektion und funktionieren oft normal. Dies führt zu hartnäckigen Infektionen, und das Virus schlummert oft über viele Monate oder Jahre. Dies ist häufig bei Herpesviren der Fall. ⓘ
Wirtsspektrum
Viren sind die bei weitem am häufigsten vorkommenden biologischen Einheiten auf der Erde und übertreffen alle anderen zusammengenommen. Sie infizieren alle Arten von zellulärem Leben, einschließlich Tieren, Pflanzen, Bakterien und Pilzen. Verschiedene Virustypen können nur eine begrenzte Anzahl von Wirten infizieren, und viele sind artspezifisch. Einige, wie z. B. das Pockenvirus, können nur eine Spezies infizieren - in diesem Fall den Menschen - und haben daher einen engen Wirtsbereich. Andere Viren, wie z. B. das Tollwutvirus, können verschiedene Säugetierarten infizieren und gelten als weit verbreitet. Die Viren, die Pflanzen befallen, sind für Tiere harmlos, und die meisten Viren, die andere Tiere befallen, sind für Menschen harmlos. Das Wirtsspektrum einiger Bakteriophagen ist auf einen einzigen Bakterienstamm beschränkt, und mit Hilfe einer Methode, die als Phagentypisierung bezeichnet wird, lässt sich die Quelle von Infektionsausbrüchen zurückverfolgen. Die Gesamtheit der Viren in einem Organismus oder Lebensraum wird als Virom bezeichnet; so bilden beispielsweise alle menschlichen Viren das menschliche Virom. ⓘ
Klassifizierung
Bei der Klassifizierung wird versucht, die Vielfalt der Viren zu beschreiben, indem sie auf der Grundlage von Ähnlichkeiten benannt und gruppiert werden. André Lwoff, Robert Horne und Paul Tournier waren 1962 die ersten, die ein System zur Klassifizierung von Viren entwickelten, das auf dem hierarchischen System von Linnaean basiert. Dieses System basiert auf der Einteilung in Stamm, Klasse, Ordnung, Familie, Gattung und Art. Die Viren wurden nach ihren gemeinsamen Eigenschaften (nicht nach denen ihrer Wirte) und nach der Art der Nukleinsäure, die ihr Genom bildet, gruppiert. 1966 wurde das International Committee on Taxonomy of Viruses (ICTV) gegründet. Das von Lwoff, Horne und Tournier vorgeschlagene System wurde vom ICTV zunächst nicht akzeptiert, da die geringe Genomgröße der Viren und ihre hohe Mutationsrate die Bestimmung ihrer Abstammung über die Reihenfolge hinaus erschwerten. Daher wurde das Baltimore-Klassifikationssystem als Ergänzung zur traditionellen Hierarchie verwendet. Ab 2018 hat das ICTV begonnen, tiefere evolutionäre Beziehungen zwischen Viren anzuerkennen, die im Laufe der Zeit entdeckt wurden, und ein 15-stufiges Klassifizierungssystem eingeführt, das vom Reich bis zur Art reicht. Darüber hinaus werden einige Arten innerhalb derselben Gattung zu einer Genogruppe zusammengefasst. ⓘ
ICTV-Klassifizierung
Das ICTV entwickelte das derzeitige Klassifizierungssystem und verfasste Richtlinien, in denen bestimmte Viruseigenschaften stärker gewichtet wurden, um die Einheitlichkeit der Familie zu wahren. Es wurde eine einheitliche Taxonomie (ein universelles System zur Klassifizierung von Viren) geschaffen. Nur ein kleiner Teil der gesamten Virenvielfalt ist erforscht. Bis 2021 hat das ICTV 6 Reiche, 10 Königreiche, 17 Phyla, 2 Subphyla, 39 Klassen, 65 Ordnungen, 8 Unterordnungen, 233 Familien, 168 Unterfamilien, 2.606 Gattungen, 84 Untergattungen und 10.434 Arten von Viren definiert. ⓘ
Die allgemeine taxonomische Struktur der Taxonreihen und die in den taxonomischen Namen verwendeten Suffixe werden im Folgenden dargestellt. Ab 2021 sind die Ränge Subrealm, Subkingdom und Subclass unbenutzt, während alle anderen Ränge in Gebrauch sind. ⓘ
- Reich (-viria)
- Unterreich (-vira)
- Königreich (-virae)
- Unterkönigreich (-virites)
- Stamm (-viricota)
- Unterstamm (-viricotina)
- Klasse (-viricetes)
- Unterklasse (-viricetidae)
- Ordnung (-virales)
- Unterordnung (-virineae)
- Familie (-viridae)
- Unterfamilie (-virinae)
- Gattung (-virus)
- Untergattung (-virus)
- Arten ⓘ
- Untergattung (-virus)
- Gattung (-virus)
- Unterfamilie (-virinae)
- Familie (-viridae)
- Unterordnung (-virineae)
- Ordnung (-virales)
- Unterklasse (-viricetidae)
- Klasse (-viricetes)
- Unterstamm (-viricotina)
- Stamm (-viricota)
- Unterkönigreich (-virites)
- Königreich (-virae)
- Unterreich (-vira)
Baltimore-Klassifikation
Der mit dem Nobelpreis ausgezeichnete Biologe David Baltimore hat das Baltimore-Klassifikationssystem entwickelt. Das ICTV-Klassifikationssystem wird in Verbindung mit dem Baltimore-Klassifikationssystem in der modernen Virusklassifikation verwendet. ⓘ
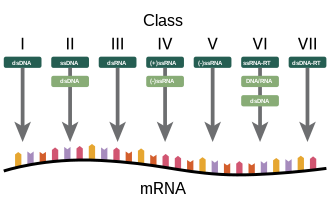
Die Baltimore-Klassifikation von Viren basiert auf dem Mechanismus der mRNA-Produktion. Viren müssen aus ihren Genomen mRNAs erzeugen, um Proteine zu produzieren und sich selbst zu replizieren, aber in jeder Virusfamilie werden dafür unterschiedliche Mechanismen verwendet. Virale Genome können einzelsträngig (ss) oder doppelsträngig (ds), RNA oder DNA sein und können eine reverse Transkriptase (RT) verwenden oder nicht. Darüber hinaus können ssRNA-Viren entweder Sense (+) oder Antisense (-) sein. Diese Klassifizierung teilt die Viren in sieben Gruppen ein:
- I: dsDNA-Viren (z. B. Adenoviren, Herpesviren, Pockenviren)
- II: ssDNA-Viren (+ Strang oder "Sense"-DNA) (z. B. Parvoviren)
- III: dsRNA-Viren (z. B. Reoviren)
- IV: (+)ssRNA-Viren (+ Strang oder "sense") RNA (z. B. Coronaviren, Picornaviren, Togaviren)
- V: (-)ssRNA-Viren (--Strang oder Antisense)-RNA (z. B. Orthomyxoviren, Rhabdoviren)
- VI: ssRNA-RT-Viren (+Strang oder sense) RNA mit DNA-Zwischenstufe im Lebenszyklus (z. B. Retroviren)
- VII: dsDNA-RT-Viren DNA mit RNA-Zwischenstufe im Lebenszyklus (z. B. Hepadnaviren) ⓘ
Rolle bei menschlichen Krankheiten

Beispiele für häufige, durch Viren verursachte Krankheiten beim Menschen sind Erkältung, Grippe, Windpocken und Fieberbläschen. Viele schwere Krankheiten wie Tollwut, Ebola-Virus, AIDS (HIV), Vogelgrippe und SARS werden durch Viren verursacht. Die relative Fähigkeit von Viren, Krankheiten zu verursachen, wird mit dem Begriff Virulenz beschrieben. Andere Krankheiten werden untersucht, um herauszufinden, ob sie von einem Virus verursacht werden, wie z. B. der mögliche Zusammenhang zwischen dem humanen Herpesvirus 6 (HHV6) und neurologischen Krankheiten wie Multipler Sklerose und chronischem Müdigkeitssyndrom. Es ist umstritten, ob das Bornavirus, von dem man bisher annahm, dass es neurologische Erkrankungen bei Pferden verursacht, auch für psychiatrische Erkrankungen beim Menschen verantwortlich sein könnte. ⓘ
Viren haben unterschiedliche Mechanismen, mit denen sie in einem Organismus Krankheiten auslösen, was weitgehend von der Virusspezies abhängt. Zu den Mechanismen auf zellulärer Ebene gehört in erster Linie die Zelllyse, das Aufbrechen und anschließende Absterben der Zelle. Wenn in mehrzelligen Organismen genügend Zellen absterben, werden die Auswirkungen auf den gesamten Organismus spürbar. Obwohl Viren eine Störung der gesunden Homöostase verursachen und zu Krankheiten führen, können sie in einem Organismus relativ harmlos existieren. Ein Beispiel dafür ist die Fähigkeit des Herpes-Simplex-Virus, das Fieberbläschen verursacht, im menschlichen Körper in einem Ruhezustand zu bleiben. Dies wird als Latenzzeit bezeichnet und ist ein Merkmal der Herpesviren, einschließlich des Epstein-Barr-Virus, das Drüsenfieber verursacht, und des Varizella-Zoster-Virus, das Windpocken und Gürtelrose verursacht. Die meisten Menschen haben sich mit mindestens einem dieser Herpesvirustypen infiziert. Diese latenten Viren können manchmal von Vorteil sein, da das Vorhandensein des Virus die Immunität gegen bakterielle Krankheitserreger, wie Yersinia pestis, erhöhen kann. ⓘ
Einige Viren können lebenslange oder chronische Infektionen verursachen, bei denen sich die Viren trotz der Abwehrmechanismen des Wirts weiterhin im Körper vermehren. Dies ist häufig bei Infektionen mit dem Hepatitis-B- und Hepatitis-C-Virus der Fall. Chronisch infizierte Menschen werden als Träger bezeichnet, da sie als Reservoir für infektiöse Viren dienen. In Populationen mit einem hohen Anteil an Virusträgern wird die Krankheit als endemisch bezeichnet. ⓘ
Epidemiologie
Die Virusepidemiologie ist der Zweig der medizinischen Wissenschaft, der sich mit der Übertragung und Bekämpfung von Virusinfektionen beim Menschen befasst. Die Übertragung von Viren kann vertikal, d. h. von der Mutter auf das Kind, oder horizontal, d. h. von Mensch zu Mensch, erfolgen. Beispiele für eine vertikale Übertragung sind das Hepatitis-B-Virus und HIV, bei denen das Baby bereits mit dem Virus infiziert geboren wird. Ein weiteres, selteneres Beispiel ist das Varizella-Zoster-Virus, das zwar bei Kindern und Erwachsenen relativ milde Infektionen verursacht, für den Fötus und das Neugeborene jedoch tödlich sein kann. ⓘ
Die horizontale Übertragung ist der häufigste Mechanismus zur Verbreitung von Viren in der Bevölkerung. Zu einer horizontalen Übertragung kann es kommen, wenn bei sexuellen Aktivitäten Körperflüssigkeiten ausgetauscht werden, durch den Austausch von Speichel oder durch die Aufnahme von kontaminierten Lebensmitteln oder Wasser. Sie kann auch erfolgen, wenn virushaltige Aerosole eingeatmet werden oder durch Insektenvektoren, z. B. wenn infizierte Stechmücken in die Haut eines Wirtes eindringen. Die meisten Virustypen beschränken sich auf nur einen oder zwei dieser Mechanismen und werden als "respiratorische Viren" oder "enterische Viren" usw. bezeichnet. Die Geschwindigkeit der Übertragung von Virusinfektionen hängt von Faktoren wie der Bevölkerungsdichte, der Zahl der empfänglichen Personen (d. h. derjenigen, die nicht immun sind), der Qualität der Gesundheitsversorgung und dem Wetter ab. ⓘ
Die Epidemiologie wird eingesetzt, um die Infektionskette in der Bevölkerung bei Ausbrüchen von Viruserkrankungen zu unterbrechen. Es werden Bekämpfungsmaßnahmen ergriffen, die auf dem Wissen über die Übertragung des Virus beruhen. Es ist wichtig, die Quelle(n) des Ausbruchs zu finden und das Virus zu identifizieren. Sobald das Virus identifiziert ist, kann die Übertragungskette manchmal durch Impfstoffe unterbrochen werden. Wenn keine Impfstoffe verfügbar sind, können Hygiene und Desinfektion wirksam sein. Oft werden infizierte Personen vom Rest der Gemeinschaft isoliert und diejenigen, die dem Virus ausgesetzt waren, unter Quarantäne gestellt. Um den Ausbruch der Maul- und Klauenseuche bei Rindern in Großbritannien im Jahr 2001 zu bekämpfen, wurden Tausende von Rindern geschlachtet. Die meisten Virusinfektionen bei Menschen und anderen Tieren haben eine Inkubationszeit, in der die Infektion keine Anzeichen oder Symptome verursacht. Die Inkubationszeiten für Viruskrankheiten reichen von einigen Tagen bis zu Wochen, sind aber für die meisten Infektionen bekannt. Etwas überlappend, aber hauptsächlich im Anschluss an die Inkubationszeit, gibt es eine Zeit der Ansteckungsfähigkeit - eine Zeit, in der eine infizierte Person oder ein infiziertes Tier ansteckend ist und eine andere Person oder ein anderes Tier infizieren kann. Auch dies ist für viele Virusinfektionen bekannt, und die Kenntnis der Länge beider Zeiträume ist wichtig für die Kontrolle von Ausbrüchen. Wenn Ausbrüche einen ungewöhnlich hohen Anteil an Fällen in einer Bevölkerung, Gemeinde oder Region verursachen, werden sie als Epidemien bezeichnet. Wenn sich Ausbrüche weltweit ausbreiten, werden sie als Pandemien bezeichnet. ⓘ
Epidemien und Pandemien
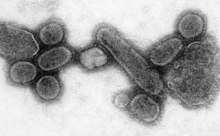
Eine Pandemie ist eine weltweite Epidemie. Die Grippepandemie von 1918, die bis 1919 andauerte, war eine Grippepandemie der Kategorie 5, die durch ein ungewöhnlich schweres und tödliches Influenza-A-Virus verursacht wurde. Bei den Opfern handelte es sich häufig um gesunde junge Erwachsene, im Gegensatz zu den meisten Grippeepidemien, die überwiegend Jugendliche, ältere oder anderweitig geschwächte Patienten betreffen. Älteren Schätzungen zufolge starben 40-50 Millionen Menschen an der Grippe, während neuere Forschungen darauf hindeuten, dass sie bis zu 100 Millionen Menschen oder 5 % der Weltbevölkerung im Jahr 1918 getötet haben könnte. ⓘ
Virale Pandemien sind zwar selten, aber HIV - das sich aus Viren entwickelt hat, die bei Affen und Schimpansen vorkommen - ist mindestens seit den 1980er Jahren eine Pandemie. Im 20. Jahrhundert gab es vier Pandemien, die durch Influenzaviren verursacht wurden, und die 1918, 1957 und 1968 aufgetretenen Pandemien waren schwerwiegend. Die meisten Forscher gehen davon aus, dass HIV im 20. Jahrhundert in Afrika südlich der Sahara entstanden ist; heute ist es eine Pandemie, und weltweit leben schätzungsweise 37,9 Millionen Menschen mit dieser Krankheit. Im Jahr 2018 starben etwa 770.000 Menschen an AIDS. Das Gemeinsame Programm der Vereinten Nationen für HIV/AIDS (UNAIDS) und die Weltgesundheitsorganisation (WHO) schätzen, dass AIDS seit seiner erstmaligen Anerkennung am 5. Juni 1981 mehr als 25 Millionen Todesopfer gefordert hat und damit eine der zerstörerischsten Epidemien der Geschichte ist. Im Jahr 2007 gab es 2,7 Millionen neue HIV-Infektionen und 2 Millionen HIV-bedingte Todesfälle.
Mehrere hochgradig tödliche virale Krankheitserreger gehören zur Familie der Filoviridae. Filoviren sind fadenförmige Viren, die virales hämorrhagisches Fieber verursachen, darunter Ebolaviren und Marburgviren. Das Marburg-Virus, das erstmals 1967 entdeckt wurde, erregte im April 2005 wegen eines Ausbruchs in Angola große Aufmerksamkeit in der Presse. Auch das Ebola-Virus hat seit seiner Entdeckung 1976 immer wieder Ausbrüche mit hoher Sterblichkeitsrate verursacht. Der schlimmste und jüngste Ausbruch war die Epidemie 2013-2016 in Westafrika. ⓘ
Mit Ausnahme der Pocken werden die meisten Pandemien durch neu entwickelte Viren verursacht. Bei diesen "auftauchenden" Viren handelt es sich in der Regel um Mutanten von weniger gefährlichen Viren, die zuvor beim Menschen oder bei anderen Tieren zirkulierten. ⓘ
Das Schwere Akute Respiratorische Syndrom (SARS) und das Middle East Respiratory Syndrom (MERS) werden durch neue Typen von Coronaviren verursacht. Andere Coronaviren sind dafür bekannt, dass sie beim Menschen leichte Infektionen hervorrufen, so dass die Virulenz und die rasche Ausbreitung der SARS-Infektionen - die bis Juli 2003 etwa 8.000 Fälle und 800 Todesfälle verursachten - unerwartet waren und die meisten Länder nicht darauf vorbereitet waren. ⓘ
Ein verwandtes Coronavirus, das Schwere Akute Respiratorische Syndrom Coronavirus 2 (SARS-Cov-2), von dem man annimmt, dass es von Fledermäusen stammt, tauchte im November 2019 in Wuhan, China, auf und verbreitete sich rasch in der ganzen Welt. Infektionen mit dem Virus verursachten die COVID-19-Pandemie, die im Jahr 2020 begann. Als Reaktion auf die Pandemie wurden in Friedenszeiten noch nie dagewesene Einschränkungen für den internationalen Reiseverkehr verhängt und in mehreren Großstädten weltweit Ausgangssperren verhängt. ⓘ
Krebs
Viren sind nachweislich eine Ursache für Krebs beim Menschen und bei anderen Tierarten. Virusbedingte Krebserkrankungen treten nur bei einer Minderheit der infizierten Personen (oder Tiere) auf. Krebsviren gehören zu einer Reihe von Virusfamilien, darunter sowohl RNA- als auch DNA-Viren, so dass es keinen einzigen Typ von "Onkoviren" gibt (ein veralteter Begriff, der ursprünglich für akut transformierende Retroviren verwendet wurde). Die Entwicklung von Krebs wird durch eine Vielzahl von Faktoren bestimmt, wie z. B. die Immunität des Wirtes und Mutationen im Wirt. Zu den Viren, die als Verursacher von Krebs beim Menschen gelten, gehören einige Genotypen des humanen Papillomavirus, das Hepatitis-B-Virus, das Hepatitis-C-Virus, das Epstein-Barr-Virus, das Kaposi-Sarkom-assoziierte Herpesvirus und das humane T-Lymphotropie-Virus. Das jüngst entdeckte menschliche Krebsvirus ist ein Polyomavirus (Merkelzell-Polyomavirus), das die meisten Fälle einer seltenen Form von Hautkrebs namens Merkelzellkarzinom verursacht. Hepatitisviren können sich zu einer chronischen Virusinfektion entwickeln, die zu Leberkrebs führt. Eine Infektion mit dem humanen T-lymphotropen Virus kann zu tropischer spastischer Paraparese und adulter T-Zell-Leukämie führen. Humane Papillomviren sind eine etablierte Ursache für Krebserkrankungen des Gebärmutterhalses, der Haut, des Anus und des Penis. Innerhalb der Herpesviridae verursacht das Kaposi-Sarkom-assoziierte Herpesvirus das Kaposi-Sarkom und das Körperhöhlen-Lymphom, und das Epstein-Barr-Virus verursacht das Burkitt-Lymphom, das Hodgkin-Lymphom, die lymphoproliferative B-Störung und das Nasopharynx-Karzinom. Merkelzell-Polyomavirus, eng verwandt mit SV40 und Maus-Polyomaviren, die seit über 50 Jahren als Tiermodelle für Krebsviren verwendet werden. ⓘ
Abwehrmechanismen des Wirts
Die erste Verteidigungslinie des Körpers gegen Viren ist das angeborene Immunsystem. Es besteht aus Zellen und anderen Mechanismen, die den Wirt auf unspezifische Weise vor einer Infektion schützen. Das bedeutet, dass die Zellen des angeborenen Systems Krankheitserreger auf generische Weise erkennen und auf sie reagieren, aber im Gegensatz zum adaptiven Immunsystem verleiht es dem Wirt keine dauerhafte oder schützende Immunität. ⓘ
Die RNA-Interferenz ist eine wichtige angeborene Abwehr gegen Viren. Viele Viren haben eine Replikationsstrategie, die doppelsträngige RNA (dsRNA) beinhaltet. Wenn ein solches Virus eine Zelle infiziert, setzt es sein RNA-Molekül oder seine RNA-Moleküle frei, die sofort an einen Proteinkomplex, den so genannten Dicer, binden, der die RNA in kleinere Stücke schneidet. Ein biochemischer Weg - der RISC-Komplex - wird aktiviert, der das Überleben der Zelle durch den Abbau der viralen mRNA sicherstellt. Rotaviren haben sich so entwickelt, dass sie diesen Abwehrmechanismus umgehen, indem sie sich in der Zelle nicht vollständig enthüllen und neu produzierte mRNA durch Poren im inneren Kapsid des Partikels freisetzen. Ihre genomische dsRNA bleibt im Kern des Virions geschützt. ⓘ
Wenn das adaptive Immunsystem eines Wirbeltiers auf ein Virus stößt, produziert es spezifische Antikörper, die an das Virus binden und es oft nicht mehr infektiös machen. Dies wird als humorale Immunität bezeichnet. Zwei Arten von Antikörpern sind wichtig. Der erste, IgM genannt, ist hochwirksam bei der Neutralisierung von Viren, wird aber von den Zellen des Immunsystems nur für einige Wochen produziert. Der zweite, IgG genannt, wird unbegrenzt produziert. Das Vorhandensein von IgM im Blut des Wirtes wird zum Nachweis einer akuten Infektion verwendet, während IgG auf eine Infektion in der Vergangenheit hinweist. IgG-Antikörper werden bei Immunitätstests gemessen. ⓘ
Antikörper können auch dann noch ein wirksamer Abwehrmechanismus sein, wenn es den Viren gelungen ist, in die Wirtszelle einzudringen. Ein zelleigenes Protein namens TRIM21 kann sich an die Antikörper auf der Oberfläche des Viruspartikels anlagern. Dadurch wird die anschließende Zerstörung des Virus durch die Enzyme des Proteosomensystems der Zelle eingeleitet. ⓘ
Eine zweite Abwehrmaßnahme von Wirbeltieren gegen Viren ist die so genannte zellvermittelte Immunität, an der Immunzellen, so genannte T-Zellen, beteiligt sind. Wenn eine T-Zelle dort ein verdächtiges Virusfragment erkennt, wird die Wirtszelle von "Killer-T-Zellen" zerstört, und die virusspezifischen T-Zellen vermehren sich. Zellen wie die Makrophagen sind Spezialisten für diese Antigenpräsentation. Die Produktion von Interferon ist ein wichtiger Abwehrmechanismus des Wirts. Dabei handelt es sich um ein Hormon, das vom Körper produziert wird, wenn Viren vorhanden sind. Seine Rolle in der Immunität ist komplex; es stoppt schließlich die Vermehrung der Viren, indem es die infizierte Zelle und ihre nahen Nachbarn abtötet. ⓘ
Nicht alle Virusinfektionen lösen auf diese Weise eine schützende Immunreaktion aus. HIV entzieht sich dem Immunsystem, indem es die Aminosäuresequenz der Proteine auf der Oberfläche des Virions ständig verändert. Dies wird als "Escape-Mutation" bezeichnet, da die viralen Epitope der Erkennung durch die Immunantwort des Wirts entgehen. Diese persistenten Viren entziehen sich der Immunkontrolle durch Sequestrierung, Blockierung der Antigenpräsentation, Zytokinresistenz, Umgehung der Aktivitäten der natürlichen Killerzellen, Flucht vor der Apoptose und Antigenshift. Andere Viren, so genannte "neurotrope Viren", werden durch neurale Ausbreitung verbreitet, wo das Immunsystem sie aufgrund der Immunprivilegierung möglicherweise nicht erreichen kann. ⓘ
Prävention und Behandlung
Da Viren lebenswichtige Stoffwechselwege in den Wirtszellen nutzen, um sich zu vermehren, lassen sie sich nur schwer beseitigen, ohne dass Medikamente eingesetzt werden, die toxische Wirkungen auf die Wirtszellen im Allgemeinen haben. Die wirksamsten medizinischen Ansätze zur Behandlung von Viruserkrankungen sind Impfungen, die eine Immunität gegen die Infektion vermitteln, und antivirale Medikamente, die selektiv in die Virusvermehrung eingreifen. ⓘ
Impfstoffe
Impfungen sind eine kostengünstige und wirksame Methode zur Verhinderung von Virusinfektionen. Impfstoffe wurden schon lange vor der Entdeckung der eigentlichen Viren zur Vorbeugung von Virusinfektionen eingesetzt. Ihr Einsatz hat zu einem dramatischen Rückgang der Morbidität (Krankheit) und Mortalität (Tod) im Zusammenhang mit Virusinfektionen wie Polio, Masern, Mumps und Röteln geführt. Die Pockeninfektionen wurden ausgerottet. Es gibt Impfstoffe zur Vorbeugung von über dreizehn Virusinfektionen beim Menschen, und weitere werden zur Vorbeugung von Virusinfektionen bei Tieren eingesetzt. Impfstoffe können aus abgeschwächten oder abgetöteten Lebendviren, viralen Proteinen (Antigenen) oder RNA bestehen. Lebendimpfstoffe enthalten abgeschwächte Formen des Virus, die die Krankheit nicht auslösen, aber dennoch eine Immunität verleihen. Solche Viren werden als abgeschwächt bezeichnet. Lebendimpfstoffe können gefährlich sein, wenn sie Menschen mit einer schwachen Immunität verabreicht werden (die als immungeschwächt bezeichnet werden), denn bei diesen Menschen kann das abgeschwächte Virus die ursprüngliche Krankheit auslösen. Biotechnologie und gentechnische Verfahren werden zur Herstellung von Subunit-Impfstoffen eingesetzt. Für diese Impfstoffe werden nur die Kapsidproteine des Virus verwendet. Der Hepatitis-B-Impfstoff ist ein Beispiel für diese Art von Impfstoff. Subunit-Impfstoffe sind für immungeschwächte Patienten sicher, da sie die Krankheit nicht auslösen können. Der Impfstoff gegen das Gelbfiebervirus, ein abgeschwächter Lebendstamm namens 17D, ist wahrscheinlich der sicherste und wirksamste Impfstoff, der je hergestellt wurde. ⓘ
Antivirale Medikamente

Antivirale Medikamente sind häufig Nukleosidanaloga (falsche DNA-Bausteine), die Viren während der Replikation irrtümlich in ihr Genom einbauen. Der Lebenszyklus des Virus wird dann gestoppt, weil die neu synthetisierte DNA inaktiv ist. Das liegt daran, dass diesen Analoga die Hydroxylgruppen fehlen, die zusammen mit den Phosphoratomen das starke "Rückgrat" des DNA-Moleküls bilden. Dies wird als DNA-Kettenabbruch bezeichnet. Beispiele für Nukleosidanaloga sind Aciclovir bei Infektionen mit dem Herpes-simplex-Virus und Lamivudin bei Infektionen mit dem HIV- und Hepatitis-B-Virus. Aciclovir ist eines der ältesten und am häufigsten verschriebenen antiviralen Arzneimittel. Andere antivirale Medikamente, die eingesetzt werden, zielen auf verschiedene Stadien des viralen Lebenszyklus ab. HIV ist auf ein proteolytisches Enzym, die HIV-1-Protease, angewiesen, um vollständig infektiös zu werden. Es gibt eine große Klasse von Arzneimitteln, die Protease-Inhibitoren, die dieses Enzym inaktivieren. Es gibt etwa dreizehn Klassen von antiviralen Medikamenten, die jeweils auf verschiedene Viren oder Stadien der viralen Replikation abzielen. ⓘ
Hepatitis C wird durch ein RNA-Virus verursacht. Bei 80 % der Infizierten verläuft die Krankheit chronisch, und ohne Behandlung bleiben sie für den Rest ihres Lebens infiziert. Es gibt wirksame Behandlungen, bei denen direkt wirkende antivirale Medikamente zum Einsatz kommen. Auch für die Behandlung chronischer Träger des Hepatitis-B-Virus wurden ähnliche Strategien entwickelt, die Lamivudin und andere antivirale Medikamente umfassen. ⓘ
Infektion bei anderen Arten
Viren infizieren alles zelluläre Leben, und obwohl Viren universell vorkommen, hat jede zelluläre Spezies ihr eigenes spezifisches Spektrum, das oft nur diese Spezies infiziert. Einige Viren, so genannte Satellitenviren, können sich nur in Zellen vermehren, die bereits von einem anderen Virus infiziert worden sind. ⓘ
Tierviren
Viren sind wichtige Krankheitserreger in der Tierhaltung. Krankheiten wie die Maul- und Klauenseuche und die Blauzungenkrankheit werden durch Viren verursacht. Haustiere wie Katzen, Hunde und Pferde sind, wenn sie nicht geimpft sind, anfällig für schwere Virusinfektionen. Das Hundeparvovirus wird durch ein kleines DNA-Virus verursacht, und Infektionen sind bei Welpen oft tödlich. Wie alle wirbellosen Tiere ist auch die Honigbiene anfällig für viele Virusinfektionen. Die meisten Viren koexistieren harmlos in ihrem Wirt und verursachen keine Krankheitsanzeichen oder -symptome. ⓘ
Pflanzenviren

Es gibt viele Arten von Pflanzenviren, aber oft verursachen sie nur Ertragseinbußen, und es ist wirtschaftlich nicht sinnvoll, sie zu bekämpfen. Pflanzenviren werden oft durch Organismen, so genannte Vektoren, von Pflanze zu Pflanze übertragen. Dabei handelt es sich in der Regel um Insekten, aber auch einige Pilze, Fadenwürmer, Einzeller und parasitische Pflanzen sind Vektoren. Wenn die Bekämpfung von Pflanzenvirusinfektionen als wirtschaftlich angesehen wird, z. B. bei mehrjährigen Früchten, konzentrieren sich die Bemühungen auf die Abtötung der Vektoren und die Beseitigung alternativer Wirte wie Unkraut. Pflanzenviren können Menschen und andere Tiere nicht infizieren, da sie sich nur in lebenden Pflanzenzellen vermehren können. ⓘ
Die ursprünglich aus Peru stammende Kartoffel hat sich weltweit zu einem Grundnahrungsmittel entwickelt. Das Kartoffelvirus Y verursacht Krankheiten bei Kartoffeln und verwandten Arten wie Tomaten und Paprika. In den 1980er Jahren erlangte dieses Virus wirtschaftliche Bedeutung, als es sich in Saatkartoffelkulturen als schwer bekämpfbar erwies. Das von Blattläusen übertragene Virus kann die Ernteerträge um bis zu 80 Prozent verringern und so erhebliche Einbußen bei den Kartoffelerträgen verursachen. ⓘ
Pflanzen verfügen über ausgeklügelte und wirksame Abwehrmechanismen gegen Viren. Einer der wirksamsten ist das Vorhandensein so genannter Resistenzgene (R-Gene). Jedes R-Gen verleiht Resistenz gegen ein bestimmtes Virus, indem es örtlich begrenzte Bereiche des Zelltods um die infizierte Zelle herum auslöst, die oft mit bloßem Auge als große Flecken zu erkennen sind. Dadurch wird die Ausbreitung der Infektion verhindert. Auch bei Pflanzen ist die RNA-Interferenz ein wirksamer Schutz. Wenn sie infiziert sind, produzieren Pflanzen häufig natürliche Desinfektionsmittel, die Viren abtöten, wie z. B. Salicylsäure, Stickstoffoxid und reaktive Sauerstoffmoleküle. ⓘ
Pflanzenviruspartikel oder virusähnliche Partikel (VLPs) finden sowohl in der Biotechnologie als auch in der Nanotechnologie Anwendung. Die Kapside der meisten Pflanzenviren sind einfache und robuste Strukturen und können in großen Mengen entweder durch die Infektion von Pflanzen oder durch Expression in einer Vielzahl heterologer Systeme hergestellt werden. Pflanzenviruspartikel können genetisch und chemisch so modifiziert werden, dass sie Fremdmaterial einkapseln und in supramolekulare Strukturen zur Verwendung in der Biotechnologie eingebaut werden können. ⓘ
Bakterielle Viren

Bakteriophagen sind eine weit verbreitete und vielfältige Gruppe von Viren und die am häufigsten vorkommende biologische Einheit in der aquatischen Umwelt - in den Ozeanen gibt es bis zu zehnmal mehr dieser Viren als Bakterien, wobei eine Konzentration von 250.000.000 Bakteriophagen pro Milliliter Meerwasser erreicht wird. Diese Viren infizieren bestimmte Bakterien, indem sie an Oberflächenrezeptormoleküle binden und dann in die Zelle eindringen. Innerhalb kurzer Zeit, in manchen Fällen nur wenige Minuten, beginnt die bakterielle Polymerase, die virale mRNA in Proteine zu übersetzen. Diese Proteine werden dann entweder zu neuen Virionen in der Zelle, zu Helferproteinen, die den Zusammenbau neuer Virionen unterstützen, oder zu Proteinen, die an der Zelllyse beteiligt sind. Virale Enzyme helfen bei der Zerstörung der Zellmembran, und im Falle des T4-Phagen konnten in etwas mehr als zwanzig Minuten nach der Injektion über dreihundert Phagen freigesetzt werden. ⓘ
Die wichtigste Art und Weise, wie sich Bakterien gegen Bakteriophagen verteidigen, ist die Produktion von Enzymen, die fremde DNA zerstören. Diese Enzyme, Restriktionsendonukleasen genannt, zerschneiden die virale DNA, die Bakteriophagen in die Bakterienzellen injizieren. Bakterien verfügen auch über ein System, das CRISPR-Sequenzen verwendet, um Fragmente der Genome von Viren zu speichern, mit denen die Bakterien in der Vergangenheit in Kontakt gekommen sind. Dies ermöglicht ihnen, die Replikation des Virus durch eine Art RNA-Interferenz zu blockieren. Dieses genetische System verleiht den Bakterien eine erworbene Immunität gegen Infektionen. ⓘ
Archaeen-Viren
Einige Viren vermehren sich in Archaeen: Es handelt sich um DNA-Viren mit ungewöhnlichen und manchmal einzigartigen Formen. Am eingehendsten wurden diese Viren in den thermophilen Archaeen untersucht, insbesondere in den Ordnungen Sulfolobales und Thermoproteales. Die Abwehr dieser Viren beruht auf der RNA-Interferenz von sich wiederholenden DNA-Sequenzen in Archaeen-Genomen, die mit den Genen der Viren verwandt sind. Die meisten Archaeen verfügen über CRISPR-Cas-Systeme, die eine adaptive Verteidigung gegen Viren darstellen. Diese ermöglichen es den Archaeen, Abschnitte der viralen DNA zu bewahren, die dann dazu verwendet werden, nachfolgende Infektionen durch das Virus mithilfe eines der RNA-Interferenz ähnlichen Prozesses gezielt zu eliminieren. ⓘ
Rolle in aquatischen Ökosystemen
Viren sind die am häufigsten vorkommende biologische Einheit in der aquatischen Umwelt. In einem Teelöffel Meerwasser befinden sich etwa zehn Millionen von ihnen. Bei den meisten dieser Viren handelt es sich um Bakteriophagen, die heterotrophe Bakterien infizieren, und um Cyanophagen, die Cyanobakterien befallen. Sie sind für die Regulierung von Salz- und Süßwasserökosystemen von wesentlicher Bedeutung. Bakteriophagen sind für Pflanzen und Tiere unschädlich und spielen bei der Regulierung von Meeres- und Süßwasserökosystemen eine wichtige Rolle, da sie das Phytoplankton, die Basis der Nahrungskette in der aquatischen Umwelt, abtöten. Sie infizieren und zerstören Bakterien in mikrobiellen Gemeinschaften im Wasser und sind einer der wichtigsten Mechanismen für das Recycling von Kohlenstoff und den Nährstoffkreislauf in der Meeresumwelt. Die von den toten Bakterienzellen freigesetzten organischen Moleküle stimulieren das Wachstum neuer Bakterien und Algen in einem Prozess, der als viraler Shunt bekannt ist. Insbesondere hat sich gezeigt, dass die Lyse von Bakterien durch Viren den Stickstoffkreislauf verbessert und das Wachstum des Phytoplanktons anregt. Virale Aktivität kann auch die biologische Pumpe beeinflussen, den Prozess, durch den Kohlenstoff in der Tiefsee gebunden wird. ⓘ
Mikroorganismen machen mehr als 90 % der Biomasse im Meer aus. Man schätzt, dass Viren jeden Tag etwa 20 % dieser Biomasse abtöten und dass es in den Ozeanen 10- bis 15-mal so viele Viren wie Bakterien und Archaeen gibt. Viren sind auch die Hauptverantwortlichen für die Zerstörung des Phytoplanktons, einschließlich schädlicher Algenblüten, Die Zahl der Viren in den Ozeanen nimmt mit zunehmender Entfernung von der Küste und tiefer im Wasser ab, wo es weniger Wirtsorganismen gibt. ⓘ
Im Januar 2018 berichteten Wissenschaftler, dass täglich 800 Millionen Viren, hauptsächlich marinen Ursprungs, aus der Erdatmosphäre auf jeden Quadratmeter der Erdoberfläche gelangen, als Ergebnis eines globalen atmosphärischen Virenstroms, der oberhalb des Wettersystems, aber unterhalb der Flughöhe üblicher Flugreisen zirkuliert und die Viren rund um den Planeten verteilt. ⓘ
Wie alle Organismen sind auch Meeressäugetiere anfällig für Virusinfektionen. In den Jahren 1988 und 2002 wurden in Europa Tausende von Seehunden durch das Phokine Staupe-Virus getötet. Viele andere Viren, darunter Caliciviren, Herpesviren, Adenoviren und Parvoviren, zirkulieren in Meeressäugerpopulationen. ⓘ
Rolle in der Evolution
Viren sind ein wichtiges natürliches Mittel zur Übertragung von Genen zwischen verschiedenen Arten, was die genetische Vielfalt erhöht und die Evolution vorantreibt. Man geht davon aus, dass Viren in der frühen Evolution eine zentrale Rolle spielten, bevor sich der letzte gemeinsame Vorfahre in Bakterien, Archaeen und Eukaryonten diversifizierte. Viren sind nach wie vor eines der größten Reservoirs an unerforschter genetischer Vielfalt auf der Erde. ⓘ
Anwendungen
Biowissenschaften und Medizin

Viren sind wichtig für die Erforschung der Molekular- und Zellbiologie, da sie einfache Systeme darstellen, mit denen die Funktionen von Zellen manipuliert und untersucht werden können. Die Untersuchung und Verwendung von Viren hat wertvolle Informationen über Aspekte der Zellbiologie geliefert. So haben sich Viren beispielsweise bei der Untersuchung der Genetik als nützlich erwiesen und zu unserem Verständnis der grundlegenden Mechanismen der Molekulargenetik beigetragen, wie DNA-Replikation, Transkription, RNA-Verarbeitung, Translation, Proteintransport und Immunologie. ⓘ
Genetiker verwenden häufig Viren als Vektoren, um Gene in die zu untersuchenden Zellen einzuschleusen. Dies ist nützlich, um die Zelle dazu zu bringen, eine fremde Substanz zu produzieren, oder um die Auswirkungen der Einführung eines neuen Gens in das Genom zu untersuchen. In ähnlicher Weise werden in der Virotherapie Viren als Vektoren zur Behandlung verschiedener Krankheiten eingesetzt, da sie gezielt Zellen und DNA angreifen können. Sie ist vielversprechend für die Behandlung von Krebs und für die Gentherapie. Osteuropäische Wissenschaftler setzen seit einiger Zeit die Phagentherapie als Alternative zu Antibiotika ein, und das Interesse an diesem Ansatz nimmt angesichts der hohen Antibiotikaresistenz einiger pathogener Bakterien zu. Die Expression heterologer Proteine durch Viren ist die Grundlage mehrerer Herstellungsverfahren, die derzeit für die Produktion verschiedener Proteine wie Impfstoffantigene und Antikörper verwendet werden. In jüngster Zeit wurden industrielle Verfahren unter Verwendung viraler Vektoren entwickelt, und mehrere pharmazeutische Proteine befinden sich derzeit in präklinischen und klinischen Versuchen. ⓘ
Virotherapie
Bei der Virotherapie werden genetisch veränderte Viren zur Behandlung von Krankheiten eingesetzt. Die Viren wurden von Wissenschaftlern so verändert, dass sie sich in Krebszellen vermehren und diese zerstören, aber keine gesunden Zellen infizieren. Talimogene laherparepvec (T-VEC) beispielsweise ist ein modifiziertes Herpes-simplex-Virus, bei dem ein Gen, das für die Vermehrung des Virus in gesunden Zellen erforderlich ist, entfernt und durch ein menschliches Gen (GM-CSF) ersetzt wurde, das die Immunität stimuliert. Wenn dieses Virus Krebszellen infiziert, zerstört es diese und lockt dabei durch das Vorhandensein des GM-CSF-Gens dendritische Zellen aus dem umliegenden Körpergewebe an. Die dendritischen Zellen verarbeiten die abgetöteten Krebszellen und geben Bestandteile davon an andere Zellen des Immunsystems weiter. Nach erfolgreichen klinischen Studien wurde das Virus Ende 2015 für die Behandlung von Melanomen zugelassen. Viren, die umprogrammiert wurden, um Krebszellen abzutöten, werden als onkolytische Viren bezeichnet. ⓘ
Materialwissenschaft und Nanotechnologie
Die aktuellen Trends in der Nanotechnologie versprechen eine vielseitigere Nutzung von Viren. Aus der Sicht eines Materialwissenschaftlers können Viren als organische Nanopartikel betrachtet werden. Auf ihrer Oberfläche befinden sich spezifische Werkzeuge, die es ihnen ermöglichen, die Barrieren ihrer Wirtszellen zu überwinden. Die Größe und Form von Viren sowie die Anzahl und Art der funktionellen Gruppen auf ihrer Oberfläche sind genau definiert. Daher werden Viren in der Materialwissenschaft häufig als Gerüst für kovalent verbundene Oberflächenmodifikationen verwendet. Eine besondere Eigenschaft von Viren ist, dass sie durch gezielte Evolution maßgeschneidert werden können. Die von den Biowissenschaften entwickelten leistungsstarken Techniken werden zur Grundlage für die Entwicklung von Nanomaterialien und eröffnen ein breites Spektrum von Anwendungen, die weit über Biologie und Medizin hinausgehen. ⓘ
Aufgrund ihrer Größe, Form und gut definierten chemischen Strukturen wurden Viren als Vorlagen für die Organisation von Materialien auf der Nanoskala verwendet. Jüngste Beispiele sind Arbeiten des Naval Research Laboratory in Washington, D.C., bei denen Partikel des Cowpea mosaic virus (CPMV) zur Verstärkung von Signalen in Sensoren auf Basis von DNA-Mikroarrays eingesetzt werden. Bei dieser Anwendung trennen die Viruspartikel die für die Signalgebung verwendeten Fluoreszenzfarbstoffe, um die Bildung von nicht fluoreszierenden Dimeren zu verhindern, die als Quencher wirken. Ein weiteres Beispiel ist die Verwendung von CPMV als nanoskaliges Breadboard für molekulare Elektronik. ⓘ
Synthetische Viren
Viele Viren können de novo ("von Grund auf") synthetisiert werden. Das erste synthetische Virus wurde im Jahr 2002 entwickelt. Obwohl es sich hierbei um ein Missverständnis handelt, wird nicht das eigentliche Virus synthetisiert, sondern sein DNA-Genom (im Falle eines DNA-Virus) oder eine cDNA-Kopie seines Genoms (im Falle von RNA-Viren). Bei vielen Virusfamilien ist die nackte synthetische DNA oder RNA (nach enzymatischer Rückumwandlung der synthetischen cDNA) infektiös, wenn sie in eine Zelle eingebracht wird. Das heißt, sie enthalten alle notwendigen Informationen, um neue Viren zu produzieren. Diese Technologie wird nun zur Erforschung neuer Impfstoffstrategien eingesetzt. Die Fähigkeit, Viren zu synthetisieren, hat weitreichende Folgen, da Viren nicht mehr als ausgestorben gelten können, solange die Information ihrer Genomsequenz bekannt ist und permissive Zellen zur Verfügung stehen. Ab Juni 2021 sind die vollständigen Genomsequenzen von 11 464 verschiedenen Viren, einschließlich der Pocken, in einer von den National Institutes of Health unterhaltenen Online-Datenbank öffentlich zugänglich. ⓘ
Waffen
Die Fähigkeit von Viren, verheerende Epidemien in menschlichen Gesellschaften auszulösen, hat zu der Sorge geführt, dass Viren als Waffe für die biologische Kriegsführung eingesetzt werden könnten. Die erfolgreiche Nachbildung des berüchtigten Influenzavirus von 1918 in einem Labor gab Anlass zu weiterer Besorgnis. Das Pockenvirus hat im Laufe der Geschichte zahlreiche Gesellschaften verwüstet, bevor es ausgerottet wurde. Weltweit gibt es nur zwei Zentren, die von der WHO ermächtigt sind, Pockenviren zu lagern: das staatliche Forschungszentrum für Virologie und Biotechnologie VECTOR in Russland und die Centers for Disease Control and Prevention in den Vereinigten Staaten. Da der Pockenimpfstoff mitunter schwere Nebenwirkungen hatte, wird er in keinem Land mehr routinemäßig eingesetzt, sondern kann als Waffe verwendet werden. Ein großer Teil der modernen menschlichen Bevölkerung hat daher kaum eine Resistenz gegen Pocken entwickelt und wäre dem Virus gegenüber anfällig. ⓘ
Erforschungsgeschichte
Noch zur Mitte des 19. Jahrhunderts verwendete man die Bezeichnung Virus lediglich synonym für „Gift“ bzw. „Miasma“. Erst seit dem späten 19. Jahrhundert sind Viren als eigene biologische Einheit bekannt. Die Beschreibungen von Viruskrankheiten sind aber sehr viel älter, ebenso die ersten Behandlungsmethoden. Aus Mesopotamien ist ein Gesetzestext aus der Zeit um 1780 v. Chr. überliefert, der von der Bestrafung eines Mannes handelt, dessen wahrscheinlich von Tollwut befallener Hund einen Menschen beißt und dadurch tötet (Codex Eschnunna §§ 56 und 57). Aus ägyptischen Hieroglyphen sind Darstellungen bekannt, die vermutlich die Folgen einer Polio-Infektion zeigen. ⓘ
Die Bezeichnung „Virus“ wurde zum ersten Mal von Cornelius Aulus Celsus im ersten Jahrhundert v. Chr. verwendet. Er bezeichnete den Speichel, durch den Tollwut übertragen wurde, als „giftig“. Im Jahr 1882 führte Adolf Mayer bei Experimenten mit der Tabakmosaikkrankheit erstmals unwissentlich eine virale Erregerübertragung (Transmission) durch, indem er den Pflanzensaft infizierter Pflanzen auf gesunde Pflanzen übertrug und bei diesen so ebenfalls die Krankheit auslöste. ⓘ
Diese Übertragung war bereits im 18. Jahrhundert mit dem Wort Virus assoziiert. So beschreibt die Londoner Times in einem Nachruf auf einen Arzt dessen Virusinfektion: Beim Zunähen einer sezierten Leiche hatte er sich in die Hand gestochen, „which introduced some of the virus matter, or, in other words, inoculated him with putridity“ (wobei ein wenig Virussubstanz übertragen wurde, oder anders gesagt, ihm wurde Fäulnis eingeimpft). ⓘ
Dimitri Iwanowski wies unabhängig von Mayer im Jahr 1892 in einem Experiment nach, dass die Mosaikkrankheit bei Tabakpflanzen durch einen Stoff ausgelöst werden kann, der durch Filtration mittels bakteriendichter Filter (Chamberland-Filter) nicht entfernt werden konnte und dessen Partikel deshalb deutlich kleiner als Bakterien sein mussten. Iwanowski vermutete ein Gift als Auslöser der Krankheit. Dem widersprach Martinus Willem Beijerinck, nachdem er beobachtet hatte, dass besonders die noch im Wachstum befindlichen Blätter von der Krankheit befallen wurden: „Während ein Gift ebenso in die bereits bestehenden Zellen eindringen würde, brauchen die Viren offensichtlich die Zellteilung, um sich zu entfalten.“ ⓘ
In den 1910er Jahren vermuteten Wissenschaftler noch, Viren könnten auf sterilen Nährmedien gezüchtet werden und bräuchten nicht notwendigerweise lebende Zellen, um sich zu vermehren. Die erfolglosen Arbeiten von Frederick Twort auf diesem Gebiet führten jedoch zur Entdeckung der Bakterienviren, der sogenannten Bakteriophagen. ⓘ
Der erste Nachweis eines tierischen Virus gelang 1898 Friedrich Loeffler und Paul Frosch, die das Maul-und-Klauenseuche-Virus entdeckten (siehe auch virologische Diagnostik). Die Größe vieler Viren wurde in den 1930er Jahren durch William Joseph Elford mit Methoden der Ultrafiltration bestimmt. ⓘ
Der bislang älteste – indirekte – Beleg für eine durch Viren verursachte Erkrankung wurde aus den deformierten Knochen eines 150 Millionen Jahre alten, kleinen zweibeinigen Dinosauriers (Dysalotosaurus lettowvorbecki) abgeleitet, der im Berliner Museum für Naturkunde verwahrt wird und Symptome von Osteodystrophia deformans aufweist, die auf eine Infektion mit Paramyxoviren zurückgeführt werden. ⓘ
Eigenschaften
Systematische Stellung
Viren sind im Wesentlichen bloße stoffliche Programme zu ihrer eigenen Reproduktion in Form einer Nukleinsäure. Sie besitzen zwar spezifische genetische Informationen, aber nicht den für ihre Replikation notwendigen Synthese-Apparat. Ob Viren als Lebewesen bezeichnet werden können, ist abhängig von der Definition von Leben. Eine allgemein anerkannte, unwidersprochene Definition gibt es bislang nicht. Die meisten Wissenschaftler stufen Viren nicht als Lebewesen ein – wobei die wissenschaftliche Diskussion noch nicht abgeschlossen ist, da beispielsweise bei der Genomgröße des Cafeteria-roenbergensis-Virus eine Abgrenzung anhand der Größe des Genoms zu verwischen beginnt. ⓘ
Viren werden normalerweise auch nicht zu den Parasiten gerechnet, weil Parasiten Lebewesen sind. Einige Wissenschaftler betrachten Viren dennoch als Parasiten, weil sie einen Wirtsorganismus infizieren und seinen Stoffwechsel für ihre eigene Vermehrung benutzen. Diese Forscher definieren Viren als „obligat intrazelluläre Parasiten“ (Lebensformen, die immer Parasiten innerhalb einer Zelle sind), die mindestens aus einem Genom bestehen und zur Replikation eine Wirtszelle benötigen. Man kann sich – unabhängig von der Klassifizierung als Lebewesen oder Nicht-Lebewesen – darauf einigen, dass das Verhalten von Viren dem von gewöhnlichen Parasiten sehr ähnlich ist. Viren können wie Prionen, funktionslose DNA-Sequenzen und Transposons in diesem Sinne als „parasitär“ bezeichnet werden. ⓘ
Koevolution
Eine Beobachtung bei der Pathogenese in natürlichen Wirten ist, dass an den Wirt angepasste Krankheitserreger ihm meist nicht sehr schaden, da sie ihn für ihre eigene Entwicklung benötigen und das Immunsystem durch Zellschäden und Apoptose aktiviert wird. Die Vermeidung einer Immunreaktion erleichtert die Replikation und die Übertragung (synonym Transmission) an weitere Wirte. Manche Viren verbleiben lebenslang im Körper. Es kann von Zeit zu Zeit zu einer Reaktivierung kommen, auch ohne Symptome. (Siehe hierzu auch: Erregerpersistenz.) So erreichen beispielsweise Herpes-simplex-Viren Infektionsquoten (synonym Durchseuchung) von über 90 % der deutschen Bevölkerung mit wenig ausgeprägten Symptomen. Das simiane Immundefizienz-Virus erzeugt in seinen natürlichen Wirten kein AIDS, im Gegensatz zu HIV im Menschen. Dagegen löschen sich Infektionen mit Ebolavirus im Menschen, nicht aber in ihren natürlichen Wirten, gelegentlich durch ihre hohe Virulenz selbst aus, bevor eine effiziente Transmission erfolgt, da der Wirt stark geschwächt ist und bald verstirbt, folglich ist sein Bewegungsradius und somit die Verbreitung des Virus begrenzt. Ein schwerer Infektionsverlauf mit hoher Sterblichkeit (siehe Letalität und Mortalität) ist zumeist ein Anzeichen dafür, dass der verursachende Erreger noch nicht an den betreffenden Organismus als seinen Reservoirwirt angepasst ist. Jedoch ist der Übergang von Pathogenen mit einer hohen Replikation (und erzeugten Schäden) zu einer dauerhaften Infektionsquote (Infect and persist, unter Vermeidung von Schäden) fließend. Anders ausgedrückt, neigen angepasste infektiöse Objekte zur Persistenz und einer regulierten Reproduktionsrate, während weniger angepasste Pathogene tendenziell zur vorzeitigen Beendigung der Infektionskette führen. Ausnahmen sind z. B. H5N1-Viren in Vögeln und humane Pockenviren im Menschen. Die Anpassung erfolgt jedoch meistens seitens des Wirts, da die Pathogene mit ihren Artgenossen in Konkurrenz stehen und ein weniger reproduktives Pathogen schneller untergehen würde. Daher tritt eine Minderung der Pathogenität bei Pathogenen vor allem in Verbindung mit einer erhöhten Reproduktionsrate auf. ⓘ
Die Anpassung des Wirts an das Pathogen wird als Wirtsrestriktion oder -resistenz bezeichnet. Zu den bekannten antiviralen und antibakteriellen Mechanismen gehören also, wie schon unter Wirtsreaktionen bei Eukaryoten ausgeführt, beim Menschen beispielsweise der Myxovirus-Resistenzfaktor Mx1, die PAMP-Rezeptoren, der dsRNA-aktivierte Inhibitor der Translation DAI, das MDA5, die Oligoadenylatsynthase OAS1, das Langerin, das Tetherin, das APOBEC3, das TRIM5alpha und die Proteinkinase R. Darüber hinaus erfolgt die Immunantwort. ⓘ
Einteilung
Herkömmliche Virusklassifikation
Im Jahre 1962 wurde von André Lwoff, Robert W. Horne und Paul Tournier entsprechend der von Carl von Linné begründeten binären Klassifikation der Lebewesen eine Taxonomie der Viren („LHT-System“) eingeführt, die folgende Stufen umfasst (Muster für Namensendungen der Taxa in Klammern):
- Virosphäre (Phylum: Vira)
- Subphylum (…vira)
- Klasse (Biologie) (…ica)
- Ordnung (…virales)
- Familie (…viridae)
- Unterfamilie (…virinae)
- Gattung oder Genus (…virus)
- Art oder Species (nach der hervorgerufenen <Krankheit> …virus) ⓘ
- Gattung oder Genus (…virus)
- Unterfamilie (…virinae)
- Familie (…viridae)
- Ordnung (…virales)
- Klasse (Biologie) (…ica)
- Subphylum (…vira)
Damit einher geht eine Zuordnung in Gruppen, die sich an den Wirten orientieren
- Bakterien und Archaeen (Befall durch Bakteriophagen/Archaeophagen)
- Algen, Pilze und Protozoen
- Pflanzen (Befall auch durch Viroide)
- Tiere, mit drei Untergruppen:
- wirbellose Tiere (Invertebraten)
- Wirbeltiere (Vertebraten)
- Vertreter beider Gruppen ⓘ
Die meisten Viren gehören nur zu einer der obigen vier Gruppen, doch Virusarten der Familie Rhabdoviridae und Bunyaviridae können sowohl Pflanzen als auch Tiere infizieren. Einige Viren vermehren sich nur in Vertebraten, werden jedoch auch von Invertebraten mechanisch übertragen (siehe Vektor), vor allem von Insekten. Viren, die auf die Nutzung von Genen anderer Viren (Mamaviren) während der gemeinsamen Infektion einer Wirtszelle angewiesen sind, werden Virophagen genannt. ⓘ
Schreibweise der Virusartnamen
Der offizielle internationale, wissenschaftliche Name eines Virus ist die englischsprachige Bezeichnung, nach der sich stets auch die international gebräuchliche Abkürzung richtet, wie bei Lagos bat virus (LBV). Diese Abkürzung wird unverändert auch im Deutschen verwendet. Folgerichtig lautet die Abkürzung für die deutsche Virusbezeichnung Lagos-Fledermaus-Virus ebenfalls LBV. ⓘ
In den englischen Virusnamen wie zum Beispiel bei West Nile virus werden normalerweise keine Bindestriche benutzt und virus wird kleingeschrieben. Der Bindestrich taucht im Englischen nur bei Adjektiven auf, also bei Tick-borne encephalitis virus oder Avian encephalomyelitis-like virus. ⓘ
Im Deutschen werden die Virusnamen teilweise mit Bindestrichen geschrieben, also West-Nil-Virus, Hepatitis-C-Virus, Humanes Herpes-Virus, Lagos-Fledermaus-Virus, Europäisches Fledermaus-Lyssa-Virus, teilweise auch zusammen. Vor den Nummern von Subtypen steht (wie im Englischen) ein Leerzeichen, bei den Abkürzungen ein Bindestrich, z. B. Europäisches Fledermaus-Lyssa-Virus 1 (EBLV-1), Herpes-simplex-Virus 1 (HSV-1) und Humanes Herpes-Virus 1 (HHV-1). ⓘ
Im Gegensatz zur belebten Welt werden nach den Regeln des ICTV auch die Namen der höheren Taxa (wie Familie, Ordnung, Klasse, Phylum (Abteilung) etc. kursiv geschrieben. Sie werden gewöhnlich nicht übersetzt. Im Gegensatz dazu werden Bezeichnungen für Subtypen (Stämme – im Sinn von englisch strains) – und Isolate) nie kursiv gesetzt (auch nicht evtl. darin enthaltene Gattungs- und/oder Artnamen ihrer Wirte). ⓘ
Werden häufig gebrauchte Namen einmal doch übersetzt (wie Coronaviren für Coronaviridae), dann werden diese Bezeichnungen nicht kursiv gesetzt. Da sich oft Taxa in aufsteigender Rangfolge nur durch die Endung unterscheiden (wie Herpesviridae und Herpesvirales), entstehen allerdings bei der Verwendung eingedeutschter Namen (hier: Herpesviren) oft Mehrdeutigkeiten. Das gilt auch für die Verwendung solcher Namen (z. B. Coronaviren für die prominentesten Vertreter wie SARS-CoV-2). ⓘ
Behüllte Viren
Doppelsträngige DNA-Viren (dsDNA)
- Familie Poxviridae
- Unterfamilie Chordopoxvirinae
- Gattung Orthopoxvirus
- Orthopoxvirus variola = Variolavirus – Pocken, Echte Pocken
- Orthopoxvirus variola var. alastrim = Kaffernpockenvirus – Pocken, Weiße Pocken
- Monkeypoxvirus (MPV) = Orthopoxvirus simiae – Affenpocken; auch auf den Menschen übertragbar, Symptome wie bei Menschenpocken, aber deutlich milder
- Gattung Parapoxvirus
- Parapoxvirus ovis = Orf-Virus – Orf
- Gattung Molluscipoxvirus
- Molluscum-Contagiosum-Virus – Dellwarze (Molluscum contagiosum)
- Gattung Orthopoxvirus
- Unterfamilie Chordopoxvirinae
- Familie Herpesviridae
- Unterfamilie Alphaherpesvirinae
- Gattung Simplexvirus
- Herpes-simplex-Virus 1 (HSV-1) = Humanes Herpes-Virus 1 (HHV-1) – Herpes simplex, Herpes labialis, Stomatitis aphtosa
- Herpes-simplex-Virus 2 (HSV-2) = Humanes Herpes-Virus 2 (HHV-2) – Herpes simplex, Herpes genitalis
- Herpes-B-Virus = (Herpesvirus simiae)
- Gattung Varicellovirus
- Varizella-Zoster-Virus (VZV) = Humanes Herpes-Virus 3 (HHV-3) – Windpocken = Varizellen (Herpes zoster), Gürtelrose
- suid Herpesvirus Typ 1 (SHV-1) = Pseudowut-Virus, Aujeszky-Virus u. a. – Aujeszkysche Krankheit = Pseudowut, Juckseuche, Tollkrätze u. a. (bei Tieren, mit geringer Pathogenität auch auf den Menschen übertragbar)
- Gattung Simplexvirus
- Unterfamilie Betaherpesvirinae
- Gattung Cytomegalovirus
- Humanes Cytomegalievirus (HCMV) = Humanes Zytomegalievirus (HZMV) = Humanes Herpes-Virus 5 (HHV-5) – Zytomegalie
- Gattung Reseolovirus
- Humanes Herpesvirus 6 (HHV-6) – Drei-Tage-Fieber
- Humanes Herpesvirus 7 (HHV-7) – Drei-Tage-Fieber
- Gattung Cytomegalovirus
- Unterfamilie Gammaherpesvirinae
- Gattung Lymphocryptovirus
- Epstein-Barr-Virus (EBV) = Humanes Herpes-Virus 4 (HHV-4) – Pfeiffer-Drüsenfieber, Burkitt-Lymphom
- Gattung Rhadinovirus
- Humanes Herpes-Virus 8 (HHV-8) – Kaposi-Sarkom
- Gattung Lymphocryptovirus
- Unterfamilie Alphaherpesvirinae
- Familie Hepadnaviridae
- Gattung Orthohepadnavirus
- Hepatitis-B-Virus (HBV) – Hepatitis B ⓘ
- Gattung Orthohepadnavirus
Einzel(+)-Strang-RNA-Viren (ss(+)RNA)
- Familie Togaviridae
- Gattung Alphavirus – Erreger von Arbovirosen
- Barmah-Forest-Virus – Barmah-Forest-Fieber mit grippeähnlichen Symptomen, epidemische Polyarthritis
- Chikungunya-Virus (CHIKV) – Chikungunya-Fieber
- Eastern-Equine-Encephalitis-Virus (EEEV) = Östliches-Pferdeenzephalitis-Virus – Übertragung durch Stechmücken auch auf den Menschen möglich (selten!) → Östliche Pferdeenzephalomyelitis (Enzephalitis/ Enzephalomyelitis)
- Western-Equine-Encephalitis-Virus (WEEV) = Westliches-Pferdeenzephalitis-Virus – Übertragung durch Stechmücken auch auf den Menschen möglich (selten!) → Westliche Pferdeenzephalomyelitis (Enzephalitis/ Enzephalomyelitis)
- Everglades-Virus – Everglades-Fieber
- O'nyong-nyong-Virus (ONNV) – O’nyong-nyong-Fieber
- Mayaro-Fieber-Virus (MAYV) – Mayaro-Fieber
- Semliki-Forest-Virus (SFV) – Semliki-Forest-Fieber
- Mucambo-Virus – Mucambo-Fieber
- Ross-River-Virus (RRV) – Ross-River-Fieber
- Sindbis-Virus (SINV) – Sindbis-Fieber (Gelenkentzündung [„epidemische Polyarthritis“], zum Teil mit Hautausschlägen und selten mit Enzephalitis)
- Gattung Rubiviren
- Rubivirus = Rötelnvirus = Rubellavirus (RUBV) – Röteln
- Gattung Alphavirus – Erreger von Arbovirosen
- Familie Flaviviridae
- Gattung Hepacivirus
- Hepatitis-C-Virus (HCV) – Hepatitis C
- GB-Virus-C (ohne Krankheitswert)
- Gattung Flavivirus
- West-Nil-Virus (WNV) – West-Nil-Fieber
- Dengue-Virus (DENV) – Dengue-Fieber
- Gelbfieber-Virus (YFV) – Gelbfieber
- Louping-ill-Virus (LIV) – Louping-ill-Enzephalitis
- St.-Louis-Enzephalitis-Virus (SLEV) – St.-Louis-Enzephalitis
- Japan-Enzephalitis-Virus (JEV) – Japanische Enzephalitis
- Usutu-Virus (USUV) – unspezifische Symptome wie Fieber und/oder Hautausschläge
- Kyasanur-Forest-Disease-Virus (KFDV) – Kyasanur-Wald-Fieber
- Powassan-Virus (POWV) – Powassan-Enzephalitis
- FSME-Virus [englisch: tick-borne encephalitic virus (TBEV)] – FSME (Frühsommer-Meningoenzephalitis)
- Subtyp European / Western tick-borne encephalitis virus (WTBEV)
- Subtyp Siberian tick-borne encephalitis virus (STBEV)
- Subtyp Far-Eastern tick-borne encephalitis virus (Far-Eastern TBEV); ehemals Russian-Spring-Summer-Enzephalitis-Virus (RSSEV) – RSSE, auch RFSE (Russian-Spring-Summer-Enzephalitis, Russische Frühsommerenzephalitis)
- Zika-Virus (ZIKV) (2 Hauptgruppen; diverse Subtypen) – meist nur Hautausschlag, Fieber, Gelenkschmerzen, Konjunktivitis
- Gattung Hepacivirus
- Familie Coronaviridae
- Unterfamilie Orthocoronavirinae
- Gattung Alphacoronavirus
- Untergattung Duvinacovirus
- Humanes Coronavirus 229E (HCoV-229E) – Erkältung
- Untergattung Setracovirus
- Humanes Coronavirus NL63 (HCoV-NL63) – Erkältung
- Untergattung Duvinacovirus
- Gattung Betacoronavirus
- Untergattung Embecovirus
- Spezies Betacoronavirus 1
- Subspezies Humanes Coronavirus OC43 (HCoV-OC43) – Erkältung; gelegentlich auch schwere Infektion der Atemwege, Pneumonie
- Spezies Humanes Coronavirus HKU1 (HCoV-HKU1) – Erkältung
- Spezies Betacoronavirus 1
- Untergattung Merbecovirus
- Middle East respiratory syndrome coronavirus (MERS-CoV) – grippeähnliche Symptome, schwere Infektion der Atemwege, Pneumonie und ggf. Nierenversagen
- Untergattung Sarbecovirus
- SARS-assoziiertes Coronavirus (SARS-CoV) – SARS (atypische Lungenentzündung Pneumonie), mit Subtyp
- Subtyp SARS-CoV-2 (eng. 2019-novel Coronavirus, 2019-nCoV, bzw. Wuhan seafood market pneumonia virus) – COVID-19: Infektion der unteren Atemwege bis zur Lungenentzündung
- SARS-assoziiertes Coronavirus (SARS-CoV) – SARS (atypische Lungenentzündung Pneumonie), mit Subtyp
- Untergattung Embecovirus
- Gattung Alphacoronavirus
- Unterfamilie Torovirinae
- Gattung Torovirus
- diverse Arten – Gastroenteritis
- Gattung Torovirus
- Unterfamilie Orthocoronavirinae
- Familie Retroviridae – Einzel(+)-Strang-RNA-Viren mit dsDNA-Zwischenstufe
- Unterfamilie Orthoretrovirinae
- Gattung Deltaretrovirus
- Humanes T-lymphotropes Virus 1 (HTLV-1) – Adulte T-Zell-Leukämie, Tropische Spastische Paraparese
- Humanes T-lymphotropes Virus 2 (HTLV-2) – Leukämie (?)
- Humanes T-lymphotropes Virus 3 (HTLV-3) – unbekannt
- Humanes T-lymphotropes Virus 4 (HTLV-4) – unbekannt
- Gattung Lentivirus
- Humanes Immundefizienz-Virus Typ 1 (HIV-1) – AIDS
- Humanes Immundefizienz-Virus Typ 2 (HIV-2) – AIDS ⓘ
- Gattung Deltaretrovirus
- Unterfamilie Orthoretrovirinae
Einzel(−)-Strang-RNA-Viren (ss(−)RNA)
- Familie Arenaviridae
- Gattung Mammarenavirus
- Untergattung LCMV/Lassa-Komplex (Altwelt-Arenaviren)
- Lassa-Virus (Lassa mammarenavirus / LASV) – Lassa-Fieber
- lymphozytäre-Chorio-Meningitis-Virus (Lymphocytic choriomeningitis Virus / Lymphocytic choriomeningitis mammarenavirus / LCMV) – Lymphozytäre Choriomeningitis
- Mopeia-Virus (MOPV) - Mopeia-Fieber (hämorrhagisches Fieber mit teilweise sehr schwerem bis tödlichem Verlauf.)
- Lujo-Virus (Lujo mammarenavirus / LUJV) - Lujo-Fieber (hämorrhagisches Fieber)
- Untergattung Tacaribe-Komplex: (Neuwelt-Arenaviren)
- Chapare-Virus (Chapare mammarenavirus / CHAV) – Hämorrhagisches Fieber
- Tacaribe-Virus (Tacaribe mammarenavirus / TCRV) – Hämorrhagisches Fieber
- Junin-Virus (Argentinian mammarenavirus / JUNV) – Junin-Fieber (argentinisches hämorrhagisches Fieber / AHF)
- Machupo-Virus (Machupo mammarenavirus / MACV) – Machupo-Fieber (bolivianisches hämorrhagisches Fieber / BHF mit hoher Letalität.)
- Guanarito-Virus (Guanarito mammarenavirus / GTOV) - Guanarito-Fieber (venezolanisches hämorrhagisches Fieber / VHF)
- Sabia-Virus (SPH 114202 virus / Brazilian mammarenavirus / Sabiá mammarenavirus / SABV) - Sabia-Fieber (brasilianisches hämorrhagisches Fieber / BzHF)
- Untergattung LCMV/Lassa-Komplex (Altwelt-Arenaviren)
- Gattung Mammarenavirus
- Familie Bornaviridae
- Gattung Orthobornavirus (früher Bornavirus)
- Spezies Mammalian 1 orthobornavirus
- Virus der Bornaschen Krankheit (engl. Borna disease virus 1 = BoDV-1) – Erreger der Borna-Krankheit bei Pferden, Schafen und anderen Säugetieren, in seltenen Fällen auch auf den Menschen übertragbar – schwere (Enzephalitiden)
- Spezies Mammalian 2 orthobornavirus
- Bunthörnchen-Bornavirus 1 (engl. variegated squirrel Bornavirus 1 = VSBV-1) bei Bunthörnchen (Sciurus variegatoides) nachgewiesen, auch auf den Menschen übertragbar – potenziell tödlich verlaufende Encephalitis
- Spezies Mammalian 1 orthobornavirus
- Gattung Orthobornavirus (früher Bornavirus)
- Familie Bunyaviridae – Erreger von Arbovirosen
- Gattung Orthobunyavirus
- Bunyamwera-Virus (Serogruppe)
- Batai-Virus (BATV) – grippeähnliche Symptome und Hautausschläge
- California-Encephalitis-Virus (Serogruppe) – Encephalitis
- Gattung Phlebovirus
- Rift-Valley-Fieber-Virus (3 Subtypen) – Rift-Tal-Fieber
- Sandmückenfieber-Virus (SFNV) – Sandfly fever = Sandmückenfieber
- Subtyp Karimabad-Virus (KARV)
- Subtyp Sandmückenfieber-Virus Sabin (SFNV-Sabin)
- Subtyp Teheran-Virus (THEV)
- Subtyp Toscana-Virus (TOSV) – Pappataci-Fieber
- Serotypen: Toskana (T), Sizilien (S) und Neapel (N)
- Gattung Nairovirus
- Krim-Kongo-Fieber-Virus (Serogruppe):
- Subtyp Krim-Kongo-Hämorrhagisches-Fieber-Virus (CCHFV) – Krim-Kongo-Fieber
- Subtyp Hazara-Virus (HAZV) – Krim-Kongo-Fieber
- Subtyp Khasan-Virus (KHAV) – Krim-Kongo-Fieber
- Krim-Kongo-Fieber-Virus (Serogruppe):
- Gattung Hantavirus
- Hantaan-Virus (4 Subtypen) – hämorrhagisches Fieber, Nephritis
- Seoul-Virus (Serogruppe) – hämorrhagisches Fieber
- Prospect-Hill-Virus (2 Subtypen) – hämorrhagisches Fieber
- Puumala-Virus (Serogruppe) – hämorrhagisches Fieber, Pneumonie, Nephritis
- Dobrava-Belgrad-Virus – hämorrhagisches Fieber
- Tula-Virus – hämorrhagisches Fieber
- Sin-Nombre-Virus (Serogruppe) – hämorrhagisches Fieber mit schwerem Lungenödem
- Gattung Orthobunyavirus
- Familie Filoviridae
- Gattung Marburg-Virus
- Lake-Victoria-Marburgvirus (Serogruppe) – Marburg-Fieber (hämorrhagisches Fieber)
- Gattung Ebolavirus
- Zaire-Ebolavirus (ZEBOV) Serogruppe – Ebolafieber (hämorrhagisches Fieber)
- Sudan-Ebolavirus (SEBOV) Serogruppe – Ebolafieber (hämorrhagisches Fieber)
- Reston-Ebolavirus (REBOV) Serogruppe – nicht humanpathogen, nur bei Makaken und Schweinen hämorrhagisches Fieber
- Côte d'Ivoire-Ebolavirus (CIEBOV) Serogruppe – Ebolafieber (hämorrhagisches Fieber)
- Bundibugyo-Ebolavirus (BEBOV) Serogruppe – Ebolafieber (hämorrhagisches Fieber)
- Gattung Marburg-Virus
- Familie Orthomyxoviridae
- Gattung Influenzavirus A – Influenza (Grippe)
- Influenzavirus A-Variante H1N1 – Influenza (Grippe)
- Influenzavirus A-Variante H3N2 – Influenza (Grippe)
- (aviäres) Influenzavirus-A-Variante H5N1, hoch pathogenes aviäres Influenzavirus (HPAIV) – „Vogelgrippe“, bei Tieren, auch auf den Menschen übertragbar, aber kaum von Mensch zu Mensch.
- Gattung Influenzavirus B – Influenza (Grippe)
- Influenzavirus B/Victoria-Linie – Influenza (Grippe)
- Influenzavirus B/Yamagata-Linie – Influenza (Grippe)
- Gattung Influenzavirus C – Influenza (Grippe)
- Gattung Influenzavirus D – Influenza (Grippe)
- Gattung Influenzavirus A – Influenza (Grippe)
- Familie Paramyxoviridae
- Gattung Avulavirus
- Humanes Parainfluenzavirus (Typ 1, 3) – Erkältung, Parainfluenza
- Gattung Morbillivirus
- Masernvirus – Masern
- Gattung Henipavirus
- Hendra-Virus, (früher Equines Morbillivirus) – Pneumonie; Enzephalitis
- Nipah-Virus – Pneumonie; Enzephalitis
- Gattung Rubulaviren
- Humanes Parainfluenzavirus (Typ 2, 4) – Erkältung, Parainfluenza
- Mumpsvirus – Mumps
- Gattung Avulavirus
- Familie Pneumoviridae
- Gattung Orthopneumovirus (früher: Pneumovirus)
- Humanes Respiratorisches Synzytial-Virus (HRSV) (Typ A, B) – Atemwegsinfektion, Erkältung
- Gattung Metapneumovirus
- Humanes Metapneumovirus (HMPV) (Typ A1 bis 2, B1 bis 2) – Atemwegsinfektion, Erkältung
- Gattung Orthopneumovirus (früher: Pneumovirus)
- Familie Rhabdoviridae
- Gattung Vesiculovirus
- Vesicular-Stomatitis-Indiana-Virus (VSV) – Stomatitis vesicularis (Mundschleimhautentzündung mit Bläschenbildung) bei Tieren, auch auf den Menschen übertragbar
- Gattung Lyssavirus
- Rabiesvirus (RABV) (ehemals Genotyp 1) = Tollwutvirus – Tollwut, bei Tieren, auch auf den Menschen übertragbar
- Mokola-Virus (MOKV) (ehemals Genotyp 3) – Tollwut, bei Tieren, auch auf den Menschen übertragbar
- Duvenhage-Virus (DUVV) (ehemals Genotyp 4) – Tollwut, bei Tieren, auch auf den Menschen übertragbar
- Europäisches Fledermaus-Lyssa-Virus 1 + 2 (EBLV-1, -2) (ehemals Genotypen 5 und 6) – Tollwut, bei Tieren, auch auf den Menschen übertragbar
- Australisches Fledermaus-Lyssa-Virus (ABLV) (ehemals Genotyp 7) – Tollwut, bei Tieren, auch auf den Menschen übertragbar ⓘ
- Gattung Vesiculovirus
Unbehüllte Viren
Doppelsträngige DNA-Viren (dsDNA)
- Familie Adenoviridae
- Familie Polyomaviridae
- Gattung Polyomavirus
- BK Polyomavirus (BKPyV) = BK-Virus (BKV) = Polyomavirus hominis Typ 1 – führt bei immunsuppressiver Behandlung nach Transplantation ev. zum Verlust des Transplantates
- JC Polyomavirus (JCPyV) = JC-Virus (JCV) = Polyomavirus hominis Typ 2 – bei zellulär Immunsupprimierten (AIDS) zu Progressiver multifokalen Leukoenzephalopathie (PML)
- Gattung Polyomavirus
- Familie Papillomaviridae
- Gattung Papillomavirus
- Untergattung Humane Papillomviren
- diverse Humane Papillomviren (HPV) – Warzen;
- Kondyloma-Virus 6 (HPV-6) – Feigwarzen; sehr selten: Rezidivierende respiratorische Papillomatose (multiples, rezidivierendes Auftreten von meist gutartigen Plattenepithelpapillomen im Kehlkopf bis in Luftröhre (Trachea) und Lunge.
- Kondyloma-Virus 11 (HPV-11) – Feigwarzen; sehr selten: Rezidivierende respiratorische Papillomatose (multiples, rezidivierendes Auftreten von meist gutartigen Plattenepithelpapillomen im Kehlkopf bis in Luftröhre (Trachea) und Lunge).
- Humanes Papillomvirus 16 /18 /30 … (HPV-16 /-18 /-30 …) – Zervixkarzinom (Gebärmutterhalskrebs) ⓘ
- Untergattung Humane Papillomviren
- Gattung Papillomavirus
Einzelsträngige DNA-Viren (ssDNA)
- Familie Parvoviridae
- Unterfamilie Parvovirinae
- Gattung Dependoparvovirus (alias Dependovirus)
- Spezies Adenoassoziiertes Virus A (AAV-A)
- Adenoassoziiertes Virus 1 bis 4 (AAV-1 bis AAV-4)
- Spezies Adenoassoziiertes Virus B (AAV-B)
- Adenoassoziiertes Virus 5 (AAV-5)
- Gattung Erythroparvovirus (alias Erythrovirus)
- Spezies Primate erythroparvovirus 1
- Parvovirus B19 – Ringelröteln ⓘ
Doppelsträngige RNA-Viren (dsRNA)
- Familie Reoviridae
- Gattung Rotavirus
- diverse Arten – Gastroenteritis mit Durchfall
- Gattung Coltivirus
- Colorado-Tick-Fever-Virus – Colorado-Zeckenfieber ⓘ
- Gattung Rotavirus
Einzel(+)-Strang-RNA-Viren (ss(+)RNA)
- Familie Caliciviridae
- Gattung Norovirus
- Norovirus (NV) = Norwalk-Like-Virus (NLV)
- Humane Noroviren der Gruppen GGI, GGII und GGIV – Brechdurchfall = Gastroenteritis
- Norovirus (NV) = Norwalk-Like-Virus (NLV)
- Gattung Sapovirus
- Sapovirus (SV) – Gastroenteritis
- Gattung Norovirus
- Familie Hepeviridae
- Gattung Hepevirus
- Hepatitis-E-Virus (HEV) – Hepatitis E
- Gattung Hepevirus
- Familie Picornaviridae
- Gattung Enterovirus
- Poliovirus Typ 1–3 – Kinderlähmung
- Coxsackievirus A/B – von Erkältung bis Meningitis, Pankreatitis oder Myocarditis, selten auch Lähmungen
- Coxsackievirus B1 (CVB-1) bis B 6 – Erkältung
- Echovirus – Exantheme Enantheme, Infektionen des oberen Respirationstrakts (Erkältung), Herpangina, Myoperikarditis, verstreute (disseminierte) Infektion bei Neugeborenen, chronische Meningoenzephalitis bei immunsupprimierten Patienten, Meningitis, Enzephalitis selten Paralyse
- Enterovirus
- Humane Enteroviren – Erkältung
- Humanes Enterovirus 70 (EV-70) – akute hämorrhagische Konjunktivitis
- Humanes Enterovirus 71 (EV-71) – Meningoenzephalitis, Hautausschlag und Poliomyelitis ähnliches Syndrom = Hand-Fuß-Mund-Krankheit
- Humane Enteroviren – Erkältung
- Gattung Hepatovirus
- Hepatitis-A-Virus – Hepatitis A
- Gattung Rhinovirus
- Rhinovirus
- Humane Rhinoviren-1 A (HRV-1 A) oder 1 B bis 100 – Erkältung ⓘ
- Rhinovirus
- Gattung Enterovirus
Therapie mit Viren
Aktuell wird verstärkt an Therapien geforscht, bei denen Viren zur Heilung von Krankheiten eingesetzt werden. Diese Forschungen umfassen den Einsatz viraler Vektoren unter anderem als onkolytische Viren zur Bekämpfung von Tumoren, als Phagentherapie zur gezielten Infektion und Lyse von zum Teil antibiotikaresistenten Bakterien, als Impfstoff zur Prophylaxe und Therapie von Infektionskrankheiten, zur Erzeugung von induzierten pluripotenten Stammzellen oder zur Gentherapie von Gendefekten. ⓘ
Siehe auch
- Endogenes Retrovirus
- Extremophile
- :Kategorie:Virologe ⓘ
Literatur
- Ältere Literatur
- Feodor Lynen: Das Virusproblem. In: Angewandte Chemie. Band 51. Nr. 13, 1938, ISSN 0044-8249, S. 181–185. ⓘ
- Aktuelle Literatur
- Hans W. Doerr, Wolfram H. Gerlich (Hrsg.): Medizinische Virologie – Grundlagen, Diagnostik und Therapie virologischer Krankheitsbilder. Thieme, Stuttgart/ New York 2002, ISBN 3-13-113961-7.
- Walter Doerfler: Viren. Fischer Taschenbuch Verlag, Frankfurt a. M. 2002, ISBN 3-596-15369-7.
- Dietrich Falke, Jürgen Bohl u. a.: Virologie am Krankenbett: Klinik, Diagnostik, Therapie. Springer, Heidelberg u. a. 1998, ISBN 3-540-64261-7. (mit Literaturangaben)
- Matthias Eckoldt Virus: Partikel, Paranoia, Pandemien. Ecowin, Salzburg/ München 2021, ISBN 978-3-7110-0275-4.
- Dietrich Falke, Jürgen Podlech: Viren. In: Peter Reuter: Springer Lexikon Medizin. Springer, Berlin u. a. 2004, ISBN 3-540-20412-1, S. 2273–2282.
- S. J. Flint, L. W. Enquist, V. R. Racaniello (Hrsg.): Principles of Virology. 2. Auflage, ASM Press, Washington DC 2003, ISBN 1-55581-259-7.
- Alfred Grafe: Viren – Parasiten unseres Lebensraumes. Springer, Berlin/ Heidelberg/ New York 1977, ISBN 3-540-08482-7.
- David M. Knipe, Peter M. Howley et al. (Hrsg.): Fields’ Virology. 2 Bände, 5. Auflage, Lippincott Williams & Wilkins, Philadelphia 2007, ISBN 978-0-7817-6060-7 (Standardwerk der Virologie).
- Arnold J. Levine: Viren: Diebe, Mörder und Piraten. Spektrum Akademischer Verlag, Heidelberg 1992, ISBN 3-86025-073-6.
- Susanne Modrow, Dietrich Falke, Uwe Truyen: Molekulare Virologie. Eine Einführung für Biologen und Mediziner (= Spektrum-Lehrbuch). 2. Auflage, Spektrum Akademischer Verlag, Heidelberg 2002, ISBN 3-8274-1086-X.
- Stephen S. Morse: The Evolutionary Biology of Viruses. Raven Press, New York 1994, ISBN 0-7817-0119-8.
- Sven P. Thoms: Ursprung des Lebens: wie und wann entstand Leben auf der Erde? ... (= Fischer-Taschenbücher; Fischer kompakt.). Fischer Taschenbuch Verlag, Frankfurt a. M. 2005, ISBN 3-596-16128-2.
- Luis P. Villarreal: Viruses and the Evolution of Life. ASM Press, Washington 2005, ISBN 1-55581-309-7.
- Ernst-Ludwig Winnacker: Viren: Die heimlichen Herrscher. Wie Grippe, Aids und Hepatitis unsere Welt bedrohen. Eichborn, Frankfurt a. M. 1999, ISBN 3-8218-1598-1.
- Gottfried Schuster: Viren in der Umwelt. Teubner, Stuttgart 1998, ISBN 3-519-00209-4.
- Dorothy H. Crawford: The invisible enemy: a natural history of viruses. Oxford University Press, Oxford 2002, ISBN 0-19-856481-3.
- Brian W. Mahy: The dictionary of virology. Elsevier, Amsterdam 2008, ISBN 0-12-373732-X.
- Susanne Modrow: Viren: Grundlagen, Krankheiten, Therapien. [Eine allgemeinverständliche Einführung für medizinische Laien]. Beck, München 2001. ISBN 3-406-44777-5.
- Hartwig Klinker: Infektionen durch Viren. In: Marianne Abele-Horn (Hrsg.): Antimikrobielle Therapie. Entscheidungshilfen zur Behandlung und Prophylaxe von Infektionskrankheiten. (Unter Mitarbeit von Werner Heinz, Hartwig Klinker, Johann Schurz und August Stich) 2., überarbeitete und erweiterte Auflage, Peter Wiehl, Marburg 2009, ISBN 978-3-927219-14-4, S. 297–307.
- Marilyn J. Roossinck: Viren! Helfer, Feinde, Lebenskünstler – in 101 Porträts. Springer, Berlin 2018, ISBN 978-3-662-57543-7.
- Sunit K. Singh (Hrsg.): Viral Infections and Global Change. [Über den Einfluss der Globalisierung und des Klimawandels auf die Verbreitung und Übertragung von Viren, speziell tropischen Viren]. Wiley-Blackwell, Hoboken NJ 2014, ISBN 978-1-118-29787-2 (Print); ISBN 978-1-118-29809-1 (eBook).
- Eugene V. Koonin, Tatiana G. Senkevich, Valerian V. Dolja: The ancient Virus World and evolution of cells. In: Biology Direct. 19. September 2006, Band 1, Artikel 29, doi:10.1186/1745-6150-1-29, PMID 16984643, PMC 1594570 (freier Volltext) (englisch). ⓘ
Film
- Wie Viren unseren Körper angreifen – Abwehrkampf im Inneren der Zelle. Dokumentation von Mike Davies, Wide-Eyed Entertainment für BBC, 2012, 46 min, deutsch (Englisches Original: Battlefield Cell in der Internet Movie Database (englisch)) ⓘ