Desoxyribonukleinsäure

| Teil einer Serie über ⓘ |
| Genetik |
|---|
 |
|
Die Desoxyribonukleinsäure (/diːˈɒksɪˌraɪboʊnjuːˌkliːɪk, -ˌkleɪ-/ (![]() hören); DNA) ist ein Polymer, das aus zwei Polynukleotidketten besteht, die sich umeinander winden und eine Doppelhelix bilden, die die genetischen Anweisungen für die Entwicklung, das Funktionieren, das Wachstum und die Fortpflanzung aller bekannten Organismen und vieler Viren enthält. DNA und Ribonukleinsäure (RNA) sind Nukleinsäuren. Neben Proteinen, Lipiden und komplexen Kohlenhydraten (Polysacchariden) sind Nukleinsäuren eine der vier großen Arten von Makromolekülen, die für alle bekannten Lebensformen wesentlich sind. ⓘ
hören); DNA) ist ein Polymer, das aus zwei Polynukleotidketten besteht, die sich umeinander winden und eine Doppelhelix bilden, die die genetischen Anweisungen für die Entwicklung, das Funktionieren, das Wachstum und die Fortpflanzung aller bekannten Organismen und vieler Viren enthält. DNA und Ribonukleinsäure (RNA) sind Nukleinsäuren. Neben Proteinen, Lipiden und komplexen Kohlenhydraten (Polysacchariden) sind Nukleinsäuren eine der vier großen Arten von Makromolekülen, die für alle bekannten Lebensformen wesentlich sind. ⓘ
Die beiden DNA-Stränge werden als Polynukleotide bezeichnet, da sie aus einfacheren monomeren Einheiten, den Nukleotiden, zusammengesetzt sind. Jedes Nukleotid besteht aus einer von vier stickstoffhaltigen Nukleobasen (Cytosin [C], Guanin [G], Adenin [A] oder Thymin [T]), einem Zucker namens Desoxyribose und einer Phosphatgruppe. Die Nukleotide sind durch kovalente Bindungen (die so genannte Phospho-Diester-Bindung) zwischen dem Zucker eines Nukleotids und dem Phosphat des nächsten Nukleotids zu einer Kette verbunden, so dass ein abwechselndes Zucker-Phosphat-Grundgerüst entsteht. Die stickstoffhaltigen Basen der beiden getrennten Polynukleotidstränge sind nach den Regeln der Basenpaarung (A mit T und C mit G) durch Wasserstoffbrückenbindungen miteinander verbunden, so dass eine doppelsträngige DNA entsteht. Die komplementären Stickstoffbasen werden in zwei Gruppen unterteilt, die Pyrimidine und die Purine. In der DNA sind die Pyrimidine Thymin und Cytosin; die Purine sind Adenin und Guanin. ⓘ
Beide Stränge der doppelsträngigen DNA speichern die gleiche biologische Information. Diese Informationen werden repliziert, wenn sich die beiden Stränge trennen. Ein großer Teil der DNA (beim Menschen mehr als 98 %) ist nicht codierend, d. h. diese Abschnitte dienen nicht als Muster für Proteinsequenzen. Die beiden DNA-Stränge verlaufen in entgegengesetzter Richtung zueinander und sind somit antiparallel. An jeden Zucker ist eine von vier Arten von Nukleobasen (oder Basen) gebunden. Die Abfolge dieser vier Nukleobasen entlang des Rückgrats kodiert die genetische Information. Die RNA-Stränge werden auf der Grundlage von DNA-Strängen als Vorlage in einem als Transkription bezeichneten Prozess erstellt, bei dem die DNA-Basen gegen die entsprechenden Basen ausgetauscht werden, mit Ausnahme von Thymin (T), das durch Uracil (U) ersetzt wird. Im Rahmen des genetischen Codes spezifizieren diese RNA-Stränge die Sequenz der Aminosäuren in den Proteinen in einem Prozess, der Translation genannt wird. ⓘ
In eukaryontischen Zellen ist die DNA in langen Strukturen, den Chromosomen, angeordnet. Vor der typischen Zellteilung werden diese Chromosomen im Prozess der DNA-Replikation verdoppelt, so dass jede Tochterzelle einen vollständigen Chromosomensatz erhält. Eukaryotische Organismen (Tiere, Pflanzen, Pilze und Protisten) speichern den größten Teil ihrer DNA im Zellkern als Kern-DNA und einen Teil in den Mitochondrien als mitochondriale DNA oder in den Chloroplasten als Chloroplasten-DNA. Im Gegensatz dazu speichern Prokaryoten (Bakterien und Archaeen) ihre DNA nur im Zytoplasma, in zirkulären Chromosomen. Innerhalb der eukaryotischen Chromosomen verdichten und organisieren Chromatinproteine, wie z. B. Histone, die DNA. Diese verdichtenden Strukturen steuern die Wechselwirkungen zwischen der DNA und anderen Proteinen und tragen dazu bei, zu kontrollieren, welche Teile der DNA transkribiert werden. ⓘ

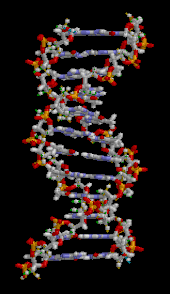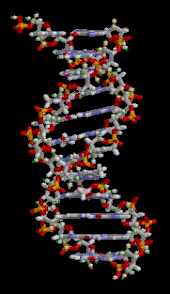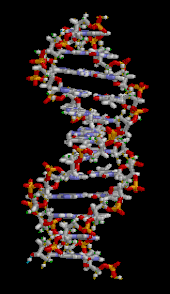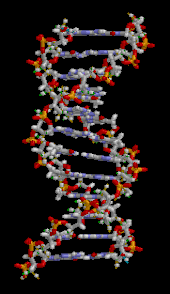
Das Genom einer Zelle liegt zumeist als DNA-Doppelstrang vor, bei dem die beiden basenpaarend einander komplementären Stränge räumlich die Form einer Doppelhelix bilden (siehe Abbildung). Bei der Replikation werden sie entwunden und getrennt jeweils durch Basenpaarung wieder komplementär ergänzt, sodass anschließend zwei (nahezu) identische doppelsträngige DNA-Moleküle vorliegen. Fehler beim Replikationsvorgang sind eine Quelle von Mutationen, die nach Kernteilung und Zellteilung in entstandenen Zellen als Veränderung genetischer Information vorliegen und weitergegeben werden können. ⓘ
Eigenschaften

Die DNA ist ein langes Polymer, das aus sich wiederholenden Einheiten, den so genannten Nukleotiden, besteht, die in der Regel durch einen einzigen Buchstaben symbolisiert werden: entweder A, T, C oder G. Die Struktur der DNA ist entlang ihrer Länge dynamisch und kann sich zu engen Schleifen und anderen Formen aufrollen. Bei allen Arten besteht sie aus zwei schraubenförmigen Ketten, die durch Wasserstoffbrücken miteinander verbunden sind. Beide Ketten sind um dieselbe Achse gewickelt und haben die gleiche Steigung von 34 ångströms (3,4 nm). Das Kettenpaar hat einen Radius von 10 Å (1,0 nm). Einer anderen Studie zufolge war die DNA-Kette in einer anderen Lösung 22-26 Å (2,2-2,6 nm) breit, und eine Nukleotideinheit war 3,3 Å (0,33 nm) lang. Obwohl jedes einzelne Nukleotid sehr klein ist, kann ein DNA-Polymer sehr lang sein und Hunderte von Millionen von Nukleotiden enthalten, wie z. B. im Chromosom 1. Chromosom 1 ist mit ca. 220 Millionen Basenpaaren das größte menschliche Chromosom und wäre, wenn es begradigt würde, 85 mm lang. ⓘ
Die DNA besteht normalerweise nicht aus einem einzigen Strang, sondern aus zwei Strängen, die fest miteinander verbunden sind. Diese beiden langen Stränge winden sich in Form einer Doppelhelix umeinander. Das Nukleotid enthält sowohl ein Segment des Molekülrückgrats (das die Kette zusammenhält) als auch eine Nukleobase (die mit dem anderen DNA-Strang in der Helix interagiert). Eine Nukleobase, die an einen Zucker gebunden ist, wird als Nukleosid bezeichnet, und eine Base, die an einen Zucker und an eine oder mehrere Phosphatgruppen gebunden ist, wird als Nukleotid bezeichnet. Ein Biopolymer, das aus mehreren verknüpften Nukleotiden besteht (wie in der DNA), wird als Polynukleotid bezeichnet. ⓘ
Das Grundgerüst des DNA-Strangs besteht aus abwechselnden Phosphat- und Zuckergruppen. Der Zucker in der DNA ist 2-Desoxyribose, ein Pentosezucker (mit fünf Kohlenstoffatomen). Die Zucker sind durch Phosphatgruppen verbunden, die Phosphodiesterbindungen zwischen dem dritten und fünften Kohlenstoffatom benachbarter Zuckerringe bilden. Diese werden als 3′-Ende (drei primäre Enden) und 5′-Ende (fünf primäre Enden) bezeichnet, wobei das Primzahlsymbol verwendet wird, um diese Kohlenstoffatome von denen der Base zu unterscheiden, an die die Desoxyribose eine glykosidische Bindung eingeht. Daher hat jeder DNA-Strang normalerweise ein Ende, an dem eine Phosphatgruppe an das 5′-Kohlenstoffatom einer Ribose gebunden ist (das 5′-Phosphoryl) und ein anderes Ende, an dem eine freie Hydroxylgruppe an das 3′-Kohlenstoffatom einer Ribose gebunden ist (das 3′-Hydroxyl). Die Ausrichtung der 3′- und 5′-Kohlenstoffe entlang des Zucker-Phosphat-Rückgrats verleiht jedem DNA-Strang eine bestimmte Richtung (manchmal auch Polarität genannt). In einer Nukleinsäure-Doppelhelix ist die Richtung der Nukleotide in einem Strang entgegengesetzt zu ihrer Richtung im anderen Strang: Die Stränge sind antiparallel. Die asymmetrischen Enden der DNA-Stränge haben eine Richtung von fünf Primzahlen (5′ ) und drei Primzahlen (3′), wobei das 5′-Ende eine endständige Phosphatgruppe und das 3′-Ende eine endständige Hydroxylgruppe aufweist. Ein wesentlicher Unterschied zwischen DNA und RNA ist der Zucker, wobei die 2-Desoxyribose in der DNA durch den verwandten Pentosezucker Ribose in der RNA ersetzt wird. ⓘ
Die DNA-Doppelhelix wird hauptsächlich durch zwei Kräfte stabilisiert: Wasserstoffbrücken zwischen den Nukleotiden und Basen-Stapel-Wechselwirkungen zwischen den aromatischen Nukleobasen. Die vier Basen der DNA sind Adenin (A), Cytosin (C), Guanin (G) und Thymin (T). Diese vier Basen sind an das Zuckerphosphat gebunden, um ein vollständiges Nukleotid zu bilden, wie das Adenosinmonophosphat zeigt. Adenin paart sich mit Thymin und Guanin mit Cytosin und bildet die Basenpaare A-T und G-C. ⓘ
Ein Palindrom ist eine Folge von Nukleotiden, bei denen sich die beiden komplementären Stränge jeweils von rechts genauso lesen lassen wie von links. ⓘ
Unter natürlichen Bedingungen (bei hoher Drehspannung der DNA) oder künstlich im Reagenzglas kann sich diese lineare Helix als Kreuzform (cruciform) herausbilden, indem zwei Zweige entstehen, die aus dem linearen Doppelstrang herausragen. Die Zweige stellen jeweils für sich eine Helix dar, allerdings bleiben am Ende eines Zweiges mindestens drei Nukleotide ungepaart. Beim Übergang von der Kreuzform in die lineare Helix bleibt die Basenpaarung wegen der Biegungsfähigkeit des Phosphodiester-Zucker-Rückgrates erhalten. ⓘ
Die spontane Zusammenlagerung von komplementären Basen zu sog. Stamm-Schleifen-Strukturen wird häufig auch bei Einzelstrang-DNA oder -RNA beobachtet. ⓘ
Die Phosphatreste sind aufgrund ihrer negativen Ladung hydrophil, sie geben DNA in wässriger Lösung insgesamt eine negative Ladung. Da diese negativ geladene, in Wasser gelöste DNA keine weiteren Protonen abgeben kann, handelt es sich streng genommen nicht (mehr) um eine Säure. Der Begriff Desoxyribonukleinsäure bezieht sich auf einen ungeladenen Zustand, in dem Protonen an die Phosphatreste angelagert sind. ⓘ
Klassifizierung der Nukleobasen
Die Nukleobasen werden in zwei Typen eingeteilt: die Purine, A und G, die verschmolzene fünf- und sechsgliedrige heterocyclische Verbindungen sind, und die Pyrimidine, die sechsgliedrigen Ringe C und T. Eine fünfte Pyrimidin-Nukleobase, Uracil (U), nimmt in der Regel den Platz von Thymin in der RNA ein und unterscheidet sich von Thymin durch das Fehlen einer Methylgruppe an seinem Ring. Neben RNA und DNA wurden viele künstliche Nukleinsäureanaloga geschaffen, um die Eigenschaften von Nukleinsäuren zu untersuchen oder um sie in der Biotechnologie zu verwenden. ⓘ
Nicht-kanonische Basen
In natürlicher DNA kommen auch modifizierte Basen vor. Insbesondere werden Methylierungen der kanonischen Basen im Rahmen der Epigenetik untersucht:
- Zunächst wurde im Jahr 1925 5-Methylcytosin (m5C) im Genom von Mycobacterium tuberculosis gefunden. Im Genom des Xanthomonas oryzae-Bakteriophagen Xp12 („Xanthomonas phage Xp12“), Gattung Pamexvirus, Morphotyp Siphoviren) und des Halovirus ΦH (Spezies Halobacterium-Virus phiH, wiss. Myohalovirus phiH, Familie Vertoviridae, Morphotyp Myoviren) ist das gesamte Cystosin-Kontingent durch 5-Methylcytosin ersetzt.
- Einen kompletten Ersatz von Cytosin durch 5-Glycosylhydroxymethylcytosin (syn. Glycosyl-5-hydroxymethylcytosin) in den Phagen T2, T4 und T6 der Spezies Escherichia-Virus T4 (Gattung Tequatrovirus, Unterfamilie Tevenvirinae, Morphotyp Myoviren) wurde 1953 beobachtet.
- Wie 1955 entdeckt wurde, ist N6-Methyladenin (6mA, m6A) in der DNA von Colibakterien vorhanden.
- N6-Carbamoylmethyladenin wurde 1975 in den Bakteriophagen Mu (ICTV: Spezies Escherichia virus Mu, früher Enterobacteria phage Mu (wiss. Muvirus mu, Morphotyp Myovirien) und Lambda-Mu beschrieben.
- 7-Methylguanin (m7G) wurde 1976 im Phagen DDVI (‚Enterobacteria phage DdVI‘ alias ‚DdV1‘, Gattung T4virus) von Shigella disenteriae beschrieben.
- N4-Methylcytosin (m4C) in DNA wurde 1983 beschrieben (in Bacillus centrosporus).
- 1985 wurde 5-Hydroxycytosin im Genom des Rhizobium-Phagen RL38JI gefunden.
- α-Putrescinylthymin (Alpha-Putrescinylthymin, putT) und α-Glutamylthymidin (Alpha-Glutamylthymidin) kommt im Genom sowohl des Delftia-Phagen ΦW-14 (Phi W-14, Spezies ‚Dellftia virus PhiW14‘, Gattung Ionavirus, Familie Myovrirdae) als auch des Bacillus-Phagen SP10 (Spezies „Bacillus phage SP-10“, Familie Herelleviridae, Morphotyp Siphoviren) vor.
- 5-Dihydroxypentyluracil wurde im Bacillus-Phagen SP15 (auch SP-15, Spezies Bacillus-Virus SP15, wiss. Thornevirus SP15, Morphotyp Myoviren) gefunden. a.) ⓘ
Die Funktion dieser nicht-kanonischen Basen in der DNA ist nicht bekannt. Sie wirken zumindest teilweise als molekulares Immunsystem und helfen, die Bakterien vor einer Infektion durch Viren zu schützen. a.) ⓘ
Nicht-Standard und modifizierte Basen bei Mikroben sind aber noch nicht alles:
- Es wurde auch über vier Modifikationen der Cytosinreste in humaner DNA berichtet. Diese Modifikationen bestehen aus dem Zusatz folgender Gruppen:
- Methyl (–CH3)
- Hydroxymethyl (–CH2OH)
- Formyl (–CHO)
- Carboxyl (–COOH)
Es wird angenommen, dass diese Modifikationen regulatorische Funktionen haben, Stichwort Epigenetik.
- Uracil ist in den Zentromer-Regionen von mindestens zwei menschlichen Chromosomen (6 und 11) zu finden. a.) ⓘ
Auflistung der nicht-kanonischen Basen in der DNA
Es ist bekannt, dass eine Reihe von nicht-kanonischen Basen in der DNA vorkommen. Die meisten von ihnen sind Modifikationen der kanonischen Basen plus Uracil. ⓘ
- Modifiziertes Adenosin
- N6-Carbamoyl-Methyladenin
- N6-Methyadenin
- Modifiziertes Guanin
- 7-Deazaguanin
- 7-Methylguanin
- Modifiziertes Cytosin
- N4-Methylcytosin
- 5-Carboxylcytosin
- 5-Formylcytosin
- 5-Glycosylhydroxymethylcytosin
- 5-Hydroxycytosin
- 5-Methylcytosin
- Modifiziertes Thymidin
- α-Glutamythymidin
- α-Putrescinylthymidin
- Uracil und Modifikationen
- Basis J
- Uracil
- 5-Dihydroxypentauracil
- 5-Hydroxymethyldeoxyuracil
- Andere
- Desoxyarchaeosin
- 2,6-Diaminopurin (2-Aminoadenin) ⓘ

Furchen
Doppelhelixstränge bilden das Rückgrat der DNA. Eine weitere Doppelhelix findet sich in den Zwischenräumen, den Furchen, zwischen den Strängen. Diese Hohlräume befinden sich neben den Basenpaaren und können eine Bindungsstelle darstellen. Da die Stränge nicht symmetrisch zueinander angeordnet sind, sind die Furchen ungleichmäßig groß. Die Hauptrille ist 22 ångströms (2,2 nm) breit, während die Nebenrille 12 Å (1,2 nm) breit ist. Aufgrund der größeren Breite der Hauptfurche sind die Kanten der Basen in der Hauptfurche besser zugänglich als in der Nebenfurche. Folglich kommen Proteine wie Transkriptionsfaktoren, die an bestimmte Sequenzen in der doppelsträngigen DNA binden können, in der Regel mit den Seiten der Basen in Kontakt, die in der Hauptrille exponiert sind. Diese Situation variiert bei ungewöhnlichen Konformationen der DNA innerhalb der Zelle (siehe unten), aber die Haupt- und Nebenrillen werden immer so benannt, dass sie die Unterschiede in der Breite widerspiegeln, die sich ergeben würden, wenn die DNA in die normale B-Form zurückgedreht würde. ⓘ
Basenpaarung
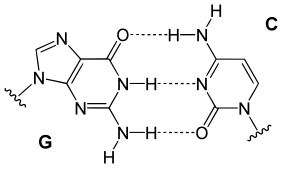
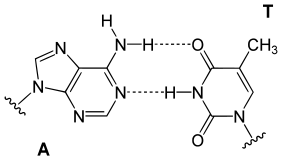
In einer DNA-Doppelhelix verbindet sich jede Nukleobase auf einem Strang mit nur einer Nukleobase auf dem anderen Strang. Dies wird als komplementäre Basenpaarung bezeichnet. Purine bilden Wasserstoffbrücken mit Pyrimidinen, wobei Adenin nur mit zwei Wasserstoffbrücken mit Thymin und Cytosin nur mit drei Wasserstoffbrücken mit Guanin verbunden ist. Diese Anordnung von zwei Nukleotiden, die sich über die Doppelhelix hinweg (von Sechs-Kohlenstoff-Ring zu Sechs-Kohlenstoff-Ring) verbinden, wird als Watson-Crick-Basenpaar bezeichnet. DNA mit hohem GC-Gehalt ist stabiler als DNA mit niedrigem GC-Gehalt. Ein Hoogsteen-Basenpaar (Wasserstoffbindungen zwischen dem 6-Kohlenstoff-Ring und dem 5-Kohlenstoff-Ring) ist eine seltene Variante der Basenpaarung. Da Wasserstoffbrücken nicht kovalent sind, können sie relativ leicht gebrochen und wieder verbunden werden. Die beiden DNA-Stränge einer Doppelhelix können also wie ein Reißverschluss auseinandergezogen werden, entweder durch mechanische Kraft oder durch hohe Temperatur. Dank dieser Komplementarität der Basenpaare werden alle Informationen in der doppelsträngigen Sequenz einer DNA-Helix auf jedem Strang dupliziert, was für die DNA-Replikation unerlässlich ist. Diese reversible und spezifische Interaktion zwischen komplementären Basenpaaren ist entscheidend für alle Funktionen der DNA in Organismen.
|
|
ssDNA vs. dsDNA
Wie bereits erwähnt, bestehen die meisten DNA-Moleküle aus zwei Polymersträngen, die durch nichtkovalente Bindungen schraubenförmig miteinander verbunden sind; diese doppelsträngige (dsDNA) Struktur wird hauptsächlich durch die Stapelung von Basen innerhalb des Strangs aufrechterhalten, die bei G,C-Stapeln am stärksten ist. Die beiden Stränge können sich voneinander lösen - ein Prozess, der als Schmelzen bekannt ist - und zwei einzelsträngige DNA-Moleküle (ssDNA) bilden. Das Schmelzen findet bei hoher Temperatur, niedrigem Salzgehalt und hohem pH-Wert statt (bei niedrigem pH-Wert schmilzt die DNA ebenfalls, aber da die DNA aufgrund der Säureabspaltung instabil ist, wird ein niedriger pH-Wert selten verwendet). ⓘ
Die Stabilität der dsDNA-Form hängt nicht nur vom GC-Gehalt (% G,C-Basenpaare) ab, sondern auch von der Sequenz (da die Stapelung sequenzspezifisch ist) und von der Länge (längere Moleküle sind stabiler). Die Stabilität kann auf verschiedene Weise gemessen werden; eine gängige Methode ist die Schmelztemperatur (auch Tm-Wert genannt), d. h. die Temperatur, bei der 50 % der Doppelstrangmoleküle in Einzelstrangmoleküle umgewandelt werden; die Schmelztemperatur ist von der Ionenstärke und der Konzentration der DNA abhängig. Die Schmelztemperatur hängt von der Ionenstärke und der DNA-Konzentration ab. Folglich bestimmen sowohl der prozentuale Anteil der GC-Basenpaare als auch die Gesamtlänge einer DNA-Doppelhelix die Stärke der Verbindung zwischen den beiden DNA-Strängen. Lange DNA-Helices mit einem hohen GC-Gehalt haben stärker interagierende Stränge, während kurze Helices mit einem hohen AT-Gehalt schwächer interagierende Stränge haben. In der Biologie neigen Teile der DNA-Doppelhelix, die sich leicht trennen lassen müssen, wie die TATAAT Pribnow-Box in einigen Promotoren, zu einem hohen AT-Gehalt, wodurch sich die Stränge leichter auseinanderziehen lassen. ⓘ
Im Labor kann die Stärke dieser Wechselwirkung gemessen werden, indem die Schmelztemperatur Tm ermittelt wird, die erforderlich ist, um die Hälfte der Wasserstoffbrücken zu brechen. Wenn alle Basenpaare in einer DNA-Doppelhelix schmelzen, trennen sich die Stränge und existieren in Lösung als zwei völlig unabhängige Moleküle. Diese einzelsträngigen DNA-Moleküle haben keine einheitliche Form, aber einige Konformationen sind stabiler als andere. ⓘ
Sense und Antisense
Eine DNA-Sequenz wird als "sense"-Sequenz bezeichnet, wenn sie mit der Kopie einer Boten-RNA übereinstimmt, die in ein Protein übersetzt wird. Die Sequenz auf dem gegenüberliegenden Strang wird als "Antisense"-Sequenz bezeichnet. Sowohl Sense- als auch Antisense-Sequenzen können auf verschiedenen Teilen desselben DNA-Strangs vorhanden sein (d. h. beide Stränge können sowohl Sense- als auch Antisense-Sequenzen enthalten). Sowohl in Prokaryonten als auch in Eukaryonten werden Antisense-RNA-Sequenzen produziert, aber die Funktionen dieser RNAs sind nicht ganz klar. Ein Vorschlag ist, dass Antisense-RNAs an der Regulierung der Genexpression durch RNA-RNA-Basenpaarung beteiligt sind. ⓘ
Einige wenige DNA-Sequenzen in Prokaryonten und Eukaryonten, und noch mehr in Plasmiden und Viren, verwischen die Unterscheidung zwischen Sinn- und Antisense-Strängen, indem sie überlappende Gene aufweisen. In diesen Fällen erfüllen einige DNA-Sequenzen eine Doppelfunktion, indem sie für ein Protein kodieren, wenn sie an einem Strang abgelesen werden, und für ein zweites Protein, wenn sie in der entgegengesetzten Richtung an dem anderen Strang abgelesen werden. Bei Bakterien kann diese Überlappung bei der Regulierung der Gentranskription eine Rolle spielen, während bei Viren überlappende Gene die Menge an Informationen erhöhen, die in dem kleinen viralen Genom kodiert werden können. ⓘ
Supercoiling
Die DNA kann wie ein Seil verdreht werden, was als DNA-Supercoiling bezeichnet wird. Im "entspannten" Zustand der DNA umkreist ein Strang normalerweise alle 10,4 Basenpaare die Achse der Doppelhelix, aber wenn die DNA verdreht wird, werden die Stränge enger oder lockerer gewickelt. Wenn die DNA in Richtung der Helix verdreht ist, handelt es sich um positive Supercoiling, und die Basen werden fester zusammengehalten. Wenn sie in die entgegengesetzte Richtung verdreht ist, handelt es sich um negative Supercoiling, und die Basen lösen sich leichter voneinander. In der Natur weist die meiste DNA eine leichte negative Supercoiling auf, die durch Enzyme namens Topoisomerasen erzeugt wird. Diese Enzyme werden auch benötigt, um die Verdrehungsspannungen abzubauen, die bei Prozessen wie der Transkription und der DNA-Replikation in die DNA-Stränge eingebracht werden. ⓘ

Alternative DNA-Strukturen
Die DNA existiert in vielen möglichen Konformationen, darunter A-DNA, B-DNA und Z-DNA, obwohl nur B-DNA und Z-DNA direkt in funktionellen Organismen beobachtet wurden. Welche Konformation die DNA annimmt, hängt vom Hydratationsgrad, der DNA-Sequenz, der Menge und Richtung der Supercoiling, den chemischen Modifikationen der Basen, der Art und Konzentration der Metallionen und dem Vorhandensein von Polyaminen in der Lösung ab. ⓘ
In den ersten veröffentlichten Berichten über Röntgenbeugungsmuster von A-DNA - und auch von B-DNA - wurden Analysen auf der Grundlage von Patterson-Funktionen verwendet, die nur eine begrenzte Menge an Strukturinformationen für orientierte DNA-Fasern lieferten. Eine alternative Analyse wurde 1953 von Wilkins et al. für die In-vivo-Röntgenbeugungsmuster von hoch hydratisierten DNA-Fasern in Form von Quadraten von Bessel-Funktionen vorgeschlagen. In derselben Zeitschrift präsentierten James Watson und Francis Crick ihre Molekularmodell-Analyse der DNA-Röntgenbeugungsmuster, die darauf hindeutete, dass die Struktur eine Doppelhelix war. ⓘ
Obwohl die B-DNA-Form unter den in Zellen herrschenden Bedingungen am häufigsten vorkommt, handelt es sich nicht um eine genau definierte Konformation, sondern um eine Familie verwandter DNA-Konformationen, die bei den in Zellen vorhandenen hohen Hydratationswerten auftreten. Die entsprechenden Röntgenbeugungs- und -streuungsmuster sind charakteristisch für molekulare Parakristalle mit einem hohen Grad an Unordnung. ⓘ
Im Vergleich zur B-DNA ist die A-DNA-Form eine breitere rechtshändige Spirale mit einer flachen, breiten Nebenrille und einer schmaleren, tieferen Hauptrille. Die A-Form kommt unter unphysiologischen Bedingungen in teilweise dehydrierten DNA-Proben vor, während sie in der Zelle in hybriden Paarungen von DNA- und RNA-Strängen und in Enzym-DNA-Komplexen entstehen kann. DNA-Abschnitte, deren Basen durch Methylierung chemisch verändert wurden, können eine größere Konformationsänderung erfahren und die Z-Form annehmen. Hier drehen sich die Stränge in einer linkshändigen Spirale um die Helixachse, im Gegensatz zur häufigeren B-Form. Diese ungewöhnlichen Strukturen können von spezifischen Z-DNA-bindenden Proteinen erkannt werden und sind möglicherweise an der Regulation der Transkription beteiligt. ⓘ
Alternative DNA-Chemie
Seit vielen Jahren schlagen Exobiologen die Existenz einer Schattenbiosphäre vor, einer postulierten mikrobiellen Biosphäre der Erde, in der völlig andere biochemische und molekulare Prozesse ablaufen als bei dem derzeit bekannten Leben. Einer der Vorschläge war die Existenz von Lebensformen, die Arsen anstelle von Phosphor in der DNA verwenden. Im Jahr 2010 wurde ein Bericht über diese Möglichkeit in dem Bakterium GFAJ-1 veröffentlicht, obwohl die Forschung umstritten war und es Hinweise darauf gibt, dass das Bakterium den Einbau von Arsen in das DNA-Rückgrat und andere Biomoleküle aktiv verhindert. ⓘ
Quadruplex-Strukturen
An den Enden der linearen Chromosomen befinden sich spezielle DNA-Regionen, die Telomere genannt werden. Die Hauptfunktion dieser Regionen besteht darin, der Zelle die Replikation der Chromosomenenden mithilfe des Enzyms Telomerase zu ermöglichen, da die Enzyme, die normalerweise die DNA replizieren, die extremen 3′-Enden der Chromosomen nicht kopieren können. Diese speziellen Chromosomenkappen tragen auch zum Schutz der DNA-Enden bei und verhindern, dass die DNA-Reparatursysteme in der Zelle sie als zu korrigierende Schäden behandeln. In menschlichen Zellen sind Telomere in der Regel Abschnitte einzelsträngiger DNA, die mehrere tausend Wiederholungen einer einfachen TTAGGG-Sequenz enthalten. ⓘ
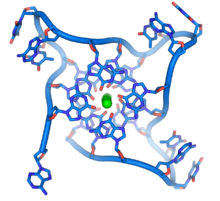
Diese guaninreichen Sequenzen können die Chromosomenenden stabilisieren, indem sie Strukturen aus gestapelten Sätzen von Vier-Basen-Einheiten bilden, anstatt der üblichen Basenpaare, die in anderen DNA-Molekülen zu finden sind. Hier bilden vier Guaninbasen, die als Guanin-Tetrade bezeichnet werden, eine flache Platte. Diese flachen Vierbaseneinheiten stapeln sich dann übereinander und bilden eine stabile G-Quadruplex-Struktur. Diese Strukturen werden durch Wasserstoffbrückenbindungen zwischen den Kanten der Basen und die Chelatbildung eines Metallions in der Mitte jeder Vierbaseneinheit stabilisiert. Es können auch andere Strukturen gebildet werden, bei denen der zentrale Satz von vier Basen entweder aus einem einzigen Strang stammt, der um die Basen gefaltet ist, oder aus mehreren verschiedenen parallelen Strängen, von denen jeder eine Base zur zentralen Struktur beiträgt. ⓘ
Zusätzlich zu diesen gestapelten Strukturen bilden Telomere auch große Schleifenstrukturen, die Telomerschleifen oder T-Schleifen genannt werden. Hier windet sich die einzelsträngige DNA in einem langen Kreis, der durch telomerbindende Proteine stabilisiert wird. Ganz am Ende der T-Schleife wird die einzelsträngige Telomer-DNA durch den Telomerstrang, der die doppelhelikale DNA unterbricht und eine Basenpaarung mit einem der beiden Stränge eingeht, an einer Region der doppelsträngigen DNA gehalten. Diese dreisträngige Struktur wird als Verdrängungsschleife oder D-Schleife bezeichnet. ⓘ

|
|
| Einzelne Verzweigung | Mehrere Verzweigungen |
Verzweigte DNA
Bei der DNA kommt es zum Ausfransen, wenn am Ende eines ansonsten komplementären DNA-Doppelstrangs nicht-komplementäre Bereiche vorhanden sind. Verzweigte DNA kann jedoch entstehen, wenn ein dritter DNA-Strang eingeführt wird, der angrenzende Bereiche enthält, die mit den ausgefransten Bereichen des bereits vorhandenen Doppelstrangs hybridisieren können. Obwohl das einfachste Beispiel für verzweigte DNA nur drei DNA-Stränge umfasst, sind auch Komplexe mit zusätzlichen Strängen und mehreren Verzweigungen möglich. Verzweigte DNA kann in der Nanotechnologie verwendet werden, um geometrische Formen zu konstruieren, siehe den Abschnitt über die Verwendung in der Technologie weiter unten. ⓘ
Künstliche Basen
Es wurden mehrere künstliche Nukleobasen synthetisiert und erfolgreich in das achtbasige DNA-Analogon namens Hachimoji-DNA eingebaut. Diese künstlichen Basen mit den Bezeichnungen S, B, P und Z sind in der Lage, sich auf vorhersehbare Weise miteinander zu verbinden (S-B und P-Z), die Doppelhelixstruktur der DNA zu erhalten und in RNA umgeschrieben zu werden. Ihre Existenz könnte als Hinweis darauf gewertet werden, dass die vier natürlichen Nukleobasen, die sich auf der Erde entwickelt haben, nichts Besonderes sind. Andererseits ist die DNA eng mit der RNA verbunden, die nicht nur als Abschrift der DNA fungiert, sondern auch als molekulare Maschine viele Aufgaben in den Zellen übernimmt. Zu diesem Zweck muss sie sich zu einer Struktur falten. Es hat sich gezeigt, dass zur Schaffung aller möglichen Strukturen mindestens vier Basen für die entsprechende RNA erforderlich sind, wobei auch eine höhere Anzahl möglich ist, was jedoch dem natürlichen Prinzip des geringsten Aufwands zuwiderlaufen würde. ⓘ
Säuregehalt
Die Phosphatgruppen der DNA verleihen ihr ähnliche saure Eigenschaften wie der Phosphorsäure und sie kann als starke Säure betrachtet werden. Bei einem normalen zellulären pH-Wert wird sie vollständig ionisiert, wobei Protonen freigesetzt werden, die negative Ladungen an den Phosphatgruppen zurücklassen. Diese negativen Ladungen schützen die DNA vor dem Abbau durch Hydrolyse, indem sie Nukleophile, die die DNA hydrolysieren könnten, abwehren. ⓘ
Makroskopisches Erscheinungsbild

Reine, aus Zellen extrahierte DNA bildet weiße, fadenförmige Klumpen. ⓘ
Chemische Modifikationen und veränderte DNA-Verpackung
|

|

|
| Cytosin | 5-Methylcytosin | Thymin |
Basenveränderungen und DNA-Verpackung
Die Expression von Genen wird durch die Art und Weise beeinflusst, wie die DNA in Chromosomen verpackt ist, in einer Struktur, die Chromatin genannt wird. Basenmodifikationen können bei der Verpackung eine Rolle spielen, wobei Regionen mit geringer oder keiner Genexpression in der Regel einen hohen Methylierungsgrad von Cytosinbasen aufweisen. Die DNA-Verpackung und ihr Einfluss auf die Genexpression kann auch durch kovalente Modifikationen des Histonproteinkerns erfolgen, um den die DNA in der Chromatinstruktur gewickelt ist, oder durch den Umbau durch Chromatin-Remodeling-Komplexe (siehe Chromatin-Remodeling). Darüber hinaus gibt es eine Wechselwirkung zwischen der DNA-Methylierung und der Histonmodifikation, so dass sie das Chromatin und die Genexpression koordiniert beeinflussen können. ⓘ
Ein Beispiel: Durch Cytosin-Methylierung entsteht 5-Methylcytosin, das für die X-Inaktivierung von Chromosomen wichtig ist. Der durchschnittliche Methylierungsgrad variiert von Organismus zu Organismus - dem Wurm Caenorhabditis elegans fehlt die Cytosin-Methylierung, während Wirbeltiere höhere Werte aufweisen, wobei bis zu 1 % ihrer DNA 5-Methylcytosin enthält. Trotz der Bedeutung von 5-Methylcytosin kann es deaminieren und eine Thyminbase hinterlassen, so dass methylierte Cytosine besonders anfällig für Mutationen sind. Weitere Basenmodifikationen sind die Methylierung von Adenin in Bakterien, das Vorhandensein von 5-Hydroxymethylcytosin im Gehirn und die Glykosylierung von Uracil zur Bildung der "J-Base" in Kinetoplastiden. ⓘ
Schädigung

Die DNA kann durch viele Arten von Mutagenen geschädigt werden, die die DNA-Sequenz verändern. Zu den Mutagenen gehören Oxidationsmittel, Alkylierungsmittel und auch hochenergetische elektromagnetische Strahlung wie ultraviolettes Licht und Röntgenstrahlen. Die Art der verursachten DNA-Schäden hängt von der Art des Mutagens ab. So kann UV-Licht die DNA durch die Bildung von Thymin-Dimeren, d. h. Querverbindungen zwischen Pyrimidinbasen, schädigen. Andererseits erzeugen Oxidationsmittel wie freie Radikale oder Wasserstoffperoxid mehrere Formen von Schäden, einschließlich Basenmodifikationen, insbesondere von Guanosin, und Doppelstrangbrüchen. Eine typische menschliche Zelle enthält etwa 150.000 Basen, die oxidativ geschädigt wurden. Von diesen oxidativen Läsionen sind Doppelstrangbrüche am gefährlichsten, da sie schwer zu reparieren sind und zu Punktmutationen, Insertionen, Deletionen in der DNA-Sequenz und chromosomalen Translokationen führen können. Diese Mutationen können Krebs verursachen. Aufgrund der inhärenten Grenzen der DNA-Reparaturmechanismen würden alle Menschen, wenn sie lange genug leben würden, irgendwann Krebs entwickeln. DNA-Schäden, die auf natürliche Weise durch normale zelluläre Prozesse entstehen, bei denen reaktive Sauerstoffspezies, die hydrolytischen Aktivitäten des Zellwassers usw. entstehen, treten ebenfalls häufig auf. Obwohl die meisten dieser Schäden repariert werden, können in jeder Zelle trotz der Reparaturprozesse einige DNA-Schäden zurückbleiben. Diese verbleibenden DNA-Schäden akkumulieren mit dem Alter in postmitotischen Geweben von Säugetieren. Diese Anhäufung scheint eine wichtige Ursache des Alterns zu sein. ⓘ
Viele Mutagene passen in den Raum zwischen zwei benachbarten Basenpaaren, was als Interkalation bezeichnet wird. Die meisten Interkalatoren sind aromatische und planare Moleküle; Beispiele sind Ethidiumbromid, Acridine, Daunomycin und Doxorubicin. Damit ein Interkalator zwischen die Basenpaare passt, müssen sich die Basen trennen, wodurch die DNA-Stränge durch Abwickeln der Doppelhelix verzerrt werden. Dies hemmt sowohl die Transkription als auch die DNA-Replikation und führt zu Toxizität und Mutationen. Infolgedessen können DNA-Interkalatoren krebserregend sein, und im Falle von Thalidomid auch teratogen. Andere wie Benzo[a]pyren-Diolepoxid und Aflatoxin bilden DNA-Addukte, die Fehler bei der Replikation hervorrufen. Aufgrund ihrer Fähigkeit, die DNA-Transkription und -Replikation zu hemmen, werden ähnliche Toxine auch in der Chemotherapie eingesetzt, um schnell wachsende Krebszellen zu hemmen. ⓘ
Biologische Funktionen

Bei Eukaryonten liegt die DNA in der Regel als lineare Chromosomen vor, bei Prokaryonten als zirkuläre Chromosomen. Der Chromosomensatz einer Zelle bildet ihr Genom; das menschliche Genom besteht aus etwa 3 Milliarden Basenpaaren DNA, die in 46 Chromosomen angeordnet sind. Die von der DNA getragene Information ist in einer Abfolge von DNA-Stücken, den Genen, enthalten. Die Übertragung der genetischen Information in den Genen erfolgt durch komplementäre Basenpaarung. Wenn eine Zelle beispielsweise bei der Transkription die Informationen eines Gens verwendet, wird die DNA-Sequenz durch die Anziehung zwischen der DNA und den richtigen RNA-Nukleotiden in eine komplementäre RNA-Sequenz kopiert. Normalerweise wird diese RNA-Kopie dann verwendet, um eine passende Proteinsequenz in einem Prozess namens Translation herzustellen, der von der gleichen Wechselwirkung zwischen RNA-Nukleotiden abhängt. Alternativ kann eine Zelle auch einfach ihre genetische Information in einem Prozess namens DNA-Replikation kopieren. Die Einzelheiten dieser Funktionen werden in anderen Artikeln behandelt; hier liegt der Schwerpunkt auf den Wechselwirkungen zwischen der DNA und anderen Molekülen, die die Funktion des Genoms vermitteln. ⓘ
Gene und Genome
Die genomische DNA ist in einem Prozess, der als DNA-Kondensation bezeichnet wird, eng und geordnet gepackt, um in das kleine verfügbare Volumen der Zelle zu passen. Bei Eukaryonten befindet sich die DNA im Zellkern und zu einem geringen Teil in den Mitochondrien und Chloroplasten. Bei Prokaryonten befindet sich die DNA in einem unregelmäßig geformten Körper im Zytoplasma, dem Nukleoid. Die genetische Information in einem Genom ist in den Genen enthalten, und der vollständige Satz dieser Information in einem Organismus wird als Genotyp bezeichnet. Ein Gen ist eine Einheit der Vererbung und ein Bereich der DNA, der ein bestimmtes Merkmal in einem Organismus beeinflusst. Gene enthalten einen offenen Leserahmen, der transkribiert werden kann, und regulatorische Sequenzen wie Promotoren und Enhancer, die die Transkription des offenen Leserahmens steuern. ⓘ
Bei vielen Arten kodiert nur ein kleiner Teil der Gesamtsequenz des Genoms für Proteine. So bestehen beispielsweise nur etwa 1,5 % des menschlichen Genoms aus proteinkodierenden Exons, während über 50 % der menschlichen DNA aus nicht-kodierenden, repetitiven Sequenzen bestehen. Die Gründe für das Vorhandensein von so viel nichtcodierender DNA in eukaryotischen Genomen und die außergewöhnlichen Unterschiede in der Genomgröße oder dem C-Wert zwischen den Arten sind ein seit langem bestehendes Rätsel, das als "C-Wert-Rätsel" bekannt ist. Einige DNA-Sequenzen, die keine Proteine kodieren, können jedoch funktionelle nicht-kodierende RNA-Moleküle kodieren, die an der Regulierung der Genexpression beteiligt sind. ⓘ
Einige nichtcodierende DNA-Sequenzen spielen strukturelle Rollen in Chromosomen. Telomere und Zentromere enthalten in der Regel nur wenige Gene, sind aber wichtig für die Funktion und Stabilität der Chromosomen. Eine häufige Form der nichtcodierenden DNA beim Menschen sind Pseudogene, d. h. Kopien von Genen, die durch Mutation deaktiviert wurden. Diese Sequenzen sind in der Regel nur molekulare Fossilien, obwohl sie gelegentlich als genetisches Rohmaterial für die Schaffung neuer Gene durch den Prozess der Genduplikation und Divergenz dienen können. ⓘ
Transkription und Translation
Ein Gen ist eine DNA-Sequenz, die genetische Informationen enthält und den Phänotyp eines Organismus beeinflussen kann. Innerhalb eines Gens definiert die Basenfolge entlang eines DNA-Strangs eine Boten-RNA-Sequenz, die wiederum eine oder mehrere Proteinsequenzen definiert. Die Beziehung zwischen den Nukleotidsequenzen der Gene und den Aminosäuresequenzen der Proteine wird durch die Regeln der Übersetzung bestimmt, die als genetischer Code bezeichnet werden. Der genetische Code besteht aus "Wörtern" mit drei Buchstaben, den Codons, die aus einer Sequenz von drei Nukleotiden gebildet werden (z. B. ACT, CAG, TTT). ⓘ
Bei der Transkription werden die Codons eines Gens von der RNA-Polymerase in Boten-RNA kopiert. Diese RNA-Kopie wird dann von einem Ribosom entschlüsselt, das die RNA-Sequenz durch Basenpaarung der Boten-RNA mit der Transfer-RNA liest, die Aminosäuren enthält. Da es 4 Basen in 3-Buchstaben-Kombinationen gibt, gibt es 64 mögliche Codons (43 Kombinationen). Diese kodieren die zwanzig Standard-Aminosäuren, wobei für die meisten Aminosäuren mehr als ein Codon möglich ist. Außerdem gibt es drei "Stopp"- oder "Nonsense"-Codons, die das Ende der kodierenden Region kennzeichnen: TAG, TAA und TGA (UAG, UAA und UGA auf der mRNA). ⓘ

Replikation
Die Zellteilung ist für das Wachstum eines Organismus unverzichtbar, aber wenn sich eine Zelle teilt, muss sie die DNA in ihrem Genom replizieren, damit die beiden Tochterzellen dieselbe genetische Information haben wie ihre Eltern. Die doppelsträngige Struktur der DNA bietet einen einfachen Mechanismus für die DNA-Replikation. Dabei werden die beiden Stränge getrennt, und anschließend wird die komplementäre DNA-Sequenz jedes Strangs durch ein Enzym namens DNA-Polymerase neu gebildet. Dieses Enzym stellt den komplementären Strang her, indem es durch komplementäre Basenpaarung die richtige Base findet und sie an den ursprünglichen Strang bindet. Da DNA-Polymerasen einen DNA-Strang nur in 5′- zu 3′-Richtung verlängern können, werden verschiedene Mechanismen verwendet, um die antiparallelen Stränge der Doppelhelix zu kopieren. Auf diese Weise bestimmt die Base des alten Strangs, welche Base auf dem neuen Strang erscheint, und die Zelle hat am Ende eine perfekte Kopie ihrer DNA. ⓘ
Extrazelluläre Nukleinsäuren
Nackte extrazelluläre DNA (eDNA), die größtenteils durch Zelltod freigesetzt wird, ist in der Umwelt nahezu allgegenwärtig. Ihre Konzentration im Boden kann bis zu 2 μg/L betragen, und ihre Konzentration in natürlichen Gewässern kann bis zu 88 μg/L betragen. Für eDNA wurden verschiedene mögliche Funktionen vorgeschlagen: Sie kann am horizontalen Gentransfer beteiligt sein, sie kann Nährstoffe liefern und als Puffer für die Aufnahme oder Titration von Ionen oder Antibiotika dienen. Extrazelluläre DNA wirkt als funktionelle extrazelluläre Matrixkomponente in den Biofilmen verschiedener Bakterienarten. Sie kann als Erkennungsfaktor fungieren, um die Anheftung und Ausbreitung bestimmter Zelltypen im Biofilm zu regulieren; sie kann zur Biofilmbildung beitragen; und sie kann zur physischen Stärke des Biofilms und zur Widerstandsfähigkeit gegen biologischen Stress beitragen. ⓘ
Zellfreie fötale DNA findet sich im Blut der Mutter und kann sequenziert werden, um viele Informationen über den sich entwickelnden Fötus zu ermitteln. ⓘ
Unter der Bezeichnung Umwelt-DNA wird eDNA in den Naturwissenschaften zunehmend als Erhebungsinstrument für die Ökologie eingesetzt, um die Bewegungen und das Vorhandensein von Arten im Wasser, in der Luft oder an Land zu überwachen und die biologische Vielfalt eines Gebiets zu bewerten. ⓘ
Neutrophile extrazelluläre Fallen
Neutrophile extrazelluläre Fallen (NETs) sind Netzwerke aus extrazellulären Fasern, die hauptsächlich aus DNA bestehen und es den Neutrophilen, einer Art weißer Blutkörperchen, ermöglichen, extrazelluläre Krankheitserreger abzutöten und dabei die Wirtszellen möglichst wenig zu schädigen. ⓘ
Wechselwirkungen mit Proteinen
Alle Funktionen der DNA hängen von Wechselwirkungen mit Proteinen ab. Diese Proteininteraktionen können unspezifisch sein, oder das Protein kann spezifisch an eine einzelne DNA-Sequenz binden. Auch Enzyme können an die DNA binden, und von diesen sind die Polymerasen, die die DNA-Basensequenz bei der Transkription und der DNA-Replikation kopieren, besonders wichtig. ⓘ
DNA-bindende Proteine

Strukturproteine, die an die DNA binden, sind wohlbekannte Beispiele für unspezifische DNA-Protein-Wechselwirkungen. In Chromosomen wird die DNA in Komplexen mit Strukturproteinen gehalten. Diese Proteine organisieren die DNA in einer kompakten Struktur, die Chromatin genannt wird. Bei Eukaryonten bindet die DNA an einen Komplex aus kleinen Basisproteinen, den Histonen, während bei Prokaryonten mehrere Arten von Proteinen beteiligt sind. Die Histone bilden einen scheibenförmigen Komplex, das so genannte Nukleosom, das zwei vollständige Windungen der doppelsträngigen DNA um seine Oberfläche gewickelt enthält. Diese unspezifischen Wechselwirkungen werden durch basische Reste in den Histonen gebildet, die ionische Bindungen mit dem sauren Zucker-Phosphat-Grundgerüst der DNA eingehen und somit weitgehend unabhängig von der Basensequenz sind. Zu den chemischen Veränderungen dieser basischen Aminosäurereste gehören Methylierung, Phosphorylierung und Acetylierung. Diese chemischen Veränderungen verändern die Stärke der Wechselwirkung zwischen der DNA und den Histonen, wodurch die DNA für die Transkriptionsfaktoren mehr oder weniger zugänglich wird und sich die Transkriptionsrate verändert. Zu den anderen unspezifischen DNA-bindenden Proteinen im Chromatin gehören die Proteine der Hochmobilitätsgruppe, die an gebogene oder verzerrte DNA binden. Diese Proteine sind wichtig für die Biegung von Nukleosomenanordnungen und deren Anordnung in den größeren Strukturen, die die Chromosomen bilden. ⓘ
Eine besondere Gruppe von DNA-bindenden Proteinen sind die DNA-bindenden Proteine, die spezifisch einzelsträngige DNA binden. Beim Menschen ist das Replikationsprotein A das am besten verstandene Mitglied dieser Familie und wird bei Prozessen eingesetzt, bei denen die Doppelhelix getrennt wird, einschließlich DNA-Replikation, Rekombination und DNA-Reparatur. Diese Bindungsproteine scheinen einzelsträngige DNA zu stabilisieren und sie vor der Bildung von Stammschleifen oder dem Abbau durch Nukleasen zu schützen. ⓘ

Im Gegensatz dazu haben sich andere Proteine so entwickelt, dass sie an bestimmte DNA-Sequenzen binden. Die am intensivsten untersuchten dieser Proteine sind die verschiedenen Transkriptionsfaktoren, d. h. Proteine, die die Transkription regulieren. Jeder Transkriptionsfaktor bindet an einen bestimmten Satz von DNA-Sequenzen und aktiviert oder hemmt die Transkription von Genen, die diese Sequenzen in der Nähe ihres Promotors haben. Die Transkriptionsfaktoren tun dies auf zwei Arten. Erstens können sie die für die Transkription verantwortliche RNA-Polymerase entweder direkt oder über andere Mediatorproteine binden; dadurch wird die Polymerase am Promotor lokalisiert und kann mit der Transkription beginnen. Alternativ dazu können Transkriptionsfaktoren Enzyme binden, die die Histone am Promotor verändern. Dadurch wird die Zugänglichkeit der DNA-Vorlage für die Polymerase verändert. ⓘ
Da diese DNA-Ziele im gesamten Genom eines Organismus vorkommen können, können sich Veränderungen in der Aktivität eines Typs von Transkriptionsfaktoren auf Tausende von Genen auswirken. Folglich sind diese Proteine häufig die Ziele der Signaltransduktionsprozesse, die die Reaktionen auf Umweltveränderungen oder die zelluläre Differenzierung und Entwicklung steuern. Die Spezifität der Interaktionen dieser Transkriptionsfaktoren mit der DNA beruht darauf, dass die Proteine mehrfache Kontakte zu den Kanten der DNA-Basen herstellen, wodurch sie die DNA-Sequenz "lesen" können. Die meisten dieser Baseninteraktionen finden in der Hauptfurche statt, wo die Basen am besten zugänglich sind. ⓘ
DNA-modifizierende Enzyme
Nukleasen und Ligasen
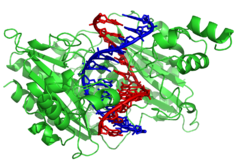
Nukleasen sind Enzyme, die DNA-Stränge schneiden, indem sie die Hydrolyse der Phosphodiesterbindungen katalysieren. Nukleasen, die Nukleotide an den Enden von DNA-Strängen hydrolysieren, werden als Exonukleasen bezeichnet, während Endonukleasen innerhalb von Strängen schneiden. Die in der Molekularbiologie am häufigsten verwendeten Nukleasen sind die Restriktionsendonukleasen, die die DNA an bestimmten Sequenzen schneiden. Das links abgebildete EcoRV-Enzym erkennt beispielsweise die 6-Basen-Sequenz 5′-GATATC-3′ und führt einen Schnitt an der horizontalen Linie aus. In der Natur schützen diese Enzyme Bakterien vor einer Phageninfektion, indem sie die Phagen-DNA verdauen, wenn sie in die Bakterienzelle eindringt, und so als Teil des Restriktionsmodifikationssystems wirken. In der Technik werden diese sequenzspezifischen Nukleasen beim molekularen Klonen und beim DNA-Fingerprinting eingesetzt. ⓘ
Als DNA-Ligasen bezeichnete Enzyme können abgeschnittene oder gebrochene DNA-Stränge wieder zusammenfügen. Ligasen sind besonders wichtig bei der Replikation von DNA-Strängen, da sie die kurzen DNA-Segmente, die an der Replikationsgabel entstehen, zu einer vollständigen Kopie der DNA-Vorlage zusammenfügen. Sie werden auch bei der DNA-Reparatur und der genetischen Rekombination eingesetzt. ⓘ
Topoisomerasen und Helikasen
Topoisomerasen sind Enzyme, die sowohl Nuklease- als auch Ligase-Aktivität besitzen. Diese Proteine verändern das Ausmaß der Supercoiling in der DNA. Einige dieser Enzyme schneiden die DNA-Helix und lassen einen Abschnitt rotieren, wodurch der Grad der Superwindung verringert wird; das Enzym verschließt dann den DNA-Bruch. Andere Arten dieser Enzyme sind in der Lage, eine DNA-Helix zu schneiden und dann einen zweiten DNA-Strang durch diese Bruchstelle zu führen, bevor sie die Helix wieder zusammenfügen. Topoisomerasen werden für viele DNA-Prozesse benötigt, z. B. für die DNA-Replikation und die Transkription. ⓘ
Helikasen sind Proteine, die eine Art molekularer Motor sind. Sie nutzen die chemische Energie der Nukleosidtriphosphate, vor allem des Adenosintriphosphats (ATP), um die Wasserstoffbrücken zwischen den Basen zu brechen und die DNA-Doppelhelix in Einzelstränge zu zerlegen. Diese Enzyme sind für die meisten Prozesse unerlässlich, bei denen Enzyme auf die DNA-Basen zugreifen müssen. ⓘ
Polymerasen
Polymerasen sind Enzyme, die Polynukleotidketten aus Nukleosidtriphosphaten synthetisieren. Die Sequenz ihrer Produkte wird auf der Grundlage bereits vorhandener Polynukleotidketten, den so genannten Templates, erstellt. Diese Enzyme funktionieren, indem sie wiederholt ein Nukleotid an die 3′-Hydroxylgruppe am Ende der wachsenden Polynukleotidkette anhängen. Folglich arbeiten alle Polymerasen in einer 5′-zu-3′-Richtung. Im aktiven Zentrum dieser Enzyme bildet das eintreffende Nukleosidtriphosphat eine Basenpaarung mit der Vorlage: So können die Polymerasen den komplementären Strang ihrer Vorlage genau synthetisieren. Polymerasen werden nach der Art der Vorlage, die sie verwenden, klassifiziert. ⓘ
Bei der DNA-Replikation erstellen DNA-abhängige DNA-Polymerasen Kopien von DNA-Polynukleotidketten. Um die biologische Information zu erhalten, ist es wichtig, dass die Basenabfolge in jeder Kopie genau komplementär zur Basenabfolge im Vorlagenstrang ist. Viele DNA-Polymerasen haben eine Korrekturlesefunktion. Dabei erkennt die Polymerase die gelegentlichen Fehler in der Synthesereaktion an der fehlenden Basenpaarung zwischen den nicht übereinstimmenden Nukleotiden. Wenn eine Fehlpaarung erkannt wird, wird eine 3′- bis 5′-Exonukleaseaktivität aktiviert und die falsche Base entfernt. In den meisten Organismen arbeiten die DNA-Polymerasen in einem großen Komplex, dem so genannten Replisom, das mehrere zusätzliche Untereinheiten wie die DNA-Klammer oder Helikasen enthält. ⓘ
RNA-abhängige DNA-Polymerasen sind eine spezielle Klasse von Polymerasen, die die Sequenz eines RNA-Strangs in die DNA kopieren. Dazu gehören die reverse Transkriptase, ein virales Enzym, das an der Infektion von Zellen durch Retroviren beteiligt ist, und die Telomerase, die für die Replikation von Telomeren erforderlich ist. So ist beispielsweise die reverse Transkriptase von HIV ein Enzym für die Replikation des AIDS-Virus. Telomerase ist eine ungewöhnliche Polymerase, da sie ihre eigene RNA-Vorlage als Teil ihrer Struktur enthält. Sie synthetisiert Telomere an den Enden der Chromosomen. Telomere verhindern die Verschmelzung der Enden benachbarter Chromosomen und schützen die Chromosomenenden vor Schäden. ⓘ
Die Transkription wird von einer DNA-abhängigen RNA-Polymerase durchgeführt, die die Sequenz eines DNA-Strangs in RNA kopiert. Um mit der Transkription eines Gens zu beginnen, bindet die RNA-Polymerase an eine DNA-Sequenz, die als Promotor bezeichnet wird, und trennt die DNA-Stränge. Anschließend kopiert sie die Gensequenz in ein Boten-RNA-Transkript, bis sie eine DNA-Region erreicht, die als Terminator bezeichnet wird, wo sie anhält und sich von der DNA ablöst. Wie die menschlichen DNA-abhängigen DNA-Polymerasen arbeitet auch die RNA-Polymerase II, das Enzym, das die meisten Gene im menschlichen Genom transkribiert, als Teil eines großen Proteinkomplexes mit mehreren regulatorischen und akzessorischen Untereinheiten. ⓘ
Genetische Rekombination

|

|

Eine DNA-Helix interagiert normalerweise nicht mit anderen DNA-Segmenten, und in menschlichen Zellen belegen die verschiedenen Chromosomen sogar getrennte Bereiche im Zellkern, die als "Chromosomengebiete" bezeichnet werden. Diese physische Trennung der verschiedenen Chromosomen ist wichtig für die Fähigkeit der DNA, als stabiler Informationsspeicher zu fungieren, denn eine der wenigen Gelegenheiten, bei denen Chromosomen miteinander interagieren, ist die Chromosomenkreuzung, die während der sexuellen Fortpflanzung stattfindet, wenn eine genetische Rekombination erfolgt. Bei der Chromosomenkreuzung trennen sich zwei DNA-Helices, tauschen einen Abschnitt aus und fügen sich dann wieder zusammen. ⓘ
Die Rekombination ermöglicht es den Chromosomen, genetische Informationen auszutauschen und neue Genkombinationen zu bilden, was die Effizienz der natürlichen Selektion erhöht und für die rasche Entwicklung neuer Proteine wichtig sein kann. Genetische Rekombination kann auch bei der DNA-Reparatur eine Rolle spielen, insbesondere bei der Reaktion der Zelle auf Doppelstrangbrüche. ⓘ
Die häufigste Form des chromosomalen Crossover ist die homologe Rekombination, bei der die beiden beteiligten Chromosomen sehr ähnliche Sequenzen aufweisen. Die nicht-homologe Rekombination kann für Zellen schädlich sein, da sie zu Chromosomentranslokationen und genetischen Anomalien führen kann. Die Rekombinationsreaktion wird durch Enzyme katalysiert, die als Rekombinasen bekannt sind, wie z. B. RAD51. Der erste Schritt bei der Rekombination ist ein doppelsträngiger Bruch, der entweder durch eine Endonuklease oder eine Beschädigung der DNA verursacht wird. Eine Reihe von Schritten, die zum Teil von der Rekombinase katalysiert werden, führt dann zur Verbindung der beiden Helices durch mindestens eine Holliday-Kreuzung, bei der ein Segment eines Einzelstrangs in jeder Helix an den komplementären Strang in der anderen Helix angeheftet wird. Bei der Holliday-Kreuzung handelt es sich um eine tetraedrische Kreuzungsstruktur, die entlang des Chromosomenpaars bewegt werden kann, wobei ein Strang gegen einen anderen ausgetauscht wird. Die Rekombinationsreaktion wird dann durch Abspaltung der Kreuzung und erneute Ligation der freigesetzten DNA gestoppt. Bei der Rekombination tauschen nur Stränge mit gleicher Polarität die DNA aus. Es gibt zwei Arten der Spaltung: Ost-West-Spaltung und Nord-Süd-Spaltung. Bei der Nord-Süd-Spaltung werden beide DNA-Stränge durchtrennt, während bei der Ost-West-Spaltung ein DNA-Strang intakt bleibt. Die Bildung einer Holliday-Kreuzung während der Rekombination ermöglicht die genetische Vielfalt, den Austausch von Genen auf Chromosomen und die Expression von Wildtyp-Virusgenomen. ⓘ
Entwicklung
Die DNA enthält die genetische Information, die es allen Lebensformen ermöglicht, zu funktionieren, zu wachsen und sich zu vermehren. Es ist jedoch unklar, wie lange in der 4 Milliarden Jahre währenden Geschichte des Lebens die DNA diese Funktion erfüllt hat, denn es wurde vorgeschlagen, dass die frühesten Lebensformen RNA als genetisches Material verwendet haben könnten. RNA könnte als zentraler Bestandteil des frühen Zellstoffwechsels fungiert haben, da sie sowohl genetische Informationen übertragen als auch als Teil von Ribozymen Katalysen durchführen kann. Diese alte RNA-Welt, in der die Nukleinsäure sowohl für die Katalyse als auch für die Genetik verwendet wurde, könnte die Entwicklung des heutigen genetischen Codes auf der Grundlage von vier Nukleotidbasen beeinflusst haben. Dies wäre der Fall, da die Anzahl der verschiedenen Basen in einem solchen Organismus einen Kompromiss darstellt zwischen einer geringen Anzahl von Basen, die die Replikationsgenauigkeit erhöht, und einer großen Anzahl von Basen, die die katalytische Effizienz von Ribozymen erhöht. Es gibt jedoch keine direkten Beweise für alte genetische Systeme, da die Wiederherstellung von DNA aus den meisten Fossilien unmöglich ist, da DNA in der Umwelt weniger als eine Million Jahre überlebt und in Lösung langsam in kurze Fragmente zerfällt. Es wurden Behauptungen über ältere DNA aufgestellt, vor allem ein Bericht über die Isolierung eines lebensfähigen Bakteriums aus einem 250 Millionen Jahre alten Salzkristall, aber diese Behauptungen sind umstritten. ⓘ
Die Bausteine der DNA (Adenin, Guanin und verwandte organische Moleküle) könnten im Weltraum entstanden sein. Komplexe organische DNA- und RNA-Verbindungen des Lebens, einschließlich Uracil, Cytosin und Thymin, wurden auch im Labor unter Bedingungen gebildet, die denen im Weltraum ähneln, wobei Ausgangschemikalien wie Pyrimidin verwendet wurden, die in Meteoriten gefunden wurden. Pyrimidin könnte wie die polyzyklischen aromatischen Kohlenwasserstoffe (PAK), die kohlenstoffreichste Chemikalie im Universum, in Roten Riesen oder in interstellaren kosmischen Staub- und Gaswolken gebildet worden sein. ⓘ
Im Februar 2021 meldeten Wissenschaftler zum ersten Mal die Sequenzierung von DNA aus tierischen Überresten, in diesem Fall von einem über eine Million Jahre alten Mammut, der ältesten bisher sequenzierten DNA. ⓘ
Anwendungen in der Technologie
Gentechnologie
Es wurden Methoden entwickelt, um die DNA aus Organismen zu reinigen, wie z. B. die Phenol-Chloroform-Extraktion, und um sie im Labor zu manipulieren, wie z. B. Restriktionsverdauungen und die Polymerase-Kettenreaktion. In der modernen Biologie und Biochemie werden diese Techniken in der rekombinanten DNA-Technologie intensiv genutzt. Rekombinante DNA ist eine künstlich hergestellte DNA-Sequenz, die aus anderen DNA-Sequenzen zusammengesetzt wurde. Sie kann in Form von Plasmiden oder im entsprechenden Format mit Hilfe eines viralen Vektors in Organismen übertragen werden. Die erzeugten gentechnisch veränderten Organismen können zur Herstellung von Produkten wie rekombinanten Proteinen verwendet werden, in der medizinischen Forschung eingesetzt oder in der Landwirtschaft angebaut werden. ⓘ
DNA-Profilierung
Kriminaltechniker können anhand der DNA in Blut, Sperma, Haut, Speichel oder Haaren, die an einem Tatort gefunden wurden, eine übereinstimmende DNA einer Person, z. B. eines Täters, identifizieren. Dieses Verfahren wird formal als DNA-Profiling bezeichnet, auch DNA-Fingerprinting genannt. Bei der Erstellung von DNA-Profilen werden die Längen variabler Abschnitte repetitiver DNA, wie z. B. kurze Tandemwiederholungen und Minisatelliten, zwischen Personen verglichen. Diese Methode ist in der Regel eine äußerst zuverlässige Technik zur Identifizierung einer übereinstimmenden DNA. Die Identifizierung kann jedoch kompliziert sein, wenn der Tatort mit DNA von mehreren Personen kontaminiert ist. Das DNA-Profiling wurde 1984 von dem britischen Genetiker Sir Alec Jeffreys entwickelt und erstmals in der forensischen Wissenschaft eingesetzt, um Colin Pitchfork im Fall der Enderby-Morde 1988 zu überführen. ⓘ
Die Entwicklung der forensischen Wissenschaft und die Möglichkeit, genetische Übereinstimmungen mit winzigen Blut-, Haut-, Speichel- oder Haarproben zu erhalten, hat dazu geführt, dass viele Fälle neu untersucht werden. Es können nun Beweise aufgedeckt werden, die zum Zeitpunkt der ursprünglichen Untersuchung wissenschaftlich unmöglich waren. In Verbindung mit der Aufhebung des Gesetzes über die doppelte Strafverfolgung an einigen Orten kann dies die Wiederaufnahme von Fällen ermöglichen, in denen frühere Verfahren nicht genügend Beweise erbracht haben, um die Geschworenen zu überzeugen. Personen, die schwerer Verbrechen angeklagt sind, können aufgefordert werden, eine DNA-Probe zu Vergleichszwecken abzugeben. Die naheliegendste Verteidigung gegen forensisch gewonnene DNA-Abgleiche ist die Behauptung, dass es zu einer Kreuzkontamination von Beweismitteln gekommen ist. Dies hat dazu geführt, dass bei neuen Fällen von Schwerverbrechen peinlich genau vorgegangen wird. ⓘ
DNA-Profiling wird auch erfolgreich eingesetzt, um Opfer von Massenunfällen, Leichen oder Leichenteile bei schweren Unfällen und einzelne Opfer in Massengräbern durch den Abgleich mit Familienangehörigen eindeutig zu identifizieren. ⓘ
DNA-Profiling wird auch bei DNA-Vaterschaftstests eingesetzt, um festzustellen, ob jemand der biologische Eltern- oder Großelternteil eines Kindes ist, wobei die Wahrscheinlichkeit der Abstammung in der Regel 99,99 % beträgt, wenn der angebliche Elternteil biologisch mit dem Kind verwandt ist. Normale DNA-Sequenzierungsmethoden werden nach der Geburt durchgeführt, aber es gibt neue Methoden, um die Vaterschaft zu testen, während eine Mutter noch schwanger ist. ⓘ
DNA-Enzyme oder katalytische DNA
Desoxyribozyme, auch als DNA-Enzyme oder katalytische DNA bezeichnet, wurden erstmals 1994 entdeckt. Es handelt sich dabei meist um einzelsträngige DNA-Sequenzen, die aus einem großen Pool zufälliger DNA-Sequenzen durch einen kombinatorischen Ansatz isoliert wurden, der als In-vitro-Auswahl oder systematische Evolution von Liganden durch exponentielle Anreicherung (SELEX) bezeichnet wird. DNA-Zyme katalysieren eine Vielzahl chemischer Reaktionen wie RNA-DNA-Spaltung, RNA-DNA-Ligation, Phosphorylierung von Aminosäuren, Bildung von Kohlenstoff-Kohlenstoff-Bindungen usw. DNA-Zyme können die katalytische Rate chemischer Reaktionen um das bis zu 100.000.000.000-fache gegenüber der unkatalysierten Reaktion erhöhen. Die am intensivsten untersuchte Klasse von DNA-Zymen sind RNA-spaltende Typen, die zum Nachweis verschiedener Metallionen und zur Entwicklung therapeutischer Wirkstoffe eingesetzt werden. Es wurde über mehrere metall-spezifische DNAzyme berichtet, darunter das GR-5-DNAzym (bleispezifisch), die CA1-3-DNAzyme (kupferspezifisch), das 39E-DNAzym (uranylspezifisch) und das NaA43-DNAzym (natriumspezifisch). Das NaA43-DNAzym, das Berichten zufolge mehr als 10.000-fach selektiv für Natrium gegenüber anderen Metallionen ist, wurde zur Herstellung eines Echtzeit-Natriumsensors in Zellen verwendet. ⓘ
Bioinformatik
Die Bioinformatik umfasst die Entwicklung von Techniken zur Speicherung, Auswertung, Suche und Bearbeitung biologischer Daten, einschließlich DNA-Nukleinsäuresequenzdaten. Dies hat zu weitreichenden Fortschritten in der Informatik geführt, insbesondere bei Algorithmen zur Stringsuche, beim maschinellen Lernen und in der Datenbanktheorie. String-Such- oder Matching-Algorithmen, die das Vorkommen einer Buchstabenfolge innerhalb einer größeren Buchstabenfolge finden, wurden entwickelt, um nach bestimmten Nukleotidsequenzen zu suchen. Die DNA-Sequenz kann mit anderen DNA-Sequenzen abgeglichen werden, um homologe Sequenzen zu identifizieren und die spezifischen Mutationen zu finden, durch die sie sich unterscheiden. Diese Techniken, insbesondere das Multiple Sequence Alignment, werden bei der Untersuchung phylogenetischer Beziehungen und der Funktion von Proteinen eingesetzt. Datensätze, die DNA-Sequenzen ganzer Genome enthalten, wie z. B. die des Humangenomprojekts, sind ohne die Anmerkungen, die die Position von Genen und regulatorischen Elementen auf jedem Chromosom angeben, schwer zu verwenden. Bereiche der DNA-Sequenz, die charakteristische Muster aufweisen, die mit Protein- oder RNA-kodierenden Genen assoziiert werden, können durch Genfindungsalgorithmen identifiziert werden, die es Forschern ermöglichen, das Vorhandensein bestimmter Genprodukte und ihre möglichen Funktionen in einem Organismus vorherzusagen, noch bevor sie experimentell isoliert wurden. Es können auch ganze Genome verglichen werden, was Aufschluss über die Evolutionsgeschichte eines bestimmten Organismus geben kann und die Untersuchung komplexer evolutionärer Ereignisse ermöglicht. ⓘ
DNA-Nanotechnologie

Die DNA-Nanotechnologie nutzt die einzigartigen molekularen Erkennungseigenschaften der DNA und anderer Nukleinsäuren, um selbstorganisierende verzweigte DNA-Komplexe mit nützlichen Eigenschaften zu schaffen. Die DNA wird also als Strukturmaterial und nicht als Träger biologischer Informationen verwendet. Dies hat zur Schaffung von zweidimensionalen periodischen Gittern (sowohl auf der Grundlage von Kacheln als auch mit der DNA-Origami-Methode) und dreidimensionalen Strukturen in Form von Polyedern geführt. Es wurden auch nanomechanische Geräte und algorithmische Selbstorganisation demonstriert, und diese DNA-Strukturen wurden als Vorlage für die Anordnung anderer Moleküle wie Goldnanopartikel und Streptavidin-Proteine verwendet. DNA und andere Nukleinsäuren bilden die Grundlage für Aptamere, synthetische Oligonukleotid-Liganden für spezifische Zielmoleküle, die in einer Reihe von biotechnologischen und biomedizinischen Anwendungen eingesetzt werden. ⓘ
Geschichte und Anthropologie
Da die DNA im Laufe der Zeit Mutationen sammelt, die dann vererbt werden, enthält sie historische Informationen, und durch den Vergleich von DNA-Sequenzen können Genetiker auf die Evolutionsgeschichte von Organismen, ihre Phylogenie, schließen. Dieser Bereich der Phylogenetik ist ein leistungsfähiges Instrument der Evolutionsbiologie. Wenn DNA-Sequenzen innerhalb einer Art verglichen werden, können Populationsgenetiker die Geschichte bestimmter Populationen in Erfahrung bringen. Dies kann in Studien von der ökologischen Genetik bis zur Anthropologie genutzt werden. ⓘ
Speicherung von Informationen
Die DNA als Informationsspeicher hat ein enormes Potenzial, da sie im Vergleich zu elektronischen Geräten eine viel höhere Speicherdichte aufweist. Die hohen Kosten, die langsamen Lese- und Schreibzeiten (Speicherlatenz) und die unzureichende Zuverlässigkeit haben jedoch ihren praktischen Einsatz verhindert. ⓘ
Entdeckungsgeschichte



1869 entdeckte der Schweizer Arzt Friedrich Miescher in einem Extrakt aus Eiter eine durch milde Säurebehandlung aus den Zellkernen der Leukozyten gewonnene Substanz, die er Nuklein nannte. Miescher arbeitete damals im Labor von Felix Hoppe-Seyler im Tübinger Schloss. 1892 (bzw. 1897 posthum, nachdem der zu Grunde liegende Brief veröffentlicht wurde) führte der „späte“ Miescher auf Basis seiner biochemischen Erkenntnisse hinsichtlich der Komplexität von Nukleinen und Proteinen als erster den Schrift- oder Code-Vergleich für den noch zu entdeckenden Träger der Erbinformation als Forschungshypothese in die Genetik ein. 1889 isoliert der Deutsche Richard Altmann aus dem Nuklein Proteine und die Nukleinsäure. Weitere Erkenntnisse zur Nukleinsäure gehen auf die Arbeiten von Albrecht Kossel (siehe „Die Entdeckung der Nukleinbasen“) zurück, für die er 1910 mit dem Nobelpreis für Physiologie oder Medizin ausgezeichnet wurde. Im Jahr 1885 teilte er mit, dass aus einer größeren Menge Rinder-Bauchspeicheldrüse eine stickstoffreiche Base mit der Summenformel C5H5N5 isoliert wurde, für die er, abgeleitet von dem griechischen Wort „aden“ für Drüse, den Namen Adenin vorschlug. 1891 konnte Kossel (nach Altmanns Verfahren) Hefe-Nukleinsäure herstellen und Adenin und Guanin als Spaltprodukte nachweisen. Es stellte sich heraus, dass auch ein Kohlenhydrat Bestandteil der Nukleinsäure sein musste. Kossel wählte für die basischen Substanzen Guanin und Adenin sowie seine Derivate den Namen Nucleinbasen. ⓘ
1893 berichtete Kossel, dass er aus den Thymusdrüsen des Kalbes Nukleinsäure gewonnen und ein gut kristallisiertes Spaltprodukt erhalten hatte, für das er den Namen Thymin vorschlug. 1894 isolierte er aus den Thymusdrüsen eine weitere (basische) Substanz. Kossel gab ihr den Namen Cytosin. ⓘ
Nachdem am Ende des 19. Jahrhunderts – im Wesentlichen durch die Synthesen Emil Fischers – die Strukturformeln des Guanins und Adenins als Purinkörper und des Thymins als Pyrimidinkörper endgültig aufgeklärt worden waren, konnte Kossel mit Hermann Steudel (1871–1967) auch die Strukturformel der Nukleinbase Cytosin als Pyrimidinkörper zweifelsfrei ermitteln. Es hatte sich inzwischen erwiesen, dass Guanin, Adenin sowie Thymin und Cytosin in allen entwicklungsfähigen Zellen zu finden sind. ⓘ
Die Erkenntnisse über diese vier Nukleinbasen sollten für die spätere Strukturaufklärung der DNA von wesentlicher Bedeutung sein. Es war Albrecht Kossel, der sie – zusammen mit einem Kohlenhydrat und der Phosphorsäure – eindeutig als Bausteine der Nukleinsäure charakterisierte: „Es gelang mir, eine Reihe von Bruchstücken zu erhalten … welche durch eine ganz eigentümliche Ansammlung von Stickstoffatomen gekennzeichnet sind. Es sind hier nebeneinander … das Cytosin, das Thymin, das Adenin und das Guanin.“ (Nobelvortrag am 12. Dezember 1910). ⓘ
Der aus Litauen stammende Biochemiker Phoebus Levene schlug eine kettenartige Struktur der Nukleinsäure vor, in welcher die Nukleotide durch die Phosphatreste zusammengefügt sind und sich wiederholen. 1929 konnte er in Zusammenarbeit mit dem russischen Physiologen Efim London (1869–1932) den Zuckeranteil der „tierische Nukleinsäure“ als Desoxyribose identifizieren (J. Biol. Chem.1929, 83. Seiten 793-802). [5a] Erst nachfolgend wurde sie als Desoxyribonukleinsäure bezeichnet. Es wurde erkannt, dass sie auch in pflanzlichen Zellkernen vorkommt. ⓘ
Als wirksamer Bestandteil der Chromosomen bzw. des Kernchromatins wurde die DNA bereits 1932 von K. Voit und Hartwig Kuhlenbeck angesehen. 1937 publizierte William Astbury erstmals Röntgenbeugungsmuster, die auf eine repetitive Struktur der DNA hinwiesen. ⓘ
1943 wies Oswald Avery nach, dass die Transformation von Bakterien, das heißt die Weitergabe erblicher Information von einem Bakterien-Stamm auf einen anderen (heute horizontaler Gentransfer genannt), auf der Übertragung von DNA beruht. Dies widersprach der damals noch allgemein favorisierten Annahme, dass nicht die DNA, sondern Proteine die Träger der Erbinformation seien. Unterstützung in seiner Interpretation erhielt Avery 1952, als Alfred Hershey und Martha Chase nachwiesen, dass DNA die Erbinformation des T2-Phagen enthält. ⓘ
Den strukturellen Aufbau der DNA zu entschlüsseln und im Modell nachzubilden gelang dem US-Amerikaner James Watson und dem Briten Francis Crick am 28. Februar 1953. Ihre Entdeckung publizierten sie in der April-Ausgabe 1953 des Magazins Nature in ihrem berühmten Artikel Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid. Watson kam 1951 nach England, nachdem er ein Jahr zuvor an der Indiana University Bloomington in den USA promoviert hatte. Er hatte zwar ein Stipendium für Molekularbiologie bekommen, beschäftigte sich aber vermehrt mit der Frage des menschlichen Erbguts. Crick widmete sich in Cambridge gerade erfolglos seiner Promotion über die Kristallstruktur des Hämoglobinmoleküls, als er 1951 Watson traf. ⓘ
Zu dieser Zeit war bereits ein erbitterter Wettlauf um die Struktur der DNA entbrannt, an dem sich neben anderen auch Linus Pauling am California Institute of Technology (Caltech) beteiligte. Watson und Crick waren eigentlich anderen Projekten zugeteilt worden und besaßen kein bedeutendes Fachwissen in Chemie. Sie bauten ihre Überlegungen auf den Forschungsergebnissen der anderen Wissenschaftler auf. ⓘ
Watson sagte, er wolle das Erbgut entschlüsseln, ohne Chemie lernen zu müssen. In einem Gespräch mit dem renommierten Chemiker und Ersteller der Chargaff-Regeln, Erwin Chargaff, vergaß Crick wichtige Molekülstrukturen, und Watson machte im selben Gespräch unpassende Anmerkungen, die seine Unkenntnis auf dem Gebiet der Chemie verrieten. Chargaff nannte die jungen Kollegen im Anschluss „wissenschaftliche Clowns“. ⓘ
Watson besuchte Ende 1952 am King’s College in London Maurice Wilkins, der ihm DNA-Röntgenaufnahmen von Rosalind Franklin zeigte (was gegen den Willen von Franklin geschah). Watson sah sofort, dass es sich bei dem Molekül um eine Doppel-Helix handeln musste; Franklin selbst hatte aufgrund der Daten auch das Vorhandensein einer Helix vermutet, jedoch hatte sie kein überzeugendes Modell für die Struktur vorzuweisen. Da bekannt war, dass die Purin- und Pyrimidin-Basen Paare bilden, gelang es Watson und Crick, die ganze Molekularstruktur herzuleiten. So entwickelten sie am Cavendish-Laboratorium der Universität Cambridge das Doppelhelix-Modell der DNA mit den Basenpaaren in der Mitte, das am 25. April 1953 in der Zeitschrift Nature publiziert wurde. ⓘ
Diese denkwürdige Veröffentlichung enthält gegen Ende den Satz „It has not escaped our notice that the specific pairing we have postulated immediately suggests a possible copying mechanism for the genetic material“. („Es ist unserer Aufmerksamkeit nicht entgangen, dass die spezifische Paarung, die wir als gegeben voraussetzen, unmittelbar auf einen möglichen Vervielfältigungsmechanismus für das genetische Material schließen lässt.“) ⓘ
„Für ihre Entdeckungen über die Molekularstruktur der Nukleinsäuren und ihre Bedeutung für die Informationsübertragung in lebender Substanz“ erhielten Watson und Crick zusammen mit Maurice Wilkins 1962 den Nobelpreis für Medizin. ⓘ
Rosalind Franklin, deren Röntgenbeugungsdiagramme wesentlich zur Entschlüsselung der DNA-Struktur beigetragen hatten, war zu diesem Zeitpunkt bereits verstorben und konnte daher nicht mehr nominiert werden. ⓘ
Für weitere geschichtliche Informationen zur Entschlüsselung der Vererbungsvorgänge siehe „Forschungsgeschichte des Zellkerns“ sowie „Forschungsgeschichte der Chromosomen“ und „Chromosomentheorie der Vererbung“. ⓘ
1909 identifizierte Phoebus Levene die Basen-, Zucker- und Phosphat-Nukleotid-Einheit der RNA (damals "Hefe-Nukleinsäure" genannt). Im Jahr 1929 identifizierte Levene den Desoxyribose-Zucker in der Thymus-Nukleinsäure (DNA). Levene vermutete, dass die DNA aus einer Kette von vier Nukleotideinheiten besteht, die durch Phosphatgruppen miteinander verbunden sind ("Tetranukleotid-Hypothese"). Levene ging davon aus, dass die Kette kurz ist und sich die Basen in einer festen Reihenfolge wiederholen. 1927 schlug Nikolai Koltsov vor, dass vererbte Eigenschaften über ein "riesiges Erbmolekül" vererbt werden, das aus "zwei Spiegelsträngen besteht, die sich auf semikonservative Weise replizieren und dabei jeden Strang als Vorlage verwenden". 1928 entdeckte Frederick Griffith in seinem Experiment, dass Merkmale der "glatten" Form von Pneumokokken auf die "raue" Form desselben Bakteriums übertragen werden konnten, indem abgetötete "glatte" Bakterien mit der lebenden "rauen" Form gemischt wurden. Dieses System lieferte den ersten eindeutigen Hinweis darauf, dass die DNA genetische Informationen trägt. ⓘ
1933 schlug Jean Brachet bei der Untersuchung jungfräulicher Seeigel-Eier vor, dass die DNA im Zellkern und die RNA ausschließlich im Zytoplasma vorkommt. Damals ging man davon aus, dass die "Hefe-Nukleinsäure" (RNA) nur in Pflanzen und die "Thymus-Nukleinsäure" (DNA) nur in Tieren vorkommt. Letztere wurde für ein Tetramer gehalten, das die Aufgabe hat, den zellulären pH-Wert zu puffern. ⓘ
In einem einflussreichen Vortrag im Jahr 1957 legte Crick das zentrale Dogma der Molekularbiologie dar, das die Beziehung zwischen DNA, RNA und Proteinen voraussagte, und formulierte die "Adaptor-Hypothese". Die endgültige Bestätigung des Replikationsmechanismus, der durch die doppelhelicale Struktur impliziert wurde, erfolgte 1958 durch das Meselson-Stahl-Experiment. Weitere Arbeiten von Crick und Mitarbeitern zeigten, dass der genetische Code auf sich nicht überschneidenden Tripletts von Basen, den so genannten Codons, beruht, und ermöglichten Har Gobind Khorana, Robert W. Holley und Marshall Warren Nirenberg die Entschlüsselung des genetischen Codes. Diese Erkenntnisse sind die Geburtsstunde der Molekularbiologie. ⓘ
Bezeichnung
Die Bezeichnung Desoxyribonukleinsäure ist ein Wort, das sich aus mehreren Komponenten zusammensetzt: des (von des-), oxy (von den ersten beiden Silben von Oxygenium für Sauerstoff), ribo (von den ersten beiden Silben von Ribose) – somit Desoxyribo (für Desoxyribose) – und nukleinsäure (von Nuklein und Säure). Im deutschen Sprachgebrauch wird die Desoxyribonukleinsäure inzwischen überwiegend mit der englischen Abkürzung für deoxyribonucleic acid als DNA bezeichnet, während die Abkürzung DNS nach dem Duden als „veraltend“ gilt. ⓘ
Aufbau und Organisation

Chromatin und Chromosomen

Organisiert ist die DNA in der eukaryotischen Zelle in Form von Chromatinfäden, genannt Chromosomen, die im Zellkern liegen. Ein einzelnes Chromosom enthält von der Anaphase bis zum Beginn der S-Phase einen langen, durchgehenden DNA-Doppelstrang (in einem Chromatid). Am Ende der S-Phase besteht das Chromosom aus zwei identischen DNA-Fäden (in zwei Chromatiden). ⓘ
Da ein solcher DNA-Faden mehrere Zentimeter lang sein kann, ein Zellkern aber nur wenige Mikrometer Durchmesser hat, muss die DNA zusätzlich komprimiert bzw. „gepackt“ werden. Dies geschieht bei Eukaryoten mit sogenannten Chromatinproteinen, von denen besonders die basischen Histone zu erwähnen sind. Sie bilden die Nukleosomen, um die die DNA auf der niedrigsten Verpackungsebene herumgewickelt wird. Während der Kernteilung (Mitose) wird jedes Chromosom zu seiner maximal kompakten Form kondensiert. Dadurch können sie mit dem Lichtmikroskop besonders gut in der Metaphase identifiziert werden. ⓘ
Bakterielle und virale DNA
In prokaryotischen Zellen liegt die doppelsträngige DNA in den bisher dokumentierten Fällen mehrheitlich nicht als lineare Stränge mit jeweils einem Anfang und einem Ende vor, sondern als zirkuläre Moleküle – jedes Molekül (d. h. jeder DNA-Strang) schließt sich mit seinem 3'- und seinem 5'-Ende zum Kreis. Diese zwei ringförmigen, geschlossenen DNA-Moleküle werden je nach Länge der Sequenz als Bakterienchromosom oder Plasmid bezeichnet. Sie befinden sich bei Bakterien auch nicht in einem Zellkern, sondern liegen frei im Plasma vor. Die Prokaryoten-DNA wird mit Hilfe von Enzymen (zum Beispiel Topoisomerasen und Gyrasen) zu einfachen „Supercoils“ aufgewickelt, die einer geringelten Telefonschnur ähneln. Indem die Helices noch um sich selbst gedreht werden, sinkt der Platzbedarf für die Erbinformation. In den Bakterien sorgen Topoisomerasen dafür, dass durch ständiges Schneiden und Wiederverknüpfen der DNA der verdrillte Doppelstrang an einer gewünschten Stelle entwunden wird (Voraussetzung für Transkription und Replikation). Viren enthalten je nach Typ als Erbinformation entweder DNA oder RNA. Sowohl bei den DNA- wie den RNA-Viren wird die Nukleinsäure durch eine Protein-Hülle geschützt. ⓘ
Chemische und physikalische Eigenschaften der DNA-Doppelhelix
Stapelwechselwirkungen
Für die Stabilität der Doppelhelix sind hauptsächlich zwei Faktoren verantwortlich: die Basenpaarung zwischen komplementären Basen sowie Stapelwechselwirkungen (stacking interactions) zwischen aufeinanderfolgenden Basen. ⓘ
Anders als zunächst angenommen, ist der Energiegewinn durch Wasserstoffbrückenbindungen vernachlässigbar, da die Basen mit dem umgebenden Wasser ähnlich gute Wasserstoffbrückenbindungen eingehen können. Die Wasserstoffbrücken eines GC-Basenpaares tragen nur minimal zur Stabilität der Doppelhelix bei, während diejenigen eines AT-Basenpaares sogar destabilisierend wirken. Stapelwechselwirkungen hingegen wirken nur in der Doppelhelix zwischen aufeinanderfolgenden Basenpaaren: Zwischen den aromatischen Ringsystemen der heterozyklischen Basen entsteht eine dipol-induzierte Dipol-Wechselwirkung, welche energetisch günstig ist. Somit ist die Bildung des ersten Basenpaares aufgrund des geringen Energiegewinnes und des -verlustes recht ungünstig, jedoch die Elongation (Verlängerung) der Helix ist energetisch günstig, da die Basenpaarstapelung unter Energiegewinn verläuft. ⓘ
Die Stapelwechselwirkungen sind jedoch sequenzabhängig und energetisch am günstigsten für gestapelte GC-GC, weniger günstig für gestapelte AT-AT. Die Unterschiede in den Stapelwechselwirkungen erklären hauptsächlich, warum GC-reiche DNA-Abschnitte thermodynamisch stabiler sind als AT-reiche, während Wasserstoffbrückenbildung eine untergeordnete Rolle spielt. ⓘ
Nicht-Standard-Basen
Gelegentlich werden in Viren und zellulären Organismen Abweichungen von den oben genannten vier kanonischen Basen (Standard-Basen) Adenin (A), Guanin (G), Thymin (T) und Cytosin (C) beobachtet; weitere Abweichungen können künstlich erzeugt werden. ⓘ
Synthetische Basen
Im Labor wurde DNA (und auch RNA) mit weiteren künstlichen Basen versehen. Ziel ist es meist, damit unnatürliche Basenpaarungen (englisch unnatural base pairs, UBP) zu erzeugen:
- Im Jahr 2004 wurde DNA erzeugt, die statt der vier Standardnukleobasen (A, T, G und C) ein erweitertes Alphabet mit sechs Nukleobasen (A, T, G, C, dP und dZ) enthielt. Dabei steht bei diesen zwei neuen Basen dP für 2-Amino-8-(1′-β-D-2′-desoxyribofuranosyl)-imidazo[1,2-a]-1,3,5-triazin-4(8H)-on und dZ für 6-Amino-5-nitro-3-(1′-β-D-2′-desoxyribofuranosyl)-2(1H)-pyridon.
- Im Jahr 2006 wurden erstmals eine DNA mit um eine Benzolgruppe bzw. eine Naphthylgruppe erweiterten Basen untersucht (je nach Stellung der Erweiterungsgruppen entweder xDNA bzw. xxDNA oder yDNA bzw. yyDNA genannt).
- Yorke Zhang et al. berichteten zur Jahreswende 2016/2017 über halbsynthetische Organismen mit einer DNA, die um die Basen X (alias NaM) und Y' (alias TPT3) bzw. die (Desoxyribo-)Nukleotide dX (dNaM) und dY' (dTPT3) erweitert wurde, die miteinander paaren. Vorausgegangen waren Versuche mit Paarungen auf Basis der Basen X und Y (alias 5SICS), d. h. der Nukleotiden dX und dY (alias d5SICS). Weitere Basen, die mit 5SICS paaren können, sind FEMO und MMO2.
- Anfangs 2019 wurde über DNA und RNA mit jeweils acht Basen (vier natürliche und vier synthetische) berichtet, die sich alle paarweise einander zuordnen (Hachimoji-DNA). ⓘ
Enantiomere
DNA tritt in Lebewesen als D-DNA auf; L-DNA als Enantiomer (Spiegelmer) kann allerdings synthetisiert werden (gleiches gilt analog für RNA). L-DNA wird langsamer von Enzymen abgebaut als die natürliche Form, was sie für die Pharmaforschung interessant macht. ⓘ
Denaturierung

Die Basenpaarung von DNA wird bei verschiedenen zellulären Vorgängen denaturiert. Die Basenpaarung wird dabei durch verschiedene DNA-bindende Proteine abschnittsweise aufgehoben, z. B. bei der Replikation oder der Transkription. Der Ort des Denaturierungsbeginns wird als Denaturierungsblase bezeichnet und im Poland-Scheraga-Modell beschrieben. Jedoch wird die DNA-Sequenz, die Steifigkeit und die Torsion nicht miteinbezogen. Die Lebensdauer einer Denaturierungsblase beträgt zwischen einer Mikrosekunde und einer Millisekunde. ⓘ
Im Labor kann DNA durch physikalische und chemische Methoden denaturiert werden. DNA wird durch Formamid, Dimethylformamid, Guanidiniumsalze, Natriumsalicylat, Sulfoxid, Dimethylsulfoxid (DMSO), verschiedene Alkohole, Propylenglykol und Harnstoff denaturiert, meist in Kombination mit Wärme. Auch konzentrierte Lösungen von Natriumhydroxid denaturieren DNA. Bei den chemischen Methoden erfolgt eine Absenkung der Schmelztemperatur der doppelsträngigen DNA. ⓘ
„Alte“ DNA
Als aDNA („ancient DNA“; alte DNA) werden Reste von Erbgutmolekülen in toten Organismen bezeichnet, wenn keine direkten Verwandten des beprobten Organismus mehr leben. Auch wird die DNA des Menschen dann als aDNA bezeichnet, wenn das Individuum mindestens 75 Jahre vor der Probenuntersuchung verstorben ist. ⓘ
