Ribonukleinsäure

| Teil einer Serie über ⓘ |
| Genetik |
|---|
 |
|
Die Ribonukleinsäure (RNA) ist ein polymeres Molekül, das in verschiedenen biologischen Funktionen bei der Kodierung, Dekodierung, Regulierung und Expression von Genen eine wichtige Rolle spielt. RNA und Desoxyribonukleinsäure (DNA) sind Nukleinsäuren. Zusammen mit Lipiden, Proteinen und Kohlenhydraten bilden die Nukleinsäuren eines der vier wichtigsten Makromoleküle, die für alle bekannten Lebensformen wesentlich sind. Wie die DNA ist auch die RNA aus einer Kette von Nukleotiden aufgebaut, doch im Gegensatz zur DNA kommt sie in der Natur als Einzelstrang vor, der auf sich selbst gefaltet ist, und nicht als gepaarter Doppelstrang. Zelluläre Organismen verwenden Boten-RNA (mRNA), um genetische Informationen zu übermitteln (unter Verwendung der Stickstoffbasen Guanin, Uracil, Adenin und Cytosin, die mit den Buchstaben G, U, A und C bezeichnet werden), die die Synthese bestimmter Proteine steuern. Viele Viren kodieren ihre genetische Information mit Hilfe eines RNA-Genoms. ⓘ
Einige RNA-Moleküle spielen eine aktive Rolle in den Zellen, indem sie biologische Reaktionen katalysieren, die Genexpression steuern oder Reaktionen auf zelluläre Signale wahrnehmen und weiterleiten. Einer dieser aktiven Prozesse ist die Proteinsynthese, eine universelle Funktion, bei der RNA-Moleküle die Synthese von Proteinen an Ribosomen steuern. Bei diesem Prozess werden Aminosäuren mithilfe von Transfer-RNA-Molekülen (tRNA) an das Ribosom geliefert, wo die ribosomale RNA (rRNA) dann Aminosäuren zu kodierten Proteinen verknüpft. ⓘ

Ribonukleinsäure (Ribo|nuklein|säure; kurz RNS; englisch RNA für ribonucleic acid) (lat.-fr.-gr. Kunstwort) ist eine Nukleinsäure, die sich als Polynukleotid aus einer Kette von vielen Nukleotiden zusammensetzt. Das Biomolekül ist bei bestimmten Virentypen (RNA-Viren, Retroviren) sowie den hypothetischen urzeitlichen Ribozyten Träger der Erbinformation, also die materielle Basis der Gene. Das Wort setzt sich zusammen aus Ribose und Nukleinsäure. ⓘ
RNA kann auch Aufgaben von Enzymen übernehmen (Ribozym) oder ähnlich Antikörpern wirken (Aptamer). ⓘ
Vergleich mit der DNA

Wie die DNA enthalten die meisten biologisch aktiven RNAs, darunter mRNA, tRNA, rRNA, snRNAs und andere nichtcodierende RNAs, selbstkomplementäre Sequenzen, die es Teilen der RNA ermöglichen, sich zu falten und mit sich selbst zu paaren, um Doppelhelixen zu bilden. Die Analyse dieser RNAs hat gezeigt, dass sie stark strukturiert sind. Im Gegensatz zur DNA bestehen ihre Strukturen nicht aus langen Doppelhelixen, sondern aus Ansammlungen von kurzen Helices, die zu proteinähnlichen Strukturen zusammengefügt sind. ⓘ
Auf diese Weise können RNAs eine chemische Katalyse durchführen (wie Enzyme). So hat die Bestimmung der Struktur des Ribosoms - eines RNA-Protein-Komplexes, der die Bildung von Peptidbindungen katalysiert - gezeigt, dass sein aktives Zentrum vollständig aus RNA besteht. ⓘ
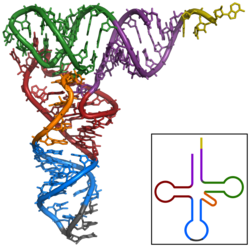
Struktur
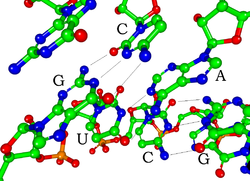
Jedes Nukleotid in der RNA enthält einen Ribosezucker, wobei die Kohlenstoffe mit 1' bis 5' nummeriert sind. An die 1'-Position ist eine Base gebunden, im Allgemeinen Adenin (A), Cytosin (C), Guanin (G) oder Uracil (U). Adenin und Guanin sind Purine, Cytosin und Uracil sind Pyrimidine. Eine Phosphatgruppe ist an der 3'-Position einer Ribose und an der 5'-Position der nächsten gebunden. Die Phosphatgruppen sind jeweils negativ geladen, was die RNA zu einem geladenen Molekül (Polyanion) macht. Die Basen bilden Wasserstoffbrücken zwischen Cytosin und Guanin, zwischen Adenin und Uracil und zwischen Guanin und Uracil. Es sind jedoch auch andere Wechselwirkungen möglich, z. B. eine Gruppe von Adeninbasen, die sich in einem Wulst aneinander binden, oder das GNRA-Tetraloop, das ein Guanin-Adenin-Basenpaar aufweist. ⓘ

Eine wichtige strukturelle Komponente der RNA, die sie von der DNA unterscheidet, ist das Vorhandensein einer Hydroxylgruppe an der 2'-Position des Ribosezuckers. Das Vorhandensein dieser funktionellen Gruppe führt dazu, dass die Helix meist die A-Form annimmt, obwohl die RNA in Einzelstrang-Dinukleotid-Kontexten selten auch die B-Form annehmen kann, die bei der DNA am häufigsten zu beobachten ist. Die A-Form-Geometrie führt zu einer sehr tiefen und schmalen Hauptrille und einer flachen und breiten Nebenrille. Eine zweite Folge des Vorhandenseins der 2'-Hydroxylgruppe ist, dass sie in konformativ flexiblen Bereichen eines RNA-Moleküls (d. h. nicht an der Bildung einer Doppelhelix beteiligt) die benachbarte Phosphodiesterbindung chemisch angreifen kann, um das Rückgrat zu spalten. ⓘ
Die RNA wird mit nur vier Basen (Adenin, Cytosin, Guanin und Uracil) transkribiert, aber diese Basen und die daran gebundenen Zucker können während der Reifung der RNA auf vielfältige Weise verändert werden. Pseudouridin (Ψ), bei dem die Bindung zwischen Uracil und Ribose von einer C-N-Bindung in eine C-C-Bindung umgewandelt wird, und Ribothymidin (T) sind an verschiedenen Stellen zu finden (am auffälligsten in der TΨC-Schleife der tRNA). Eine weitere bemerkenswerte modifizierte Base ist Hypoxanthin, eine desaminierte Adeninbase, deren Nukleosid Inosin (I) genannt wird. Inosin spielt eine Schlüsselrolle in der Wobble-Hypothese des genetischen Codes. ⓘ
Es gibt mehr als 100 weitere natürlich vorkommende modifizierte Nukleoside. Die größte strukturelle Vielfalt an Modifikationen findet sich in der tRNA, während Pseudouridin und Nukleoside mit 2'-O-Methylribose, die häufig in der rRNA vorkommen, am häufigsten sind. Die spezifischen Funktionen vieler dieser Modifikationen in der RNA sind noch nicht vollständig geklärt. Es ist jedoch bemerkenswert, dass in der ribosomalen RNA viele der posttranskriptionellen Modifikationen in hochfunktionalen Regionen wie dem Peptidyltransferasezentrum und der Schnittstelle der Untereinheiten auftreten, was bedeutet, dass sie für die normale Funktion wichtig sind. ⓘ
Die funktionelle Form einzelsträngiger RNA-Moleküle erfordert, genau wie Proteine, häufig eine spezifische Tertiärstruktur. Das Gerüst für diese Struktur wird durch sekundäre Strukturelemente bereitgestellt, die Wasserstoffbrückenbindungen innerhalb des Moleküls darstellen. Dies führt zu mehreren erkennbaren "Domänen" der Sekundärstruktur wie Haarnadelschleifen, Ausbuchtungen und internen Schleifen. Um eine RNA für eine beliebige Sekundärstruktur zu schaffen, d. h. zu entwerfen, reichen zwei oder drei Basen nicht aus, sondern vier Basen. Dies ist wahrscheinlich der Grund, warum sich die Natur für ein Alphabet mit vier Basen entschieden hat: Mit weniger als vier Basen lassen sich nicht alle Strukturen erzeugen, während mehr als vier Basen nicht notwendig sind. Da RNA geladen ist, werden Metallionen wie Mg2+ benötigt, um viele Sekundär- und Tertiärstrukturen zu stabilisieren. ⓘ
Das natürlich vorkommende Enantiomer der RNA ist die D-RNA, die aus D-Ribonukleotiden besteht. Alle Chiralitätszentren befinden sich in der D-Ribose. Durch die Verwendung von L-Ribose bzw. L-Ribonukleotiden kann L-RNA synthetisiert werden. L-RNA ist wesentlich stabiler gegenüber dem Abbau durch RNase. ⓘ
Wie andere strukturierte Biopolymere, z. B. Proteine, kann man die Topologie eines gefalteten RNA-Moleküls definieren. Dies geschieht häufig auf der Grundlage der Anordnung der Intrakettenkontakte innerhalb einer gefalteten RNA, die als Schaltkreistopologie bezeichnet wird. ⓘ
Synthese
Die Synthese von RNA wird in der Regel durch ein Enzym - die RNA-Polymerase - katalysiert, das die DNA als Vorlage verwendet, ein Prozess, der als Transkription bezeichnet wird. Die Transkription beginnt mit der Bindung des Enzyms an eine Promotorsequenz in der DNA (in der Regel "stromaufwärts" eines Gens). Die DNA-Doppelhelix wird durch die Helicase-Aktivität des Enzyms aufgerollt. Das Enzym bewegt sich dann entlang des Template-Strangs in 3'-5'-Richtung und synthetisiert ein komplementäres RNA-Molekül, wobei die Elongation in 5'-3'-Richtung erfolgt. Die DNA-Sequenz gibt auch vor, wo die RNA-Synthese beendet wird. ⓘ
Primäre Transkript-RNAs werden nach der Transkription häufig durch Enzyme modifiziert. So werden beispielsweise ein Poly(A)-Schwanz und eine 5'-Kappe an die eukaryotische prä-mRNA angehängt, und Introns werden durch das Spleißosom entfernt. ⓘ
Es gibt auch eine Reihe von RNA-abhängigen RNA-Polymerasen, die RNA als Vorlage für die Synthese eines neuen RNA-Strangs verwenden. So verwenden beispielsweise eine Reihe von RNA-Viren (wie das Poliovirus) diese Art von Enzymen zur Replikation ihres genetischen Materials. Außerdem ist die RNA-abhängige RNA-Polymerase in vielen Organismen Teil des RNA-Interferenzweges. ⓘ
Arten von RNA
Überblick

Messenger-RNA (mRNA) ist die RNA, die Informationen von der DNA zum Ribosom, dem Ort der Proteinsynthese (Übersetzung) in der Zelle, transportiert. Die mRNA ist eine Kopie der DNA. Die kodierende Sequenz der mRNA bestimmt die Aminosäuresequenz des entstehenden Proteins. Viele RNAs kodieren jedoch nicht für Proteine (etwa 97 % der Transkriptionsleistung in Eukaryonten sind nicht-protein-kodierend). ⓘ
Diese so genannten nicht-kodierenden RNAs ("ncRNA") können von ihren eigenen Genen (RNA-Genen) kodiert werden, aber auch von mRNA-Introns abstammen. Die bekanntesten Beispiele für nicht codierende RNAs sind die Transfer-RNA (tRNA) und die ribosomale RNA (rRNA), die beide am Prozess der Translation beteiligt sind. Es gibt auch nichtcodierende RNAs, die an der Genregulation, der RNA-Verarbeitung und anderen Aufgaben beteiligt sind. Bestimmte RNAs sind in der Lage, chemische Reaktionen zu katalysieren, z. B. das Schneiden und Ligieren anderer RNA-Moleküle und die Katalyse der Bildung von Peptidbindungen im Ribosom; diese sind als Ribozyme bekannt. ⓘ
Die Länge
Je nach Länge der RNA-Kette gibt es kleine RNAs und lange RNAs. Kleine RNAs sind in der Regel kürzer als 200 nt, lange RNAs sind länger als 200 nt. Zu den langen RNAs, die auch als große RNAs bezeichnet werden, gehören vor allem lange nicht codierende RNA (lncRNA) und mRNA. Zu den kleinen RNAs gehören hauptsächlich die 5.8S ribosomale RNA (rRNA), die 5S rRNA, die Transfer-RNA (tRNA), die microRNA (miRNA), die small interfering RNA (siRNA), die small nucleolar RNA (snoRNAs), die Piwi-interacting RNA (piRNA), die von der tRNA abgeleitete small RNA (tsRNA) und die small rDNA-derived RNA (srRNA). Es gibt einige Ausnahmen wie die 5S rRNA der Mitglieder der Gattung Halococcus (Archaea), die eine Einfügung aufweisen, wodurch ihre Größe erhöht wird. ⓘ
Bei der Übersetzung
Die Messenger-RNA (mRNA) überträgt die Information über eine Proteinsequenz zu den Ribosomen, den Proteinsynthesewerken in der Zelle. Sie ist so kodiert, dass alle drei Nukleotide (ein Codon) einer Aminosäure entsprechen. In eukaryontischen Zellen wird die mRNA-Vorstufe (pre-mRNA), nachdem sie von der DNA transkribiert wurde, zu reifer mRNA verarbeitet. Dabei werden die Introns - nicht codierende Abschnitte der prä-mRNA - entfernt. Die mRNA wird dann aus dem Zellkern in das Zytoplasma exportiert, wo sie an Ribosomen gebunden und mit Hilfe von tRNA in die entsprechende Proteinform übersetzt wird. In prokaryontischen Zellen, die nicht über einen Kern und ein Zytoplasma verfügen, kann die mRNA an Ribosomen binden, während sie von der DNA transkribiert wird. Nach einer gewissen Zeit wird die Nachricht mit Hilfe von Ribonukleasen in ihre einzelnen Nukleotide zerlegt. ⓘ
Die Transfer-RNA (tRNA) ist eine kleine RNA-Kette von etwa 80 Nukleotiden, die während der Translation eine bestimmte Aminosäure auf eine wachsende Polypeptidkette am ribosomalen Ort der Proteinsynthese überträgt. Sie verfügt über Stellen für die Anlagerung von Aminosäuren und eine Anticodon-Region für die Codon-Erkennung, die sich durch Wasserstoffbrückenbindungen an eine bestimmte Sequenz der Boten-RNA-Kette bindet. ⓘ
Die ribosomale RNA (rRNA) ist die katalytische Komponente der Ribosomen. Die rRNA ist der Bestandteil des Ribosoms, der die Translation beherbergt. Eukaryotische Ribosomen enthalten vier verschiedene rRNA-Moleküle: 18S, 5,8S, 28S und 5S rRNA. Drei der rRNA-Moleküle werden im Nukleolus synthetisiert, ein weiteres an anderer Stelle. Im Zytoplasma verbinden sich ribosomale RNA und Protein zu einem Nukleoprotein, dem Ribosom. Das Ribosom bindet die mRNA und führt die Proteinsynthese durch. An eine einzelne mRNA können jederzeit mehrere Ribosomen gebunden sein. Nahezu die gesamte RNA in einer typischen eukaryontischen Zelle ist rRNA. ⓘ
Transfer-Messenger-RNA (tmRNA) findet sich in vielen Bakterien und Plastiden. Sie markiert Proteine, die von mRNAs kodiert werden, denen Stoppcodons zum Abbau fehlen, und verhindert, dass das Ribosom zum Stillstand kommt. ⓘ
Regulatorische RNA
Die ersten bekannten Regulatoren der Genexpression waren Proteine, die als Repressoren und Aktivatoren bekannt sind - Regulatoren mit spezifischen kurzen Bindungsstellen in Enhancer-Regionen in der Nähe der zu regulierenden Gene. Spätere Studien haben gezeigt, dass auch RNAs Gene regulieren. In Eukaryonten gibt es mehrere Arten von RNA-abhängigen Prozessen, die die Expression von Genen an verschiedenen Stellen regulieren, z. B. RNAi, die Gene posttranskriptiv unterdrückt, lange nicht-kodierende RNAs, die Chromatinblöcke epigenetisch abschalten, und Enhancer-RNAs, die eine verstärkte Genexpression induzieren. Es wurde auch gezeigt, dass Bakterien und Archaeen regulatorische RNA-Systeme wie bakterielle kleine RNAs und CRISPR nutzen. Fire und Mello erhielten 2006 den Nobelpreis für Physiologie oder Medizin für die Entdeckung der microRNAs (miRNAs), spezifischer kurzer RNA-Moleküle, die Basenpaare mit mRNAs bilden können. ⓘ
RNA-Interferenz durch miRNAs
Die posttranskriptionale Expression vieler Gene kann durch RNA-Interferenz gesteuert werden, bei der sich miRNAs, spezifische kurze RNA-Moleküle, mit mRNA-Regionen paaren und sie gezielt abbauen. Dieser auf Antisense basierende Prozess umfasst Schritte, bei denen die RNA zunächst so bearbeitet wird, dass sie eine Basenpaarung mit einer Region ihrer Ziel-mRNA eingehen kann. Sobald die Basenpaarung stattgefunden hat, weisen andere Proteine die mRNA an, durch Nukleasen zerstört zu werden. ⓘ
Lange nicht-kodierende RNAs
Als nächstes wurden Xist und andere lange nichtcodierende RNAs, die mit der Inaktivierung des X-Chromosoms in Verbindung stehen, mit der Regulierung in Verbindung gebracht. Jeannie T. Lee und andere haben gezeigt, dass ihre zunächst rätselhafte Rolle darin besteht, Chromatinblöcke durch Rekrutierung des Polycomb-Komplexes zum Schweigen zu bringen, so dass keine Boten-RNA von ihnen transkribiert werden kann. Weitere lncRNAs, die derzeit als RNAs mit mehr als 200 Basenpaaren definiert sind, die kein kodierendes Potenzial zu haben scheinen, wurden mit der Regulierung der Pluripotenz von Stammzellen und der Zellteilung in Verbindung gebracht. ⓘ
Enhancer-RNAs
Die dritte große Gruppe der regulatorischen RNAs wird als Enhancer-RNAs bezeichnet. Derzeit ist nicht klar, ob es sich um eine eigene Kategorie von RNAs unterschiedlicher Länge handelt oder ob sie eine eigene Untergruppe der lncRNAs darstellen. In jedem Fall werden sie von Enhancern aus transkribiert, bei denen es sich um bekannte regulatorische Stellen in der DNA in der Nähe von Genen handelt, die sie regulieren. Sie regulieren die Transkription des Gens bzw. der Gene unter der Kontrolle des Enhancers, von dem sie transkribiert werden. ⓘ
Regulatorische RNA in Prokaryonten
Zunächst nahm man an, dass regulatorische RNA ein eukaryotisches Phänomen sei, ein Teil der Erklärung dafür, warum in höheren Organismen so viel mehr Transkription beobachtet wurde, als vorhergesagt worden war. Doch sobald Forscher begannen, in Bakterien nach möglichen RNA-Regulatoren zu suchen, tauchten sie auch dort auf, und zwar als so genannte kleine RNA (sRNA). Gegenwärtig wird die Allgegenwärtigkeit von Systemen der RNA-Regulierung von Genen als Unterstützung für die RNA-Welt-Theorie diskutiert. Bakterielle kleine RNAs wirken im Allgemeinen durch Antisense-Paarung mit mRNA, um deren Translation herunterzuregulieren, entweder durch Beeinträchtigung der Stabilität oder durch Beeinträchtigung der Cis-Bindungsfähigkeit. Es wurden auch Riboswitches entdeckt. Dabei handelt es sich um cis-wirksame regulatorische RNA-Sequenzen, die allosterisch wirken. Sie verändern ihre Form, wenn sie Metaboliten binden, so dass sie die Fähigkeit, Chromatin zu binden, um die Expression von Genen zu regulieren, gewinnen oder verlieren. ⓘ
Auch Archaeen verfügen über Systeme regulatorischer RNA. Das CRISPR-System, das seit kurzem zur DNA-Editierung in situ eingesetzt wird, wirkt über regulatorische RNAs in Archaeen und Bakterien und bietet Schutz vor Virusinvasoren. ⓘ
Bei der RNA-Verarbeitung

Viele RNAs sind an der Modifikation anderer RNAs beteiligt. Introns werden von Spleißosomen, die mehrere kleine Kern-RNAs (snRNAs) enthalten, aus der prä-mRNA herausgespleißt, oder die Introns können Ribozyme sein, die von selbst gespleißt werden. RNA kann auch verändert werden, indem ihre Nukleotide zu anderen Nukleotiden als A, C, G und U modifiziert werden. In Eukaryonten werden Modifikationen von RNA-Nukleotiden im Allgemeinen durch kleine nukleolare RNAs (snoRNA; 60-300 nt) gesteuert, die im Nukleolus und in den Cajal-Körpern zu finden sind. snoRNAs verbinden sich mit Enzymen und leiten sie zu einer Stelle auf einer RNA, indem sie ein Basenpaar mit dieser RNA bilden. Diese Enzyme führen dann die Nukleotidmodifikation durch. rRNAs und tRNAs werden in großem Umfang modifiziert, aber auch snRNAs und mRNAs können das Ziel einer Basenmodifikation sein. RNA kann auch methyliert werden. ⓘ
RNA-Genome
Wie die DNA kann auch die RNA genetische Informationen enthalten. RNA-Viren haben Genome, die aus RNA bestehen, die für eine Reihe von Proteinen kodiert. Das virale Genom wird von einigen dieser Proteine repliziert, während andere Proteine das Genom schützen, wenn sich das Viruspartikel zu einer neuen Wirtszelle bewegt. Viroide sind eine weitere Gruppe von Krankheitserregern, die jedoch nur aus RNA bestehen, für kein Protein kodieren und von der Polymerase einer pflanzlichen Wirtszelle repliziert werden. ⓘ
Bei der reversen Transkription
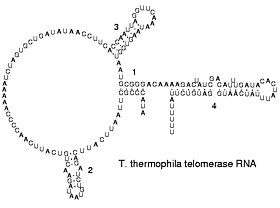
Reverse Transkriptionsviren replizieren ihre Genome durch reverse Transkription von DNA-Kopien ihrer RNA; diese DNA-Kopien werden dann in neue RNA umgeschrieben. Retrotransposons verbreiten sich ebenfalls, indem sie DNA und RNA voneinander kopieren, und Telomerase enthält eine RNA, die als Vorlage für den Aufbau der Enden eukaryontischer Chromosomen verwendet wird. ⓘ
Doppelstrangige RNA

Doppelstrangige RNA (dsRNA) ist eine RNA mit zwei komplementären Strängen, ähnlich der in allen Zellen vorkommenden DNA, bei der jedoch Thymin durch Uracil ersetzt und ein Sauerstoffatom hinzugefügt wurde. dsRNA bildet das genetische Material einiger Viren (doppelsträngige RNA-Viren). Doppelstrangige RNA, wie z. B. virale RNA oder siRNA, kann in Eukaryonten RNA-Interferenz und in Wirbeltieren eine Interferon-Reaktion auslösen. In Eukaryonten spielt doppelsträngige RNA (dsRNA) eine Rolle bei der Aktivierung des angeborenen Immunsystems gegen Virusinfektionen. ⓘ
Zirkuläre RNA
In den späten 1970er Jahren wurde gezeigt, dass es eine einzelsträngige, kovalent geschlossene, d.h. zirkuläre Form von RNA gibt, die im gesamten Tier- und Pflanzenreich vorkommt (siehe circRNA). Es wird angenommen, dass circRNAs durch eine "Back-Splice"-Reaktion entstehen, bei der das Spleißosom eine stromaufwärts gelegene 3'-Akzeptor- mit einer stromabwärts gelegenen 5'-Donor-Spleißstelle verbindet. Bisher ist die Funktion von circRNAs weitgehend unbekannt, obwohl für einige wenige Beispiele eine Sponging-Aktivität von microRNAs nachgewiesen wurde. ⓘ
Wichtige Entdeckungen in der RNA-Biologie

Die RNA-Forschung hat zu vielen wichtigen biologischen Entdeckungen und zahlreichen Nobelpreisen geführt. Nukleinsäuren wurden 1868 von Friedrich Miescher entdeckt, der das Material "Nuklein" nannte, da es sich im Zellkern befand. Später wurde entdeckt, dass auch prokaryotische Zellen, die keinen Zellkern haben, Nukleinsäuren enthalten. Die Rolle der RNA bei der Proteinsynthese wurde bereits 1939 vermutet. Severo Ochoa erhielt 1959 den Nobelpreis für Medizin (gemeinsam mit Arthur Kornberg), nachdem er ein Enzym entdeckt hatte, das RNA im Labor synthetisieren kann. Das von Ochoa entdeckte Enzym (Polynukleotidphosphorylase) erwies sich jedoch später als verantwortlich für den RNA-Abbau und nicht für die RNA-Synthese. 1956 hybridisierten Alex Rich und David Davies zwei getrennte RNA-Stränge, um den ersten RNA-Kristall zu bilden, dessen Struktur durch Röntgenkristallographie bestimmt werden konnte. ⓘ
Die Sequenz der 77 Nukleotide einer Hefe-tRNA wurde 1965 von Robert W. Holley gefunden, wofür Holley 1968 den Nobelpreis für Medizin erhielt (gemeinsam mit Har Gobind Khorana und Marshall Nirenberg). ⓘ
Anfang der 1970er Jahre wurden Retroviren und die reverse Transkriptase entdeckt, die zum ersten Mal zeigten, dass Enzyme RNA in DNA kopieren können (das Gegenteil des üblichen Weges zur Übertragung genetischer Informationen). Für diese Arbeit wurden David Baltimore, Renato Dulbecco und Howard Temin 1975 mit dem Nobelpreis ausgezeichnet. 1976 bestimmten Walter Fiers und sein Team die erste vollständige Nukleotidsequenz eines RNA-Virusgenoms, das des Bakteriophagen MS2. ⓘ
1977 wurden Introns und das RNA-Spleißen sowohl in Säugetierviren als auch in zellulären Genen entdeckt, was 1993 zu einem Nobelpreis für Philip Sharp und Richard Roberts führte. Katalytische RNA-Moleküle (Ribozyme) wurden in den frühen 1980er Jahren entdeckt und führten 1989 zur Verleihung des Nobelpreises an Thomas Cech und Sidney Altman. Im Jahr 1990 wurde in Petunia festgestellt, dass eingeführte Gene ähnliche Gene der Pflanze zum Schweigen bringen können, was heute als Ergebnis der RNA-Interferenz bekannt ist. ⓘ
Etwa zur gleichen Zeit wurde festgestellt, dass 22 nt lange RNAs, jetzt microRNAs genannt, eine Rolle bei der Entwicklung von C. elegans spielen. Für ihre Studien zur RNA-Interferenz erhielten Andrew Fire und Craig Mello 2006 den Nobelpreis, und im selben Jahr wurde Roger Kornberg ein weiterer Nobelpreis für seine Studien zur Transkription von RNA verliehen. Die Entdeckung genregulierender RNAs hat zu Versuchen geführt, Medikamente aus RNA zu entwickeln, wie z. B. siRNA, um Gene auszuschalten. Zusätzlich zu den Nobelpreisen für RNA-Forschung wurden 2009 die Preise für die Aufklärung der atomaren Struktur des Ribosoms an Venki Ramakrishnan, Thomas A. Steitz und Ada Yonath verliehen. ⓘ
Relevanz für die präbiotische Chemie und die Abiogenese
Im Jahr 1968 stellte Carl Woese die Hypothese auf, dass RNA katalytisch sein könnte, und schlug vor, dass die frühesten Formen des Lebens (selbstreplizierende Moleküle) auf RNA angewiesen gewesen sein könnten, um sowohl genetische Informationen zu tragen als auch biochemische Reaktionen zu katalysieren - eine RNA-Welt. Im Mai 2022 berichteten Wissenschaftler, dass sich RNA spontan auf präbiotischem Basalt-Lavaglas gebildet hat, das auf der frühen Erde vermutlich reichlich vorhanden war. ⓘ
Im März 2015 wurde berichtet, dass komplexe DNA- und RNA-Nukleotide, darunter Uracil, Cytosin und Thymin, im Labor unter Weltraumbedingungen mit Hilfe von Starterchemikalien wie Pyrimidin, einer organischen Verbindung, die häufig in Meteoriten gefunden wird, gebildet wurden. Pyrimidin ist wie die polyzyklischen aromatischen Kohlenwasserstoffe (PAK) eine der kohlenstoffreichsten Verbindungen im Universum und könnte in Roten Riesen oder in interstellaren Staub- und Gaswolken gebildet worden sein. Im Juli 2022 meldeten Astronomen die Entdeckung riesiger Mengen präbiotischer Moleküle, einschließlich RNA, im galaktischen Zentrum der Milchstraße. ⓘ
Aufbau und Unterschied zur DNA
Tertiärstruktur
Nukleinsäuren können ebenfalls komplexe räumliche Strukturen einnehmen: tRNAs müssen für ihre Funktion in der korrekten Tertiärstruktur vorliegen. ⓘ
Tertiärstruktur eines Pseudoknotens ⓘ
Die RNA-Welt-Hypothese
Die RNA-Welt-Hypothese besagt, dass RNA-Moleküle bei der chemischen Evolution vermutlich Vorläufer der Organismen waren. Die Hypothese lässt sich ableiten aus der Fähigkeit der RNA zur Speicherung, Übertragung und Vervielfältigung genetischer Informationen sowie aus ihrer Fähigkeit, als Ribozyme Reaktionen zu katalysieren. In einer Evolutionsumgebung würden diejenigen RNA-Moleküle gehäuft vorkommen, die sich selbst bevorzugt vermehren. ⓘ
RNA-Reinigung und Nachweis
RNA kann durch eine RNA-Reinigung, z. B. per RNA-Extraktion, von anderen Biomolekülen getrennt werden. Bestimmung der Menge und Reinheit der isolierten RNA erfolgt durch photometrische Messung bei einer Wellenlänge von 260 und 280 nm. Weitere Hinweise auf die Qualität der RNA erhält man durch Agarose-Gelelektrophorese gefolgt mit einer Anfärbung durch Farbstoffe wie SYBR Green II, Methylenblau, Stains-All oder durch eine Silberfärbung. Der qualitative Nachweis von RNA (ob eine bestimmte RNA vorliegt) erfolgt meistens durch eine RT-PCR, teilweise mit einer anschließenden DNA-Sequenzierung, oder durch einen Northern Blot. Der quantitative Nachweis (wie viel von einer bestimmten RNA vorliegt) erfolgt meistens durch eine qRT-PCR, bei gereinigten Proben mit nur einer RNA-Sequenz kann die Konzentration auch durch Photometrie bestimmt werden. Durch Molecular Combing kann die RNA gestreckt und ausgerichtet werden. Mittels In situ-Hybridisierung lassen sich spezifische RNAs in Zellen und Geweben ohne vorhergehende Isolierung nachweisen. ⓘ