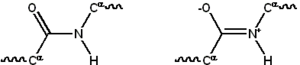Protein

Proteine sind große Biomoleküle und Makromoleküle, die aus einer oder mehreren langen Ketten von Aminosäureresten bestehen. Proteine erfüllen in Organismen eine Vielzahl von Funktionen, darunter die Katalyse von Stoffwechselreaktionen, die DNA-Replikation, die Reaktion auf Reize, die Strukturierung von Zellen und Organismen und den Transport von Molekülen von einem Ort zum anderen. Proteine unterscheiden sich voneinander in erster Linie durch ihre Aminosäuresequenz, die durch die Nukleotidsequenz ihrer Gene vorgegeben ist und in der Regel zur Faltung des Proteins in eine spezifische 3D-Struktur führt, die seine Aktivität bestimmt. ⓘ
Eine lineare Kette von Aminosäureresten wird als Polypeptid bezeichnet. Ein Protein enthält mindestens ein langes Polypeptid. Kurze Polypeptide, die weniger als 20-30 Reste enthalten, werden nur selten als Proteine betrachtet und gemeinhin als Peptide oder manchmal als Oligopeptide bezeichnet. Die einzelnen Aminosäurereste sind durch Peptidbindungen und benachbarte Aminosäurereste miteinander verbunden. Die Abfolge der Aminosäurereste in einem Protein wird durch die Sequenz eines Gens festgelegt, die im genetischen Code kodiert ist. Im Allgemeinen legt der genetische Code 20 Standardaminosäuren fest; bei bestimmten Organismen kann der genetische Code jedoch auch Selenocystein und - bei bestimmten Archaeen - Pyrrolysin enthalten. Kurz nach oder sogar während der Synthese werden die Reste eines Proteins häufig durch posttranslationale Modifikation chemisch verändert, wodurch sich die physikalischen und chemischen Eigenschaften, die Faltung, die Stabilität, die Aktivität und schließlich die Funktion der Proteine ändern. An einige Proteine sind Nicht-Peptidgruppen angehängt, die als prosthetische Gruppen oder Kofaktoren bezeichnet werden können. Proteine können auch zusammenarbeiten, um eine bestimmte Funktion zu erfüllen, und sie schließen sich oft zu stabilen Proteinkomplexen zusammen. ⓘ
Einmal gebildet, existieren Proteine nur für eine bestimmte Zeit und werden dann von der Zellmaschinerie durch den Prozess des Proteinumsatzes abgebaut und recycelt. Die Lebensdauer eines Proteins wird in Form seiner Halbwertszeit gemessen und umfasst eine große Bandbreite. Sie kann Minuten oder Jahre betragen, wobei die durchschnittliche Lebensdauer in Säugetierzellen bei 1-2 Tagen liegt. Abnormale oder falsch gefaltete Proteine werden schneller abgebaut, entweder weil sie gezielt zerstört werden sollen oder weil sie instabil sind. ⓘ
Wie andere biologische Makromoleküle, z. B. Polysaccharide und Nukleinsäuren, sind Proteine wesentliche Bestandteile von Organismen und an praktisch allen Prozessen in Zellen beteiligt. Viele Proteine sind Enzyme, die biochemische Reaktionen katalysieren und für den Stoffwechsel unerlässlich sind. Proteine haben auch strukturelle oder mechanische Funktionen, wie Aktin und Myosin im Muskel und die Proteine des Zytoskeletts, die ein Gerüstsystem bilden, das die Zellform aufrechterhält. Andere Proteine sind wichtig für die Zellsignalübertragung, Immunreaktionen, Zelladhäsion und den Zellzyklus. Bei Tieren werden Proteine in der Ernährung benötigt, um die essenziellen Aminosäuren zu liefern, die nicht synthetisiert werden können. Durch die Verdauung werden die Proteine für den Stoffwechsel aufgespalten. ⓘ
Proteine können mit Hilfe verschiedener Techniken wie Ultrazentrifugation, Ausfällung, Elektrophorese und Chromatographie von anderen Zellbestandteilen gereinigt werden; das Aufkommen der Gentechnik hat eine Reihe von Methoden zur Vereinfachung der Reinigung ermöglicht. Zu den gängigen Methoden zur Untersuchung von Proteinstruktur und -funktion gehören Immunhistochemie, ortsgerichtete Mutagenese, Röntgenkristallographie, Kernspinresonanz und Massenspektrometrie. ⓘ
Ein Protein, umgangssprachlich Eiweiß (veraltet Eiweißstoff) genannt, ist ein biologisches Makromolekül, das aus Aminosäuren aufgebaut wird, die durch Peptidbindungen verknüpft sind. ⓘ

Dieses Globin dient in Muskelzellen als Sauerstoffspeicher. Seine aus über 150 Aminosäuren aufgebaute Peptidkette mit α-Helices faltet sich kugelförmig zur räumlichen Proteinstruktur und hält eine Hämgruppe, an deren Eisenatom sich O2 anlagern kann. ⓘ
Proteine finden sich in jeder Zelle und machen zumeist mehr als die Hälfte des Trockengewichts aus. Sie dienen ihr als molekulare „Werkzeuge“ und erfüllen je nach der besonderen Struktur unterschiedliche Aufgaben, indem sie beispielsweise Zellbewegungen ermöglichen, Metabolite transportieren, Ionen pumpen, chemische Reaktionen katalysieren oder Signalstoffe erkennen können. Überwiegend aus Proteinen bestehen so auch Muskeln, Herz, Hirn, Haut und Haare. ⓘ
Die Gesamtheit aller Proteine in einem Lebewesen, einem Gewebe, einer Zelle oder einem Zellkompartiment, unter exakt definierten Bedingungen und zu einem bestimmten Zeitpunkt, wird als Proteom bezeichnet. ⓘ
Geschichte und Etymologie
Jahrhundert von Antoine Fourcroy und anderen als eine eigene Klasse biologischer Moleküle erkannt, die sich durch die Fähigkeit der Moleküle auszeichnete, bei Behandlung mit Hitze oder Säure zu koagulieren oder auszuflocken. Zu den bekannten Beispielen dieser Zeit gehörten Albumin aus Eiklar, Blutserumalbumin, Fibrin und Weizenkleber. ⓘ
Proteine wurden erstmals von dem niederländischen Chemiker Gerardus Johannes Mulder beschrieben und 1838 von dem schwedischen Chemiker Jöns Jacob Berzelius benannt. Mulder führte eine Elementaranalyse gängiger Proteine durch und stellte fest, dass fast alle Proteine die gleiche Summenformel hatten: C400H620N100O120P1S1. Er kam zu dem irrigen Schluss, dass sie aus einer einzigen Art von (sehr großen) Molekülen bestehen könnten. Der Begriff "Protein" zur Beschreibung dieser Moleküle wurde von Mulders Mitarbeiter Berzelius vorgeschlagen; Protein leitet sich vom griechischen Wort πρώτειος (proteios) ab, was so viel bedeutet wie "primär", "an der Spitze" oder "vorne stehend", + -in. Mulder fuhr fort, die Produkte des Proteinabbaus zu identifizieren, wie die Aminosäure Leucin, für die er ein (fast korrektes) Molekulargewicht von 131 Da fand. Vor dem Begriff "Protein" wurden andere Bezeichnungen verwendet, wie "Albumine" oder "Eiweißkörper". ⓘ
Frühe Ernährungswissenschaftler wie der Deutsche Carl von Voit waren der Ansicht, dass Eiweiß der wichtigste Nährstoff für die Aufrechterhaltung der Körperstruktur sei, da man allgemein glaubte, dass "Fleisch Fleisch macht". Karl Heinrich Ritthausen erweiterte die bekannten Proteinformen durch die Identifizierung der Glutaminsäure. An der Connecticut Agricultural Experiment Station wurde von Thomas Burr Osborne eine detaillierte Übersicht über die pflanzlichen Proteine erstellt. In Zusammenarbeit mit Lafayette Mendel und unter Anwendung des Liebig'schen Minimalgesetzes bei der Fütterung von Laborratten wurden die ernährungsphysiologisch wichtigen Aminosäuren ermittelt. Diese Arbeit wurde von William Cumming Rose fortgesetzt und weitergegeben. Das Verständnis der Proteine als Polypeptide kam durch die Arbeit von Franz Hofmeister und Hermann Emil Fischer im Jahr 1902. Die zentrale Rolle von Proteinen als Enzyme in lebenden Organismen wurde erst 1926 vollständig erkannt, als James B. Sumner zeigte, dass das Enzym Urease tatsächlich ein Protein ist. ⓘ
Die Schwierigkeit, Proteine in großen Mengen zu reinigen, machte es für die frühen Proteinbiochemiker sehr schwierig, sie zu untersuchen. Daher konzentrierten sich die frühen Studien auf Proteine, die in großen Mengen gereinigt werden konnten, z. B. aus Blut, Eiklar, verschiedenen Toxinen und Verdauungs-/Stoffwechselenzymen aus Schlachthöfen. In den 1950er Jahren reinigte die Armour Hot Dog Co. 1 kg reine Rinderpankreas-Ribonuklease A und stellte sie den Wissenschaftlern frei zur Verfügung; diese Geste trug dazu bei, dass die Ribonuklease A in den folgenden Jahrzehnten ein wichtiges Ziel für biochemische Untersuchungen wurde. ⓘ
Linus Pauling wird die erfolgreiche Vorhersage regelmäßiger Protein-Sekundärstrukturen auf der Grundlage von Wasserstoffbrückenbindungen zugeschrieben, eine Idee, die erstmals von William Astbury im Jahr 1933 vorgebracht wurde. Spätere Arbeiten von Walter Kauzmann über die Denaturierung, die teilweise auf früheren Studien von Kaj Linderstrøm-Lang basierten, trugen zu einem besseren Verständnis der Proteinfaltung und -struktur bei, die durch hydrophobe Wechselwirkungen vermittelt werden. ⓘ
Das erste Protein, das sequenziert wurde, war Insulin, das 1949 von Frederick Sanger entschlüsselt wurde. Sanger bestimmte korrekt die Aminosäuresequenz von Insulin und bewies damit eindeutig, dass Proteine aus linearen Aminosäurepolymeren und nicht aus verzweigten Ketten, Kolloiden oder Cyclolen bestehen. Für diese Leistung erhielt er 1958 den Nobelpreis. ⓘ

Mit der Entwicklung der Röntgenkristallographie wurde es möglich, Proteinstrukturen zu sequenzieren. Die ersten Proteinstrukturen, die gelöst wurden, waren Hämoglobin von Max Perutz und Myoglobin von Sir John Cowdery Kendrew im Jahr 1958. Der Einsatz von Computern und die zunehmende Rechenleistung ermöglichten auch die Sequenzierung komplexer Proteine. Im Jahr 1999 gelang Roger Kornberg die Sequenzierung der hochkomplexen Struktur der RNA-Polymerase mit Hilfe hochintensiver Röntgenstrahlen aus Synchrotrons. ⓘ
Seitdem wurde die Kryo-Elektronenmikroskopie für große makromolekulare Zusammenhänge entwickelt. Bei der Kryo-EM werden keine Kristalle, sondern gefrorene Proteinproben verwendet, und es werden Elektronenstrahlen statt Röntgenstrahlen eingesetzt. Dadurch wird die Probe weniger beschädigt, so dass die Wissenschaftler mehr Informationen erhalten und größere Strukturen analysieren können. Die computergestützte Vorhersage von Proteinstrukturen für kleine Proteindomänen hat den Forschern ebenfalls geholfen, sich der Auflösung von Proteinstrukturen auf atomarer Ebene zu nähern. Im Jahr 2017 verfügt die Protein Data Bank über 126.060 atomar aufgelöste Strukturen von Proteinen. ⓘ
Dass Proteine aus Aminosäurenketten über Peptidbindungen aufgebaut sind, wurde zuerst 1902 auf der 14. Versammlung deutscher Naturforscher und Ärzte unabhängig von Emil Fischer und Franz Hofmeister, die beide Vorträge hielten, vermutet. Fischer führte dabei den Begriff Peptid ein. ⓘ
Anzahl der in Genomen kodierten Proteine
Die Anzahl der in einem Genom kodierten Proteine entspricht in etwa der Anzahl der Gene (obwohl es eine beträchtliche Anzahl von Genen geben kann, die für RNA von Proteinen kodieren, z. B. ribosomale RNAs). Viren kodieren typischerweise für einige wenige bis einige hundert Proteine, Archaeen und Bakterien für einige hundert bis einige tausend, während Eukaryoten typischerweise für einige tausend bis zehntausende Proteine kodieren (siehe Genomgröße für eine Liste von Beispielen). ⓘ
Biochemie
Die meisten Proteine bestehen aus linearen Polymeren, die aus Reihen von bis zu 20 verschiedenen L-α-Aminosäuren aufgebaut sind. Alle proteinogenen Aminosäuren weisen gemeinsame strukturelle Merkmale auf, darunter einen α-Kohlenstoff, an den eine Aminogruppe, eine Carboxylgruppe und eine variable Seitenkette gebunden sind. Nur Prolin weicht von dieser Grundstruktur ab, da es einen ungewöhnlichen Ring an der N-endigen Amingruppe enthält, der die CO-NH-Amideinheit in eine feste Konformation zwingt. Die Seitenketten der Standardaminosäuren, die in der Liste der Standardaminosäuren aufgeführt sind, weisen eine große Vielfalt an chemischen Strukturen und Eigenschaften auf; es ist die kombinierte Wirkung aller Aminosäureseitenketten in einem Protein, die letztendlich seine dreidimensionale Struktur und seine chemische Reaktivität bestimmt. Die Aminosäuren in einer Polypeptidkette sind durch Peptidbindungen miteinander verbunden. Nach der Verknüpfung in der Proteinkette wird eine einzelne Aminosäure als Rest bezeichnet, und die verknüpfte Reihe von Kohlenstoff-, Stickstoff- und Sauerstoffatomen wird als Hauptkette oder Proteinrückgrat bezeichnet. ⓘ
Die Peptidbindung hat zwei Resonanzformen, die einen gewissen Doppelbindungscharakter haben und eine Drehung um ihre Achse verhindern, so dass die Alphakohlenstoffe ungefähr koplanar sind. Die beiden anderen Flächenwinkel der Peptidbindung bestimmen die lokale Form des Proteinrückgrats. Das Ende mit einer freien Aminogruppe wird als N-Terminus oder Aminoterminus bezeichnet, während das Ende des Proteins mit einer freien Carboxylgruppe als C-Terminus oder Carboxyterminus bezeichnet wird (die Sequenz des Proteins wird vom N-Terminus zum C-Terminus geschrieben, von links nach rechts). ⓘ
Die Begriffe Protein, Polypeptid und Peptid sind etwas zweideutig und können sich in ihrer Bedeutung überschneiden. Mit Protein wird im Allgemeinen das vollständige biologische Molekül in einer stabilen Konformation bezeichnet, während Peptid im Allgemeinen für kurze Aminosäure-Oligomere reserviert ist, die häufig keine stabile 3D-Struktur aufweisen. Die Grenze zwischen diesen beiden Begriffen ist jedoch nicht genau definiert und liegt in der Regel bei 20-30 Resten. Polypeptid kann sich auf jede einzelne lineare Kette von Aminosäuren beziehen, in der Regel unabhängig von ihrer Länge, impliziert aber oft das Fehlen einer definierten Konformation. ⓘ
Wechselwirkungen
Proteine können mit vielen Arten von Molekülen interagieren, u. a. mit anderen Proteinen, mit Lipiden, mit Kohlenhydraten und mit DNA. ⓘ
Häufigkeit in Zellen
Man schätzt, dass Bakterien durchschnittlicher Größe etwa 2 Millionen Proteine pro Zelle enthalten (z. B. E. coli und Staphylococcus aureus). Kleinere Bakterien wie Mykoplasmen oder Spirochäten enthalten weniger Moleküle, etwa 50.000 bis 1 Million. Im Gegensatz dazu sind eukaryontische Zellen größer und enthalten daher viel mehr Proteine. Man schätzt, dass Hefezellen etwa 50 Millionen Proteine enthalten, menschliche Zellen etwa 1 bis 3 Milliarden. Die Konzentration der einzelnen Proteinkopien reicht von einigen wenigen Molekülen pro Zelle bis zu 20 Millionen. In den meisten Zellen werden nicht alle Gene, die für Proteine kodieren, exprimiert, und ihre Anzahl hängt beispielsweise vom Zelltyp und von äußeren Reizen ab. So werden beispielsweise von den etwa 20.000 Proteinen, für die das menschliche Genom kodiert, nur 6.000 in lymphoblastoiden Zellen nachgewiesen. ⓘ
Synthese
Biosynthese

Proteine werden mit Hilfe der in den Genen kodierten Informationen aus Aminosäuren zusammengesetzt. Jedes Protein hat seine eigene, einzigartige Aminosäuresequenz, die durch die Nukleotidsequenz des Gens, das dieses Protein kodiert, festgelegt ist. Der genetische Code besteht aus einer Reihe von Drei-Nukleotid-Sätzen, die Codons genannt werden, und jede Drei-Nukleotid-Kombination bezeichnet eine Aminosäure, z. B. AUG (Adenin-Uracil-Guanin) ist der Code für Methionin. Da die DNA vier Nukleotide enthält, beträgt die Gesamtzahl der möglichen Codons 64; daher gibt es eine gewisse Redundanz im genetischen Code, wobei einige Aminosäuren durch mehr als ein Codon angegeben werden. Die in der DNA kodierten Gene werden zunächst von Proteinen wie der RNA-Polymerase in Prä-Messenger-RNA (mRNA) umgeschrieben. In den meisten Organismen wird die prä-mRNA (auch als primäres Transkript bezeichnet) dann durch verschiedene Formen der posttranskriptionellen Modifikation zur reifen mRNA verarbeitet, die dann als Vorlage für die Proteinsynthese durch das Ribosom verwendet wird. In Prokaryonten kann die mRNA entweder sofort nach ihrer Herstellung verwendet werden oder von einem Ribosom gebunden werden, nachdem sie sich vom Nukleoid entfernt hat. Im Gegensatz dazu wird bei Eukaryonten die mRNA im Zellkern gebildet und dann durch die Kernmembran in das Zytoplasma transportiert, wo die Proteinsynthese stattfindet. Die Geschwindigkeit der Proteinsynthese ist bei Prokaryonten höher als bei Eukaryonten und kann bis zu 20 Aminosäuren pro Sekunde erreichen. ⓘ
Der Prozess der Synthese eines Proteins aus einer mRNA-Vorlage wird als Translation bezeichnet. Die mRNA wird auf das Ribosom geladen und drei Nukleotide auf einmal gelesen, indem jedes Codon mit seinem basenpaarenden Anticodon auf einem Transfer-RNA-Molekül abgeglichen wird, das die dem erkannten Codon entsprechende Aminosäure trägt. Das Enzym Aminoacyl-tRNA-Synthetase "belädt" die tRNA-Moleküle mit den richtigen Aminosäuren. Das entstehende Polypeptid wird oft als naszierende Kette bezeichnet. Proteine werden immer vom N-Terminus zum C-Terminus biosynthetisiert. ⓘ
Die Größe eines synthetisierten Proteins kann anhand der Anzahl der enthaltenen Aminosäuren und seiner Gesamtmolekülmasse gemessen werden, die normalerweise in Dalton-Einheiten (gleichbedeutend mit atomaren Masseneinheiten) oder der abgeleiteten Einheit Kilodalton (kDa) angegeben wird. Die durchschnittliche Größe eines Proteins nimmt von Archaea über Bacteria bis hin zu Eukaryote zu (283, 311, 438 Reste bzw. 31, 34, 49 kDa), da die Proteine in höheren Organismen aus einer größeren Anzahl von Proteindomänen bestehen. Hefeproteine zum Beispiel sind im Durchschnitt 466 Aminosäuren lang und haben eine Masse von 53 kDa. Die größten bekannten Proteine sind die Titine, ein Bestandteil des Muskelsarkomers, mit einer Molekularmasse von fast 3.000 kDa und einer Gesamtlänge von fast 27.000 Aminosäuren. ⓘ
Chemische Synthese
Kurze Proteine können auch chemisch synthetisiert werden, und zwar mit einer Reihe von Verfahren, die als Peptidsynthese bekannt sind. Diese beruhen auf organischen Synthesetechniken wie der chemischen Ligation, um Peptide in hoher Ausbeute herzustellen. Die chemische Synthese ermöglicht das Einfügen von nicht natürlichen Aminosäuren in Polypeptidketten, z. B. das Anhängen von Fluoreszenzsonden an Aminosäureseitenketten. Diese Methoden sind in der Laborbiochemie und Zellbiologie nützlich, jedoch im Allgemeinen nicht für kommerzielle Anwendungen. Die chemische Synthese ist für Polypeptide, die länger als etwa 300 Aminosäuren sind, ineffizient, und die synthetisierten Proteine können nicht ohne weiteres ihre native Tertiärstruktur annehmen. Die meisten chemischen Synthesemethoden verlaufen vom C-Terminus zum N-Terminus, entgegengesetzt zur biologischen Reaktion. ⓘ
Struktur


Die meisten Proteine falten sich zu einzigartigen 3D-Strukturen. Die Form, in die sich ein Protein von Natur aus faltet, wird als seine native Konformation bezeichnet. Viele Proteine können sich allein aufgrund der chemischen Eigenschaften ihrer Aminosäuren falten, andere wiederum benötigen die Hilfe von molekularen Chaperonen, um sich in ihre native Form zu falten. Biochemiker sprechen oft von vier verschiedenen Aspekten der Struktur eines Proteins:
- Primärstruktur: die Aminosäuresequenz. Ein Protein ist ein Polyamid.
- Sekundärstruktur: sich regelmäßig wiederholende lokale Strukturen, die durch Wasserstoffbrücken stabilisiert werden. Die häufigsten Beispiele sind die α-Helix, das β-Faltblatt und die Windungen. Da Sekundärstrukturen lokal sind, können in ein und demselben Proteinmolekül viele Regionen mit unterschiedlichen Sekundärstrukturen vorhanden sein.
- Tertiärstruktur: die Gesamtform eines einzelnen Proteinmoleküls; die räumliche Beziehung der Sekundärstrukturen zueinander. Die Tertiärstruktur wird im Allgemeinen durch nichtlokale Wechselwirkungen stabilisiert, am häufigsten durch die Bildung eines hydrophoben Kerns, aber auch durch Salzbrücken, Wasserstoffbrücken, Disulfidbrücken und sogar durch posttranslationale Modifikationen. Der Begriff "Tertiärstruktur" wird oft als Synonym für den Begriff "Faltung" verwendet. Die Tertiärstruktur steuert die Grundfunktion des Proteins.
- Quartärstruktur: die Struktur, die von mehreren Proteinmolekülen (Polypeptidketten) gebildet wird, die in diesem Zusammenhang gewöhnlich als Proteinuntereinheiten bezeichnet werden und als ein einziger Proteinkomplex funktionieren.
- Quinäre Struktur: die Signaturen der Proteinoberfläche, die das überfüllte Zellinnere organisieren. Die quinäre Struktur hängt von den flüchtigen, aber wesentlichen makromolekularen Wechselwirkungen ab, die in lebenden Zellen auftreten. ⓘ
Proteine sind keine völlig starren Moleküle. Zusätzlich zu diesen Strukturebenen können Proteine zwischen mehreren verwandten Strukturen wechseln, während sie ihre Funktionen erfüllen. Im Zusammenhang mit diesen funktionellen Umlagerungen werden diese tertiären oder quaternären Strukturen gewöhnlich als "Konformationen" bezeichnet, und die Übergänge zwischen ihnen werden als Konformationsänderungen bezeichnet. Solche Veränderungen werden häufig durch die Bindung eines Substratmoleküls an die aktive Stelle eines Enzyms oder den physikalischen Bereich des Proteins, der an der chemischen Katalyse beteiligt ist, ausgelöst. In Lösung verändern Proteine ihre Struktur auch durch thermische Schwingungen und durch Zusammenstöße mit anderen Molekülen. ⓘ

Proteine können informell in drei Hauptklassen eingeteilt werden, die mit typischen Tertiärstrukturen korrelieren: globuläre Proteine, faserige Proteine und Membranproteine. Fast alle globulären Proteine sind löslich und viele sind Enzyme. Faserproteine sind oft strukturell, wie z. B. Kollagen, der Hauptbestandteil des Bindegewebes, oder Keratin, der Proteinbestandteil von Haaren und Nägeln. Membranproteine dienen oft als Rezeptoren oder stellen Kanäle für polare oder geladene Moleküle bereit, die die Zellmembran passieren können. ⓘ
Ein Sonderfall intramolekularer Wasserstoffbrückenbindungen innerhalb von Proteinen, die schlecht gegen den Angriff von Wasser abgeschirmt sind und daher ihre eigene Dehydratisierung fördern, werden Dehydronen genannt. ⓘ
Proteindomänen
Viele Proteine setzen sich aus mehreren Proteindomänen zusammen, d. h. aus Abschnitten eines Proteins, die sich zu unterschiedlichen Struktureinheiten falten. Domänen haben in der Regel auch spezifische Funktionen, wie enzymatische Aktivitäten (z. B. Kinase), oder sie dienen als Bindungsmodule (z. B. bindet die SH3-Domäne an prolinreiche Sequenzen in anderen Proteinen). ⓘ
Sequenz-Motiv
Kurze Aminosäuresequenzen in Proteinen dienen oft als Erkennungsstellen für andere Proteine. So binden SH3-Domänen typischerweise an kurze PxxP-Motive (d. h. 2 Prolinen [P], die durch zwei nicht spezifizierte Aminosäuren [x] getrennt sind, wobei die umgebenden Aminosäuren die genaue Bindungsspezifität bestimmen können). Viele solcher Motive wurden in der Datenbank Eukaryotic Linear Motif (ELM) gesammelt. ⓘ
Zelluläre Funktionen
Proteine sind die Hauptakteure in der Zelle und erfüllen die Aufgaben, die durch die in den Genen kodierten Informationen festgelegt sind. Mit Ausnahme bestimmter Arten von RNA sind die meisten anderen biologischen Moleküle relativ träge Elemente, auf die Proteine einwirken. Proteine machen die Hälfte des Trockengewichts einer Escherichia coli-Zelle aus, während andere Makromoleküle wie DNA und RNA nur 3 % bzw. 20 % ausmachen. Die Gesamtheit der Proteine, die in einer bestimmten Zelle oder einem bestimmten Zelltyp exprimiert werden, wird als ihr Proteom bezeichnet. ⓘ
Die wichtigste Eigenschaft von Proteinen, die auch ihre vielfältigen Funktionen ermöglicht, ist ihre Fähigkeit, andere Moleküle spezifisch und fest zu binden. Der Bereich des Proteins, der für die Bindung eines anderen Moleküls verantwortlich ist, wird als Bindungsstelle bezeichnet und ist häufig eine Vertiefung oder "Tasche" auf der Moleküloberfläche. Diese Bindungsfähigkeit wird durch die Tertiärstruktur des Proteins, die die Tasche der Bindungsstelle definiert, und durch die chemischen Eigenschaften der Seitenketten der umgebenden Aminosäuren vermittelt. Die Proteinbindung kann außerordentlich eng und spezifisch sein; so bindet beispielsweise das Ribonuklease-Inhibitorprotein an das menschliche Angiogenin mit einer subfemtomolaren Dissoziationskonstante (<10-15 M), aber überhaupt nicht an sein amphibisches Homolog Onconase (>1 M). Äußerst geringfügige chemische Veränderungen wie das Hinzufügen einer einzigen Methylgruppe zu einem Bindungspartner können manchmal ausreichen, um die Bindung nahezu auszuschließen; so diskriminiert beispielsweise die für die Aminosäure Valin spezifische Aminoacyl-tRNA-Synthetase die sehr ähnliche Seitenkette der Aminosäure Isoleucin. ⓘ
Proteine können sowohl an andere Proteine als auch an niedermolekulare Substrate binden. Wenn Proteine spezifisch an andere Kopien desselben Moleküls binden, können sie oligomerisieren und Fibrillen bilden; dieser Prozess tritt häufig bei Strukturproteinen auf, die aus kugelförmigen Monomeren bestehen, die sich selbst assoziieren und starre Fasern bilden. Protein-Protein-Wechselwirkungen regulieren auch die Aktivität von Enzymen, steuern den Ablauf des Zellzyklus und ermöglichen den Aufbau großer Proteinkomplexe, die viele eng miteinander verbundene Reaktionen mit einer gemeinsamen biologischen Funktion durchführen. Proteine können auch an Zellmembranen binden oder sogar in diese integriert werden. Die Fähigkeit von Bindungspartnern, Konformationsänderungen in Proteinen hervorzurufen, ermöglicht den Aufbau enorm komplexer Signalnetzwerke. Da die Wechselwirkungen zwischen Proteinen reversibel sind und stark von der Verfügbarkeit verschiedener Gruppen von Partnerproteinen abhängen, um Aggregate zu bilden, die in der Lage sind, diskrete Funktionen auszuführen, ist die Untersuchung der Wechselwirkungen zwischen spezifischen Proteinen ein Schlüssel zum Verständnis wichtiger Aspekte der Zellfunktionen und letztlich der Eigenschaften, die bestimmte Zelltypen auszeichnen. ⓘ
Enzyme
Die bekannteste Rolle von Proteinen in der Zelle ist die der Enzyme, die chemische Reaktionen katalysieren. Enzyme sind in der Regel sehr spezifisch und beschleunigen nur eine oder wenige chemische Reaktionen. Enzyme führen die meisten der am Stoffwechsel beteiligten Reaktionen durch und manipulieren die DNA in Prozessen wie DNA-Replikation, DNA-Reparatur und Transkription. Einige Enzyme wirken auf andere Proteine ein, um chemische Gruppen hinzuzufügen oder zu entfernen, ein Prozess, der als posttranslationale Modifikation bekannt ist. Es sind etwa 4.000 Reaktionen bekannt, die von Enzymen katalysiert werden. Die durch die enzymatische Katalyse bewirkte Beschleunigung ist oft enorm - im Fall der Orotat-Decarboxylase beträgt die Beschleunigung das 1017-fache der unkatalysierten Reaktion (78 Millionen Jahre ohne das Enzym, 18 Millisekunden mit dem Enzym). ⓘ
Die Moleküle, die von Enzymen gebunden werden und auf die sie einwirken, werden als Substrate bezeichnet. Obwohl Enzyme aus Hunderten von Aminosäuren bestehen können, ist in der Regel nur ein kleiner Teil der Reste, die mit dem Substrat in Kontakt kommen, und ein noch kleinerer Teil - im Durchschnitt drei bis vier Reste - direkt an der Katalyse beteiligt. Der Bereich des Enzyms, der das Substrat bindet und die katalytischen Reste enthält, wird als aktives Zentrum bezeichnet. ⓘ
Dirigentproteine gehören zu einer Klasse von Proteinen, die die Stereochemie einer von anderen Enzymen synthetisierten Verbindung bestimmen. ⓘ
Zellsignalisierung und Ligandenbindung
Viele Proteine sind am Prozess der Zellsignalisierung und der Signaltransduktion beteiligt. Einige Proteine, wie z. B. Insulin, sind extrazelluläre Proteine, die ein Signal von der Zelle, in der sie synthetisiert wurden, an andere Zellen in entfernten Geweben weiterleiten. Andere sind Membranproteine, die als Rezeptoren fungieren, deren Hauptfunktion darin besteht, ein Signalmolekül zu binden und eine biochemische Reaktion in der Zelle hervorzurufen. Viele Rezeptoren haben eine Bindungsstelle auf der Zelloberfläche und eine Effektordomäne in der Zelle, die enzymatisch aktiv sein oder eine Konformationsänderung erfahren kann, die von anderen Proteinen in der Zelle erkannt wird. ⓘ
Antikörper sind Proteinbestandteile des adaptiven Immunsystems, deren Hauptfunktion darin besteht, Antigene oder körperfremde Substanzen zu binden und sie gezielt zu zerstören. Antikörper können in die extrazelluläre Umgebung sezerniert werden oder in den Membranen spezialisierter B-Zellen, den Plasmazellen, verankert sein. Während Enzyme in ihrer Bindungsaffinität für ihre Substrate durch die Notwendigkeit der Durchführung ihrer Reaktion eingeschränkt sind, gibt es für Antikörper keine derartigen Beschränkungen. Die Bindungsaffinität eines Antikörpers zu seinem Ziel ist außerordentlich hoch. ⓘ
Viele Ligandentransportproteine binden bestimmte kleine Biomoleküle und transportieren sie an andere Stellen im Körper eines mehrzelligen Organismus. Diese Proteine müssen eine hohe Bindungsaffinität haben, wenn ihr Ligand in hohen Konzentrationen vorliegt, müssen aber auch den Liganden freisetzen, wenn er in niedrigen Konzentrationen im Zielgewebe vorhanden ist. Das klassische Beispiel für ein ligandenbindendes Protein ist das Hämoglobin, das in allen Wirbeltieren den Sauerstoff von der Lunge zu anderen Organen und Geweben transportiert und in jedem biologischen Reich enge Homologe hat. Lektine sind zuckerbindende Proteine, die hochspezifisch für ihre Zuckerbestandteile sind. Lektine spielen typischerweise eine Rolle bei biologischen Erkennungsphänomenen, an denen Zellen und Proteine beteiligt sind. Rezeptoren und Hormone sind hochspezifische Bindungsproteine. ⓘ
Transmembranproteine können auch als Ligandentransportproteine dienen, die die Durchlässigkeit der Zellmembran für kleine Moleküle und Ionen verändern. Die Membran selbst hat einen hydrophoben Kern, durch den polare oder geladene Moleküle nicht diffundieren können. Membranproteine enthalten interne Kanäle, durch die solche Moleküle in die Zelle ein- und austreten können. Viele Ionenkanalproteine sind so spezialisiert, dass sie nur für ein bestimmtes Ion selektieren; Kalium- und Natriumkanäle zum Beispiel unterscheiden oft nur für eines der beiden Ionen. ⓘ
Strukturelle Proteine
Strukturproteine verleihen biologischen Komponenten, die ansonsten flüssig sind, Steifigkeit und Festigkeit. Bei den meisten Strukturproteinen handelt es sich um Faserproteine; so sind beispielsweise Kollagen und Elastin wichtige Bestandteile von Bindegewebe wie Knorpel, und Keratin findet sich in harten oder fadenförmigen Strukturen wie Haaren, Nägeln, Federn, Hufen und einigen Tierpanzern. Einige kugelförmige Proteine können auch strukturelle Funktionen erfüllen, z. B. Aktin und Tubulin sind kugelförmig und als Monomere löslich, polymerisieren aber zu langen, steifen Fasern, die das Zytoskelett bilden, das es der Zelle ermöglicht, ihre Form und Größe zu erhalten. ⓘ
Andere Proteine, die strukturelle Funktionen erfüllen, sind Motorproteine wie Myosin, Kinesin und Dynein, die in der Lage sind, mechanische Kräfte zu erzeugen. Diese Proteine sind entscheidend für die zelluläre Motilität von einzelligen Organismen und den Spermien vieler mehrzelliger Organismen, die sich sexuell fortpflanzen. Sie erzeugen auch die Kräfte, die von kontrahierenden Muskeln ausgeübt werden, und spielen eine wesentliche Rolle beim intrazellulären Transport. ⓘ
Entwicklung von Proteinen
Eine Schlüsselfrage der Molekularbiologie ist, wie sich Proteine entwickeln, d. h. wie können Mutationen (oder vielmehr Änderungen der Aminosäuresequenz) zu neuen Strukturen und Funktionen führen? Die meisten Aminosäuren in einem Protein können verändert werden, ohne die Aktivität oder Funktion zu beeinträchtigen, wie zahlreiche homologe Proteine in verschiedenen Arten zeigen (die in speziellen Datenbanken für Proteinfamilien, z. B. PFAM, gesammelt werden). Um dramatische Folgen von Mutationen zu vermeiden, kann ein Gen dupliziert werden, bevor es frei mutieren kann. Dies kann jedoch auch zum vollständigen Verlust der Genfunktion und damit zu Pseudo-Genen führen. In der Regel haben Veränderungen einzelner Aminosäuren nur begrenzte Auswirkungen, obwohl einige von ihnen die Proteinfunktion erheblich verändern können, insbesondere bei Enzymen. Beispielsweise können viele Enzyme ihre Substratspezifität durch eine oder einige wenige Mutationen ändern. Änderungen der Substratspezifität werden durch die Substratpromiskuität erleichtert, d. h. durch die Fähigkeit vieler Enzyme, mehrere Substrate zu binden und zu verarbeiten. Wenn Mutationen auftreten, kann die Spezifität eines Enzyms zunehmen (oder abnehmen) und damit auch seine enzymatische Aktivität. So können sich Bakterien (oder andere Organismen) an verschiedene Nahrungsquellen anpassen, einschließlich unnatürlicher Substrate wie Plastik. ⓘ
Methoden der Untersuchung
Die Aktivitäten und Strukturen von Proteinen können in vitro, in vivo und in silico untersucht werden. In-vitro-Untersuchungen an gereinigten Proteinen in kontrollierten Umgebungen sind nützlich, um zu erfahren, wie ein Protein seine Funktion ausübt: Enzymkinetik-Studien erforschen beispielsweise den chemischen Mechanismus der katalytischen Aktivität eines Enzyms und seine relative Affinität zu verschiedenen möglichen Substratmolekülen. Im Gegensatz dazu können In-vivo-Experimente Informationen über die physiologische Rolle eines Proteins im Kontext einer Zelle oder sogar eines ganzen Organismus liefern. In silico-Studien nutzen computergestützte Methoden zur Untersuchung von Proteinen. ⓘ
Aufreinigung von Proteinen
Zur Durchführung von In-vitro-Analysen muss ein Protein von anderen zellulären Bestandteilen gereinigt werden. Dieser Prozess beginnt in der Regel mit einer Zelllyse, bei der die Zellmembran aufgebrochen und der innere Inhalt in eine Lösung, das so genannte Rohlysat, freigesetzt wird. Das resultierende Gemisch kann durch Ultrazentrifugation gereinigt werden, bei der die verschiedenen zellulären Komponenten in Fraktionen mit löslichen Proteinen, Membranlipiden und -proteinen, Zellorganellen und Nukleinsäuren fraktioniert werden. Die Proteine aus diesem Lysat können durch eine Ausfällung, das so genannte Salting out, konzentriert werden. Anschließend werden verschiedene Arten der Chromatographie eingesetzt, um das oder die interessierenden Proteine auf der Grundlage von Eigenschaften wie Molekulargewicht, Nettoladung und Bindungsaffinität zu isolieren. Der Grad der Reinigung kann durch verschiedene Arten von Gelelektrophorese überwacht werden, wenn das Molekulargewicht und der isoelektrische Punkt des gewünschten Proteins bekannt sind, durch Spektroskopie, wenn das Protein unterscheidbare spektroskopische Merkmale aufweist, oder durch Enzymtests, wenn das Protein enzymatische Aktivität besitzt. Darüber hinaus können Proteine mit Hilfe der Elektrofokussierung nach ihrer Ladung isoliert werden. ⓘ
Bei natürlichen Proteinen kann eine Reihe von Reinigungsschritten erforderlich sein, um ein für Laboranwendungen ausreichend reines Protein zu erhalten. Um diesen Prozess zu vereinfachen, werden häufig gentechnische Verfahren eingesetzt, um Proteine mit chemischen Eigenschaften zu versehen, die ihre Reinigung erleichtern, ohne ihre Struktur oder Aktivität zu beeinträchtigen. Dabei wird ein "Tag", der aus einer spezifischen Aminosäuresequenz besteht, häufig eine Reihe von Histidinresten (ein "His-Tag"), an einen Terminus des Proteins angehängt. Wenn das Lysat über eine Chromatographiesäule mit Nickel geleitet wird, binden die Histidinreste an das Nickel und bleiben an der Säule haften, während die nicht markierten Bestandteile des Lysats ungehindert passieren. Es wurde eine Reihe verschiedener Markierungen entwickelt, die den Forschern helfen, spezifische Proteine aus komplexen Gemischen zu reinigen. ⓘ
Zelluläre Lokalisierung
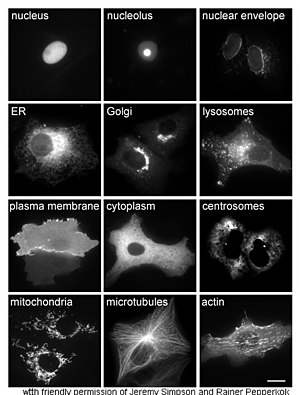
Bei der Untersuchung von Proteinen in vivo geht es häufig um die Synthese und Lokalisierung des Proteins in der Zelle. Obwohl viele intrazelluläre Proteine im Zytoplasma und membrangebundene oder sezernierte Proteine im endoplasmatischen Retikulum synthetisiert werden, ist oft unklar, wie die Proteine auf bestimmte Organellen oder Zellstrukturen ausgerichtet sind. Eine nützliche Technik zur Bewertung der zellulären Lokalisierung nutzt die Gentechnik, um in einer Zelle ein Fusionsprotein oder eine Chimäre zu exprimieren, die aus dem natürlichen Protein von Interesse besteht, das mit einem "Reporter" wie dem grün fluoreszierenden Protein (GFP) verbunden ist. Die Position des fusionierten Proteins in der Zelle kann mit Hilfe der Mikroskopie sauber und effizient sichtbar gemacht werden, wie in der nebenstehenden Abbildung gezeigt. ⓘ
Andere Methoden zur Klärung der zellulären Position von Proteinen erfordern die Verwendung bekannter Kompartimentmarker für Regionen wie das ER, die Golgi, Lysosomen oder Vakuolen, Mitochondrien, Chloroplasten, Plasmamembran usw. Durch die Verwendung von fluoreszenzmarkierten Versionen dieser Marker oder von Antikörpern gegen bekannte Marker wird es viel einfacher, die Lokalisierung eines Proteins von Interesse zu bestimmen. Die indirekte Immunfluoreszenz ermöglicht zum Beispiel die Fluoreszenz-Kolokalisierung und den Nachweis der Lokalisierung. Fluoreszenzfarbstoffe werden zur Markierung zellulärer Kompartimente zu einem ähnlichen Zweck verwendet. ⓘ
Es gibt auch noch andere Möglichkeiten. So wird bei der Immunhistochemie in der Regel ein Antikörper gegen ein oder mehrere interessierende Proteine verwendet, die mit Enzymen konjugiert sind, die entweder lumineszierende oder chromogene Signale erzeugen, die zwischen den Proben verglichen werden können und so Informationen über die Lokalisierung liefern. Eine weitere anwendbare Technik ist die Kofraktionierung in Saccharose- (oder anderen Material-) Gradienten durch isopyknische Zentrifugation. Diese Technik beweist zwar nicht die Kolokalisierung eines Kompartiments mit bekannter Dichte und des interessierenden Proteins, erhöht aber die Wahrscheinlichkeit und eignet sich besser für groß angelegte Studien. ⓘ
Die Goldstandard-Methode für die zelluläre Lokalisierung ist schließlich die Immun-Elektronenmikroskopie. Auch bei dieser Technik wird ein Antikörper gegen das betreffende Protein zusammen mit klassischen elektronenmikroskopischen Techniken verwendet. Die Probe wird für eine normale elektronenmikroskopische Untersuchung vorbereitet und dann mit einem Antikörper gegen das interessierende Protein behandelt, der an ein extrem elektrodichtes Material, in der Regel Gold, konjugiert ist. Auf diese Weise lassen sich sowohl ultrastrukturelle Details als auch das betreffende Protein lokalisieren. ⓘ
Durch eine andere gentechnische Anwendung, die so genannte ortsgerichtete Mutagenese, können die Forscher die Proteinsequenz und damit seine Struktur, seine zelluläre Lokalisierung und seine Anfälligkeit für Regulierung verändern. Diese Technik ermöglicht sogar den Einbau unnatürlicher Aminosäuren in Proteine unter Verwendung modifizierter tRNAs und kann das rationale Design neuer Proteine mit neuartigen Eigenschaften ermöglichen. ⓘ
Proteomik
Die Gesamtheit der Proteine, die zu einem bestimmten Zeitpunkt in einer Zelle oder einem Zelltyp vorhanden sind, wird als Proteom bezeichnet, und die Untersuchung solcher umfangreichen Datensätze definiert den Bereich der Proteomik, der in Analogie zum verwandten Bereich der Genomik benannt wurde. Zu den wichtigsten experimentellen Techniken in der Proteomik gehören die 2D-Elektrophorese, die die Auftrennung vieler Proteine ermöglicht, die Massenspektrometrie, die eine schnelle Identifizierung von Proteinen im Hochdurchsatz und die Sequenzierung von Peptiden (meist nach einem In-Gel-Verdau) erlaubt, Protein-Mikroarrays, die den Nachweis der relativen Mengen der verschiedenen in einer Zelle vorhandenen Proteine ermöglichen, und das Two-Hybrid-Screening, das die systematische Erforschung von Protein-Protein-Wechselwirkungen erlaubt. Die Gesamtheit der biologisch möglichen Wechselwirkungen wird als Interaktom bezeichnet. Ein systematischer Versuch, die Strukturen von Proteinen zu bestimmen, die alle möglichen Falten repräsentieren, wird als strukturelle Genomik bezeichnet. ⓘ
Bestimmung der Struktur
Die Entdeckung der Tertiärstruktur eines Proteins oder der Quartärstruktur seiner Komplexe kann wichtige Hinweise darauf liefern, wie das Protein seine Funktion ausübt und wie sie beeinflusst werden kann, z. B. bei der Entwicklung von Arzneimitteln. Da Proteine zu klein sind, um sie unter dem Lichtmikroskop zu sehen, müssen andere Methoden zur Bestimmung ihrer Struktur eingesetzt werden. Zu den gängigen experimentellen Methoden gehören die Röntgenkristallografie und die NMR-Spektroskopie, die beide Strukturinformationen mit atomarer Auflösung liefern können. NMR-Experimente sind jedoch in der Lage, Informationen zu liefern, anhand derer eine Teilmenge der Abstände zwischen Atompaaren geschätzt werden kann, und die endgültigen möglichen Konformationen eines Proteins werden durch Lösung eines Abstandsgeometrieproblems bestimmt. Die Doppelpolarisationsinterferometrie ist eine quantitative Analysemethode zur Messung der gesamten Proteinkonformation und der Konformationsänderungen aufgrund von Wechselwirkungen oder anderen Reizen. Zirkulardichroismus ist eine weitere Labortechnik zur Bestimmung der internen β-Faltblatt- / α-Helix-Zusammensetzung von Proteinen. Die Kryoelektronenmikroskopie wird eingesetzt, um Strukturinformationen mit geringerer Auflösung über sehr große Proteinkomplexe, einschließlich zusammengesetzter Viren, zu erhalten; eine als Elektronenkristallographie bekannte Variante kann in einigen Fällen auch hochauflösende Informationen liefern, insbesondere für zweidimensionale Kristalle von Membranproteinen. Gelöste Strukturen werden in der Regel in der Protein Data Bank (PDB) hinterlegt, einer frei zugänglichen Ressource, aus der Strukturdaten über Tausende von Proteinen in Form von kartesischen Koordinaten für jedes Atom des Proteins abgerufen werden können. ⓘ
Es sind viel mehr Gensequenzen als Proteinstrukturen bekannt. Außerdem ist die Menge der gelösten Strukturen auf Proteine ausgerichtet, die sich leicht den Bedingungen der Röntgenkristallographie, einer der wichtigsten Methoden zur Strukturbestimmung, unterziehen lassen. Insbesondere kugelförmige Proteine sind vergleichsweise einfach zu kristallisieren, um sie für die Röntgenkristallographie vorzubereiten. Membranproteine und große Proteinkomplexe lassen sich dagegen nur schwer kristallisieren und sind in der PDB unterrepräsentiert. Strukturelle Genomik-Initiativen haben versucht, diese Mängel zu beheben, indem sie systematisch repräsentative Strukturen der wichtigsten Faltungsklassen gelöst haben. Mit Methoden zur Vorhersage von Proteinstrukturen wird versucht, eine plausible Struktur für Proteine zu generieren, deren Strukturen noch nicht experimentell bestimmt wurden. ⓘ

Zum besseren Verständnis von Aufbau und Funktion ist es unerlässlich, die räumliche Gestalt von Proteinen mithilfe geeigneter Grafikprogramme darzustellen. ⓘ
Das meistverbreitete Dateiformat für Atompositionsdaten von Proteinen ist das PDB-Format der freizugänglichen Protein Data Bank. Eine PDB-Datei enthält zeilenweise Einträge für jedes Atom im Protein, sortiert nach der Aminosäuresequenz; im einfachsten Fall sind das Atomart und kartesische Koordinaten. Es handelt sich also um ein systemunabhängiges Klartext-Format. Auf Basis dieser Datei kann dann z. B. in Jmol die 3D-Struktur dargestellt werden. Ist die natürliche 3D-Struktur noch nicht ermittelt, hilft nur noch die Proteinstrukturvorhersage. ⓘ
Strukturvorhersage

Ergänzend zum Bereich der strukturellen Genomik entwickelt die Proteinstrukturvorhersage effiziente mathematische Modelle von Proteinen, um die molekularen Formationen in der Theorie rechnerisch vorherzusagen, anstatt die Strukturen durch Laborbeobachtung zu ermitteln. Die erfolgreichste Art der Strukturvorhersage, die so genannte Homologiemodellierung, beruht auf dem Vorhandensein einer "Vorlage"-Struktur mit Sequenzähnlichkeit zu dem zu modellierenden Protein; das Ziel der strukturellen Genomik besteht darin, eine ausreichende Repräsentation in gelösten Strukturen bereitzustellen, um die meisten der verbleibenden Strukturen zu modellieren. Obwohl die Erstellung genauer Modelle eine Herausforderung bleibt, wenn nur entfernt verwandte Vorlagestrukturen zur Verfügung stehen, wurde behauptet, dass die Sequenzausrichtung den Engpass in diesem Prozess darstellt, da recht genaue Modelle erstellt werden können, wenn eine "perfekte" Sequenzausrichtung bekannt ist. Viele Methoden zur Strukturvorhersage haben dem aufkommenden Bereich des Protein-Engineering gedient, in dem bereits neuartige Proteinfalten entworfen wurden. Auch Proteine (in Eukaryoten ~33 %) enthalten große unstrukturierte, aber biologisch funktionelle Abschnitte und können als intrinsisch ungeordnete Proteine klassifiziert werden. Die Vorhersage und Analyse der Unordnung von Proteinen ist daher ein wichtiger Bestandteil der Charakterisierung von Proteinstrukturen. ⓘ
Bioinformatik
Für die Analyse der Struktur, Funktion und Entwicklung von Proteinen wurde eine Vielzahl von Berechnungsmethoden entwickelt. Die Entwicklung solcher Werkzeuge wurde durch die große Menge an Genom- und Proteomdaten vorangetrieben, die für eine Vielzahl von Organismen, darunter auch das menschliche Genom, zur Verfügung stehen. Es ist schlichtweg unmöglich, alle Proteine experimentell zu untersuchen, daher werden nur einige wenige in Laborexperimenten untersucht, während computergestützte Werkzeuge zur Extrapolation auf ähnliche Proteine verwendet werden. Solche homologen Proteine können in weit verwandten Organismen durch Sequenzabgleich effizient identifiziert werden. Genom- und Gensequenzen können mit einer Vielzahl von Werkzeugen auf bestimmte Eigenschaften hin durchsucht werden. Sequenzprofilierungswerkzeuge können Restriktionsenzymstellen und offene Leserahmen in Nukleotidsequenzen aufspüren und Sekundärstrukturen vorhersagen. Mit spezieller Software wie ClustalW lassen sich phylogenetische Bäume erstellen und evolutionäre Hypothesen über die Abstammung moderner Organismen und der von ihnen exprimierten Gene entwickeln. Der Bereich der Bioinformatik ist heute für die Analyse von Genen und Proteinen unverzichtbar. ⓘ
In silico-Simulation dynamischer Prozesse
Ein komplexeres Rechenproblem ist die Vorhersage von intermolekularen Wechselwirkungen, wie z. B. beim molekularen Docking, der Proteinfaltung, der Protein-Protein-Wechselwirkung und der chemischen Reaktivität. Mathematische Modelle zur Simulation dieser dynamischen Prozesse beruhen auf der Molekularmechanik, insbesondere auf der Molekulardynamik. In diesem Zusammenhang haben In-silico-Simulationen die Faltung kleiner α-helicaler Proteindomänen wie des Villin-Kopfstücks und des HIV-Accessory-Proteins entdeckt, und hybride Methoden, die Standard-Molekulardynamik mit quantenmechanischer Mathematik kombinieren, haben die elektronischen Zustände von Rhodopsinen untersucht. ⓘ
Über die klassische Molekulardynamik hinaus ermöglichen quantendynamische Methoden die Simulation von Proteinen im atomistischen Detail mit einer genauen Beschreibung der quantenmechanischen Effekte. Beispiele hierfür sind die Multi-Layer-Multikonfigurations-Zeitabhängige Hartree-Methode (MCTDH) und der Ansatz der hierarchischen Bewegungsgleichungen (HEOM), die auf pflanzliche Cryptochrome bzw. bakterielle Lichtsammelkomplexe angewendet wurden. Sowohl quantenmechanische als auch klassisch-mechanische Simulationen von Systemen im biologischen Maßstab sind extrem rechenintensiv, so dass Initiativen für verteiltes Rechnen (z. B. das Projekt Folding@home) die molekulare Modellierung erleichtern, indem sie Fortschritte bei der parallelen Verarbeitung durch Grafikprozessoren und Monte-Carlo-Techniken nutzen. ⓘ
Chemische Analyse
Der Gesamtstickstoffgehalt der organischen Materie wird hauptsächlich durch die Aminogruppen in Proteinen gebildet. Der Gesamtstickstoffgehalt nach Kjeldahl (TKN) ist ein Maß für Stickstoff, das bei der Analyse von (Ab-)Wasser, Boden, Lebensmitteln, Futtermitteln und organischen Stoffen im Allgemeinen weit verbreitet ist. Wie der Name schon sagt, wird die Kjeldahl-Methode angewandt. Es gibt auch empfindlichere Methoden. ⓘ
Ernährung
Die meisten Mikroorganismen und Pflanzen können alle 20 Standardaminosäuren biosynthetisieren, während Tiere (einschließlich des Menschen) einen Teil der Aminosäuren über die Nahrung aufnehmen müssen. Die Aminosäuren, die ein Organismus nicht selbst synthetisieren kann, werden als essenzielle Aminosäuren bezeichnet. Schlüsselenzyme, die bestimmte Aminosäuren synthetisieren, sind bei Tieren nicht vorhanden, wie z. B. Aspartokinase, die den ersten Schritt der Synthese von Lysin, Methionin und Threonin aus Aspartat katalysiert. Wenn Aminosäuren in der Umwelt vorhanden sind, können Mikroorganismen Energie sparen, indem sie die Aminosäuren aus ihrer Umgebung aufnehmen und ihre Biosynthesewege herunterregeln. ⓘ
Bei Tieren werden die Aminosäuren durch den Verzehr von eiweißhaltigen Nahrungsmitteln aufgenommen. Die aufgenommenen Proteine werden dann durch Verdauung in Aminosäuren aufgespalten, was in der Regel eine Denaturierung des Proteins durch Einwirkung von Säure und eine Hydrolyse durch Enzyme, die Proteasen, beinhaltet. Einige der aufgenommenen Aminosäuren werden für die Proteinbiosynthese verwendet, während andere durch Glukoneogenese in Glukose umgewandelt oder dem Zitronensäurezyklus zugeführt werden. Diese Verwendung von Eiweiß als Brennstoff ist besonders unter Hungerbedingungen wichtig, da sie es dem Körper ermöglicht, körpereigene Eiweiße zur Lebenserhaltung zu nutzen, insbesondere solche, die in den Muskeln vorkommen. ⓘ
Bei Tieren wie Hunden und Katzen erhält Eiweiß die Gesundheit und Qualität der Haut, indem es das Wachstum der Haarfollikel und die Keratinisierung fördert und so die Wahrscheinlichkeit von Hautproblemen, die zu unangenehmen Gerüchen führen, verringert. Qualitativ minderwertige Proteine spielen auch eine Rolle für die Magen-Darm-Gesundheit, da sie bei Hunden das Potenzial für Blähungen und Geruchsstoffe erhöhen, denn wenn Proteine unverdaut in den Dickdarm gelangen, werden sie fermentiert, wobei Schwefelwasserstoffgas, Indol und Skatol entstehen. Hunde und Katzen verdauen tierische Proteine besser als pflanzliche, aber Produkte minderwertiger tierischer Herkunft werden schlecht verdaut, einschließlich Haut, Federn und Bindegewebe. ⓘ
Proteinstruktur
Klassifizierung von Proteinen
Man kann Proteine nach der äußeren Form in zwei Hauptgruppen einteilen:
- die globulären Proteine, deren Tertiär- oder Quartärstruktur annähernd kugel- oder birnenförmig aussieht und die meist in Wasser oder Salzlösungen gut löslich sind (beispielsweise das Protein des Eiklars, Ov-Albumin genannt),
- die fibrillären Proteine, die eine fadenförmige oder faserige Struktur besitzen, meist unlöslich sind und zu den Stütz- und Gerüstsubstanzen gehören (beispielsweise die Keratine in den Haaren und Fingernägeln, Kollagen, Aktin und Myosin für die Muskelkontraktion).
Weiterhin werden Proteine nach ihrer Zusammensetzung eingeteilt, z. B. bei konjugierten Proteinen. Daneben ist auch eine Einteilung nach der Funktion möglich, z. B. Strukturproteine. ⓘ
| Molekülform | Nicht-Protein-Anteil ⓘ | |
|---|---|---|
| Globuläre Proteine | Fibrilläre Proteine | |
| Albumine | Kollagen | Glykoproteine |
| Globuline | Elastin | Nukleoproteine |
| Histone | Keratin | Chromoproteine |
| Protamine | Fibrinogen | Phosphoproteine |
| Prolamine | Myosin | Lipoproteine |
Proteinoberfläche


Vereinfachend wird stellvertretend für die komplexe Proteinstruktur oft nur das Rückgrat (Backbone) des Proteins abgebildet (z. B. Abbildungen rechts oben). Zum Verständnis der Funktion ist jedoch die Oberfläche des Proteins von großer Bedeutung. Da die Seitenketten der Aminosäuren vom Rückgrat aus in den Raum ragen, tragen auch sie entscheidend zur Struktur bei: Der Verlauf des Rückgrats bestimmt den generellen dreidimensionalen Aufbau, aber die Konturen der Oberfläche und die biochemischen Eigenschaften des Proteins werden von den Seitenketten bestimmt. ⓘ
Proteinchemie
Reinigung
Die Aufreinigung und Anreicherung von Proteinen aus biologischem Material ist ein wichtiger Schritt in der biochemischen Identifikation und Charakterisierung von neu entdeckten Proteinen. ⓘ
In der Biotechnologie und dort besonders bei rekombinanten Proteinen ist die reproduzierbare, sorgfältige Proteinreinigung – meist in großem Maßstab – eine wichtige Voraussetzung zur Verwendung dieser Proteine in der Diagnostik oder Therapie. ⓘ
Quantitativer Nachweis
Folgende Nachweise, die keine Absolutmessungen darstellen und alle ihre Limitationen haben (z. B. Fehlmessungen durch interferierende Substanzen; Bezug auf ein bestimmtes Standardprotein etc.), dienen zur Quantifizierung von Proteinen:
- UV-Absorption
- Xanthoproteinreaktion
- Millon-Reaktion
- Ninhydrinreaktion
- Biuretreaktion
- Bradford-Test
- Lowry-Test
- BCA-Test
- Folin-Reagenz
- Aminosäureanalyse ⓘ
Identifikation
Der Nachweis der Identität eines Proteins kann über vielfältige Methoden erfolgen. Auch kann ein indirekter Nachweis über andere Eigenschaften als die Primärstruktur erfolgen, welche jedoch aus ihr folgen, z. B. über die Anwesenheit seiner Funktion (Enzymkinetik) im Probengefäß oder über immunologische Eigenschaften, die unter anderem in einem Western Blot verwendet werden. ⓘ
Denaturierung
Sowohl durch chemische Einflüsse, wie zum Beispiel Säuren, Salze oder organische Lösungsmittel, als auch durch physikalische Einwirkungen, wie hohe oder tiefe Temperaturen oder auch Druck, können sich die Sekundär- und Tertiärstruktur und damit auch die Quartärstruktur von Proteinen ändern, ohne dass sich die Reihenfolge der Aminosäuren (Primärstruktur) ändert. Dieser Vorgang heißt Denaturierung und ist in der Regel nicht umkehrbar, das heißt, der ursprüngliche dreidimensionale räumliche Aufbau kann ohne Hilfe nicht wiederhergestellt werden. Bekanntestes Beispiel dafür ist das Eiklar im Hühnerei, das beim Kochen fest wird, weil sich der räumliche Aufbau der Proteinmoleküle geändert hat. Der ursprüngliche flüssige Zustand kann nicht mehr hergestellt werden. ⓘ
Das Wiederherstellen des ursprünglichen Zustandes des denaturierten Proteins heißt Renaturieren. ⓘ

Mit der Kontrolle über das Feuer wurde Menschen auch das Kochen möglich, womit nicht zuletzt Speisen bekömmlicher und leicht verdaulich zubereitet werden können. Durch Denaturierung beim Erhitzen ändern sich die physikalischen und physiologischen Eigenschaften von Proteinen, etwa beim Spiegelei, das durch die Hitze in der Pfanne verändert wird. Auch sehr hohes Fieber kann oberhalb einer gewissen Temperatur körpereigene Proteine denaturieren. Diese Proteine können ihre Aufgaben im Organismus dann nicht mehr erfüllen, was für den Menschen lebensgefährlich werden kann. ⓘ
Einige Proteine der roten Blutkörperchen denaturieren beispielsweise bereits bei 42 °C. Der geregelte Fieberanstieg bleibt daher unterhalb solcher Temperaturen. Die Erhöhung der Körpertemperatur beim Fieber bedeutet eine Beschleunigung der Stoffwechselprozesse (siehe RGT-Regel) und erlaubt damit dem Abwehrsystem raschere Reaktionen. Dieses Fieber wird vom Körper selber erzeugt (siehe Pyrogen), um sich besser gegen eingedrungene Erreger oder Fremdkörper (siehe auch Antigen) wehren zu können. Viele der körperfremden Proteine denaturieren schon bei geringeren Temperaturen als die körpereigenen. ⓘ
Hydrolyse und Oxidation
Die durch hydrolytische Spaltung der Proteinketten (Proteolyse) entstehenden Teilstücke sind ein Gemisch aus Peptiden und Aminosäuren; sind diese unter katalytischer Wirkung von Pepsin entstanden, nennt man sie Pepton, im Falle von Trypsin Trypton. ⓘ
Durch reaktive Sauerstoffspezies können Proteine oxidiert werden. Dieser Vorgang heißt Proteinoxidation und spielt bei der Alterung und einer Reihe von pathologischen Prozessen eine wichtige Rolle. Die Oxidation kann einen weitgehenden Funktionsverlust bedeuten und zur Ansammlung von degenerierten Proteinen in der Zelle führen. ⓘ
Protein in der Nahrung
Proteinbedarf
Proteine erfüllen im menschlichen Körper zahlreiche Aufgaben und sind für alle Organfunktionen von Bedeutung, insbesondere bei der Heilung von Wunden und Krankheiten. Zum Aufbau, zum Erhalt und zur Erneuerung der Körperzellen brauchen Menschen eine Nahrung, die Protein enthält. Bezogen auf das Körpergewicht (KG) ist der Bedarf in den Wachstumsphasen zu Lebensbeginn am höchsten. ⓘ
Im ersten Lebensmonat sollte der Säugling täglich eine Proteinmenge von etwa 2,5 Gramm pro Kilogramm Körpergewicht (g/kg KG) aufnehmen, gegen Ende des ersten Lebensjahrs genügen etwa 1,3 g/kg KG. Ab dem zweiten Lebensjahr empfiehlt die Deutsche Gesellschaft für Ernährung für die tägliche Aufnahme von Protein mit der Nahrung lebensaltersabhängige Referenzwerte zwischen 1,0 und 0,8 g/kg. Bei Kleinkindern (1,0) ist der Bedarf höher als bei älteren Kindern und Jugendlichen (0,9), bei jüngeren Erwachsene (0,8) niedriger als bei über 65 Jahre alten (geschätzt 1,0) – jeweils bezogen auf das Normalgewicht, nicht das tatsächliche Körpergewicht. Übergewichtige brauchen nicht mehr als normalgewichtige Menschen. Dagegen ist bei schwangeren Frauen der Bedarf um circa 20 % erhöht (1,0), bei stillenden liegt er noch höher (1,2). Es gibt jedoch eine Übersichtsarbeit von 2010, die auch in einem Bericht einer Expertenkommission der Ernährungs- und Landwirtschaftsorganisation der Vereinten Nationen (2013) Beachtung fand, welche den Tagesproteinbedarf für durchschnittliche Erwachsene mit (0,91–1,2) deutlich höher einschätzt. Bei körperlicher Aktivität steigt der Bedarf an Protein laut DGE nicht an. Eine Nierenschädigung aufgrund erhöhten Eiweißkonsums wurde mehrmals in Langzeitstudien widerlegt. ⓘ
Das mit der Nahrung aufgenommene Protein wird in Magen und Darm verdaut, in kleinere Bestandteile zerlegt und in Bausteine aufgespalten. Zellen der Darmschleimhaut resorbieren diese und geben die einzelnen Aminosäuren in die (portale) Blutbahn ab, die zur Leber führt. Der menschliche Organismus kann einige der Aminosäuren nicht selber herstellen, braucht sie aber als Baustein seiner eigenen Proteine. Mit der Nahrung zugeführtes Protein muss also hinreichen, den Bedarf für jede dieser unentbehrlichen (essenziellen) Aminosäuren abzudecken. ⓘ
Proteinmangel
Ein Mangel an Protein kann eine Reihe von Symptomen hervorrufen. Andauernder Eiweißmangel führt zu Marasmus, Kwashiorkor oder zu beidem und letzten Endes zum Tod.
- Die Eiweißmangelkrankheit Kwashiorkor tritt vorwiegend in sogenannten Entwicklungsländern auf und betrifft meist unterernährte Kinder, deren fortgeschrittenes Leiden an einem Hungerbauch zu erkennen ist, hervorgerufen durch Einlagerung von Wasser. Wegen der fehlenden Bluteiweiße wie Albumin kommt es auch in anderen Körperregionen zu Ödemen (Hungerödemen). Weitere Symptome sind u. a.
Zu einem Eiweißmangel kommt es in den Industrieländern allerdings sehr selten und nur bei extrem proteinarmen Ernährungsformen. Die durchschnittliche deutsche Mischkost enthält mit 100 Gramm Eiweiß pro Tag mehr als genug Protein. Obgleich in der Werbung Eiweißpulver als für Breitensportler empfehlenswert angepriesen werden, deckt „unsere übliche Ernährung […] auch den Eiweißbedarf von Sportlern ab“, wie es dazu in einem Bericht des Ministeriums für Ernährung und Ländlichen Raum Baden-Württembergs heißt. ⓘ
Proteinquellen

Sehr proteinhaltige Nahrungsmittel (in alphabetischer Reihenfolge) sind:
- Eier
- Fisch
- Fleisch
- Hülsenfrüchte (Soja und Lupinen ca. 40 %, Bohnen und Erbsen ca. 20 %) sowie andere Pflanzensamen wie zum Beispiel Raps (ca. 40 %)
- Milchprodukte (Käse und Quark)
- Nüsse ⓘ
Eine weitere Proteinquelle ist die Quinoa-Pflanze, welche neben ihrem hohen Eiweiß-Gehalt (etwa 14 g auf 100 g) alle 9 essentiellen Aminosäuren enthält. Ihrer Bedeutung als Nahrungsquelle bewusst, erklärte der ehemalige UN-Generalsekretär Ban Ki-moon das Jahr 2013 zum Jahr der Quinoa, da sie besonders in der Zeit des Klimawandels als wichtige Nahrungsquelle dienen und den Hunger in den Entwicklungsländern bekämpfen soll. ⓘ
Gesundheitliche Aspekte
Stand bislang vor allem die Menge von Proteinen im Vordergrund, so rückt zunehmend die Quelle von Proteinen und deren gesundheitliche Wirkung in den Blick der Forschung. Heute wird aus gesundheitlicher Sicht zum vermehrten Konsum pflanzlicher Proteinquellen geraten. ⓘ
So zeigt eine Studie aus dem Jahr 2016, dass der Konsum von pflanzlichen Proteinquellen zu einer geringeren Mortalität führte, während tierisches Eiweiß zu einer erhöhten Mortalität aufgrund von Herzkreislauferkrankungen führte. ⓘ
Eine Studie aus dem Jahr 2021 bestätigt dieses Ergebnis. In ihr waren pflanzliche Eiweißquellen sowohl mit einer geringeren Gesamtmortalität verbunden als auch mit einer geringeren Mortalität aufgrund von Demenz und Herzkreislauferkrankungen. Tierisches Eiweiß wie verarbeitetes rotes Fleisch und Eier waren hingegen mit einer höheren Gesamtmortalität verbunden. ⓘ
Wertigkeit von Proteinen
Der Protein Digestibility Corrected Amino Acid Score (PDCAAS) wird von der Ernährungs- und Landwirtschaftsorganisation der Vereinten Nationen (FAO/WHO) und der US Food and Drug Administration als „die beste Methode“ zur Bestimmung der Proteinqualität angenommen. Die Kennzahl berücksichtigt sowohl die Aminosäurenzusammensetzung als auch die Verdaulichkeit dieser. Besondere Bedeutung kommt hierbei dem Gehalt an essentiellen Aminosäuren zu. Darüber hinaus gibt es das ältere und mittlerweile überholte Konzept der biologischen Wertigkeit. 2013 hat die FAO außerdem den Digestible Indispensable Amino Acid Score (DIAAS) als Bewertungsmethode zur Bestimmung der Proteinqualität ins Spiel gebracht. Hauptunterschied ist, dass beim DIAAS die echte ileale Verdaulichkeit gemessen wird. Der DIAAS wird grundsätzlich nicht gekürzt und kann auch Werte über 100 % annehmen. Nur bei der Betrachtung der gesamten Proteinzufuhr, unabhängig davon, ob es sich um eine Mischkost oder um ein einzelnes Nahrungsmittel, wie beispielsweise Säuglingsnahrung, handelt, muss der DIAAS auf 100 % gekürzt werden. ⓘ
Insgesamt bleibt eine Bewertung von verschiedenen Proteinquellen schwierig, wenn es um dem Erhalt normaler Körperfunktionen geht. So schreibt die Encyclopedia of Human Nutrition:
„Der metabolische Bedarf für die Erhaltung normaler Körperfunktionen und -zusammensetzung ist ein wenig verstandener Vorgang von Aminosäuren, die in vielen weiteren metabolischen Vorgängen zum Einsatz kommen, welche nichts mit der Proteinsynthese zu tun haben und welche jedenfalls völlig unterschiedlich von Wachstumsvorgängen (bspw. den Aminosäurevorgängen in Gewebe) sind und als solche einen weit geringeren Proteinbedarf haben. Daher ist es sehr schwierig den Unterschied von tierischem und pflanzlichen Proteinquellen zu demonstrieren und dieser ist vermutlich weniger bedeutend für die menschliche Ernährung. Derzeit herrscht beträchtliche Uneinigkeit über die nötige Höhe an essenziellen Aminosäuren in der menschlichen Ernährung und es gibt unterschiedlichste Ansichten über die relative Bedeutung der Proteinqualität in der Ernährungswissenschaft.“
Wirtschaftliche Bedeutung
Die Gesamtmenge der als nachwachsende Rohstoffe in der stofflichen Nutzung verwendeten Proteine wird für Deutschland im Regelfall mit etwa 55.000 t pro Jahr angegeben. Genaue Angaben über die Herkunft dieser Proteine gibt es nicht, es ist jedoch anzunehmen, dass sie zu einem großen Teil tierischer Herkunft sind. ⓘ
Der Großteil pflanzlicher Proteine wird für die Futtermittelindustrie aufgewendet, so die als Nebenprodukte bei der Pflanzenölpressung und -extraktion anfallenden Pressrückstände (z. B. Raps- und Sojakuchen, Extraktionsschrot) und Nebenprodukte der Gewinnung von Stärke aus Getreide. Pflanzen, die zur Hauptnutzung als Proteinpflanzen angebaut werden, wie bspw. Lupine, Eiweißerbse und Ackerbohne, haben nur eine geringe Bedeutung – die Gesamtfläche für den Anbau dieser Pflanzen als nachwachsende Rohstoffe in Deutschland liegt bei etwa 30 ha pro Jahr. Etwa 1.000 t Weizenproteine werden jährlich in der chemischen Industrie eingesetzt. ⓘ
Von zentraler Bedeutung für die chemisch-technische und biotechnologische Industrie sind dagegen tierische Proteine. Dabei spielt vor allem die Gelatine eine zentrale Rolle, die in Europa vor allem aus Rinderspalt, Schweineschwarten sowie Knochen von Rindern und Schweinen gewonnen wird. In Deutschland werden jährlich etwa 32.000 t Gelatine in Speisequalität hergestellt, die europäische Gesamtproduktion beträgt 120.000 t (70 % Schweineschwarten, 18 % Knochen, 10 % Rinderspalt, 2 % Sonstige). Verwendet werden in Deutschland etwa 90.000 t, wobei 2/3 im Ernährungsbereich und von dem Rest etwa die Hälfte für den Futtermittelbereich aufgewendet werden. Etwa 15.000 t werden in der chemischen und pharmazeutischen Industrie verwendet. Dabei finden sich die Haupteinsatzbereiche in der Pharmaindustrie, mit Umhüllungen von Tabletten und Vitaminpräparaten (Hart- und Weichkapseln) sowie Gelatinezäpfchen. Außerdem wird Gelatine für blutstillende Schwämmchen sowie als Blutplasmaersatz eingesetzt. In der analogen Fotografie stellt Gelatine die Basis für die fotoempfindlichen Schichten auf dem Film und dem Fotopapier dar. Auch moderne Druckerpapiere zum Ausdrucken von Farbbildern sind mit Gelatine beschichtet. ⓘ
Neben Gelatine stellt Casein eine wichtige Proteinquelle für die chemische Industrie dar. Das aus Milcheiweiß gewonnene Protein wird vor allem als Beschichtungsmaterial für Glanzpapiere sowie als Zusatz für Streichfarben verwendet (ca. 1–2 % je nach Hersteller). Zudem findet es Verwendung als Etikettenkleber auf Glasflaschen. Jährlich werden in Deutschland etwa 8.000 bis 10.000 t Casein eingesetzt. ⓘ
Die Nutzung von Proteinen aus Blutmehl zur Herstellung von bio-basierten Kunststoffen (z. B. Pflanztöpfe) befindet sich noch in der Entwicklung, ebenso ein Verfahren zur biotechnologischen Herstellung von Fasern aus Seidenproteinen zur Verarbeitung in Schäumen, Vliesstoffen oder Folien. ⓘ
Etwa 6.000 bis 7.000 t Proteine sind Autolyseprodukte aus Hefen (Hefeextrakte). Diese finden Anwendung vor allem in der Pharmazeutischen Industrie und der Nahrungsmittelindustrie sowie in der Biotechnologischen Industrie als Nährlösung für Mikroorganismen. ⓘ
Gewinnung von pflanzlichen Proteinen
Pflanzliche Proteine können aus Soja, Erbsen, Lupinen, Raps oder anderen proteinreichen Früchten wie z. B. Sonnenblumenkernen u. a. m. gewonnen werden. Die geernteten proteinhaltigen Früchte werden mechanisch zerkleinert und entfettet. Es entstehen Flocken oder ein proteinreiches Pulver. Anschließend wird unter Verwendung von Lösungsmitteln ein Proteinkonzentrat gewonnen, das ggf. weiter zu Proteinisolat gereinigt und aufkonzentriert wird: Die Flocken oder Mehl werden mit Wasser versetzt und angemaischt. Die proteinarmen Fasern und Feststoffe werden im nächsten Schritt mit Hilfe von Industriezentrifugen von der proteinreichen Lösung abgetrennt. Dann folgt die sogenannte Ausfällung. Hier wird der pH-Wert der proteinreichen Lösung auf den isoelektrischen Punkt eingestellt. Dadurch setzen sich die Proteinpartikel ab. Diese werden dann wiederum mittels Zentrifugen von der Lauge abgetrennt. Um alle Bestandteile der Mutterlauge aus dem ausgefällten und abgetrennten Protein zu entfernen, wird das Protein erneut mit Wasser versetzt und wieder mit Hilfe der Zentrifugalkraft abgetrennt. ⓘ
Bei einer Trockenextrusion wird unter Zuführung von Wärme, Druck und Hilfsstoffen ein Zwischenprodukt mit niedrigem Wassergehalt erzeugt. Es wird in der Fachsprache als TVP (Texturized Vegetable Protein) bezeichnet und hat eine trockene Konsistenz in Form von Körnern, Streifen oder Flocken. Dies kann durch Zugabe von Wasser zu einem schwammartigen Vorprodukt für Fleischersatzprodukte weiter verarbeitet werden. Bei einer Nassextrusion wird alternativ mit einem höheren Wassergehalt operiert. Die Feuchtigkeit des Zwischenproduktes liegt daher näher am Wassergehalt des Endproduktes. Das Zwischenprodukt wird als HMMA (High Moistured Meat Analogues) bezeichnet. ⓘ
Industrielle Nutzung neuer Proteinquellen
Als vollwertigen Ersatz des proteinreichen Fischmehls durch Protein aus Fliegenlarvenmehl züchtet ein Unternehmen auf dem agrarwissenschaftlichen Gelände der Universität Stellenbosch in Elsenburg (Südafrika) millionenfach Stubenfliegen (Musca domestica) heran. Aus einem Kilogramm Fliegeneier können in nur 72 Stunden ungefähr 380 bis 420 Kilogramm Protein entstehen. ⓘ
Bei entsprechender Großproduktion könnte ein großer Teil der weltweiten Fischmehlproduktion eingespart und die Weltmeere vom industriellen Fischfang entlastet werden. Noch 2012 will das Unternehmen „Agriprotein“ in die Massenherstellung gehen. Täglich werden 65 t Blut aus herkömmlichen Schlachtereien benötigt, um 100 t Fliegenlarven innerhalb eines Zeitraumes von ca. 3 Tagen auf eine Länge von jeweils rund 12 mm heranzuziehen. Im Wege der Trocknung, Vermahlung zu Madenmehl und anschließenden Pelletierung können so täglich 20 t des Proteinprodukts gewonnen werden. Eine weitere Pilotanlage in Deutschland wird von der deutschen Regierung mit 50 % gefördert. Das Unternehmen hat 2012 den People and Environment Achievement Award erhalten. ⓘ
Herstellung und Optimierung rekombinanter Proteine
Die Herstellung rekombinanter Proteine mit genau festgelegter Aminosäuresequenz und möglicherweise weiteren Veränderungen (z. B. eine Glykosylierung) geschieht sowohl im Labor als auch großtechnisch entweder durch Peptidsynthese oder biotechnologisch durch Überexpression in verschiedenen Organismen und folgender Proteinreinigung. ⓘ
Im Zuge des Protein-Engineerings können Eigenschaften des gewünschten Proteins gezielt (über das Proteindesign) oder zufällig (über eine gerichtete Evolution) verändert werden. Prinzipiell können industriell dieselben Verfahren angewandt werden wie im Labor, jedoch ist die Verwendung von Nutzpflanzen durch Pharming am besten für die großtechnische Nutzung geeignet, bei der mit Bioreaktoren in Reinräumen gearbeitet wird. Um die geeigneten Organismen zu erhalten, werden gentechnische Methoden eingesetzt. ⓘ
Die technische Produktion nativer Proteine findet weltweit hauptsächlich in der Pharmazie (Biopharmazeutika) und zur industriellen Verwendung von Enzymen als Waschmittelzusätze (Proteasen, Lipasen, Amylasen und Cellulasen) oder in der Milchverarbeitung (Lactasen) statt. Proteine für die Nahrungsmittelindustrie müssen nicht notwendigerweise in nativer Form hergestellt werden, da eine biologische Aktivität nicht immer erforderlich ist, z. B. bei Käse oder Tofu. ⓘ
Bedeutung für die Archäologie
Insbesondere mit Hilfe verbesserter Methoden der Massenspektrometrie ist es heute möglich, Proteine oder Teile von Proteinen aus archäologischen und paläontologischen Funden zu analysieren. Aus der Rekonstruktion „fossiler“ Proteine kann zudem auf die sie codierenden Gene und damit auf den Bau der aDNA zurückgeschlossen werden. ⓘ
Trivia
„Seidenglanz-Proteine“, die als Zusatz für Haarwaschmittel für Menschen und zur Fellpflege für Tiere beworben werden (um vorgeblich Glanz zu erzeugen), werden aus den Resten von Puppen der Seidenraupen, nach Ablösung der diese umhüllenden Seidenfäden, hergestellt. ⓘ
Im Februar 2020 berichteten Wissenschaftler den Fund eines Protein „Hemolithin“ im Inneren des 1990 gefundenen Meteoriten „Acfer 086“. Proteine sind essentielle Bauteile für die Entstehung von Leben. Falls ihre kontroverse Studie im Peer-Review bestätigt wird, könnte dies die Panspermie-Hypothese stützen. Das Protein könnte in protoplanetarem oder interstellarem Gas entstanden sein. ⓘ