Gen
In der Biologie ist ein Gen (von griechisch: γένος, génos; bedeutet Generation oder Geburt oder Geschlecht) eine Grundeinheit der Vererbung und eine Nukleotidsequenz in der DNA, die für die Synthese eines Genprodukts, entweder RNA oder Protein, kodiert. ⓘ
Bei der Genexpression wird die DNA zunächst in RNA kopiert. Die RNA kann direkt funktionsfähig sein oder als Zwischenvorlage für ein Protein dienen, das eine Funktion ausübt. Die Weitergabe von Genen an die Nachkommen eines Organismus ist die Grundlage für die Vererbung von phänotypischen Merkmalen. Diese Gene setzen sich aus verschiedenen DNA-Sequenzen zusammen, die als Genotypen bezeichnet werden. Die Genotypen bestimmen zusammen mit Umwelt- und Entwicklungsfaktoren, wie die Phänotypen aussehen werden. Die meisten biologischen Merkmale stehen unter dem Einfluss von Polygenen (vielen verschiedenen Genen) sowie von Wechselwirkungen zwischen Genen und Umwelt. Einige genetische Merkmale sind sofort sichtbar, wie z. B. die Augenfarbe oder die Anzahl der Gliedmaßen, andere nicht, wie z. B. die Blutgruppe, das Risiko für bestimmte Krankheiten oder die Tausende von grundlegenden biochemischen Prozessen, die das Leben ausmachen. ⓘ
Gene können Mutationen in ihrer Sequenz aufweisen, die zu verschiedenen Varianten, den so genannten Allelen, in der Bevölkerung führen. Diese Allele kodieren leicht unterschiedliche Versionen eines Proteins, die unterschiedliche phänotypische Merkmale verursachen. Die Verwendung des Begriffs "ein Gen haben" (z. B. "gute Gene", "Haarfarben-Gen") bezieht sich in der Regel auf das Vorhandensein eines anderen Allels desselben, gemeinsam genutzten Gens. Gene entwickeln sich durch natürliche Selektion / Überleben des Stärkeren und genetische Drift der Allele. ⓘ
Das Konzept des Gens wird immer weiter verfeinert, wenn neue Phänomene entdeckt werden. So können beispielsweise die regulatorischen Regionen eines Gens weit von den kodierenden Regionen entfernt sein, und die kodierenden Regionen können in mehrere Exons aufgeteilt sein. Einige Viren speichern ihr Genom in RNA anstelle von DNA, und einige Genprodukte sind funktionelle, nicht codierende RNAs. Daher ist eine weit gefasste, moderne Arbeitsdefinition eines Gens jeder diskrete Locus einer vererbbaren genomischen Sequenz, die die Merkmale eines Organismus beeinflusst, indem sie als funktionelles Produkt oder durch Regulierung der Genexpression exprimiert wird. ⓘ
Der Begriff Gen wurde 1909 von dem dänischen Botaniker, Pflanzenphysiologen und Genetiker Wilhelm Johannsen eingeführt. Er ist vom altgriechischen Wort γόνος, gonos, abgeleitet, das Nachkommenschaft und Fortpflanzung bedeutet. ⓘ

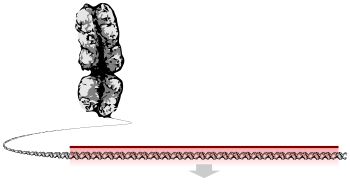
Gezeigt ist ein eukaryotisches Gen, das (hier verkürzt) Introns und Exons enthält, und im Hintergrund der zum Chromosom kondensierte DNA-Doppelstrang. ⓘ
Als Gen wird meist ein Abschnitt auf der DNA bezeichnet, der Grundinformationen für die Entwicklung von Eigenschaften eines Individuums und zur Herstellung einer biologisch aktiven RNA enthält. Bei diesem Prozess der Transkription wird vom codogenen DNA-Strangabschnitt eine komplementäre Kopie in Form einer RNA hergestellt. ⓘ
Es gibt verschiedene Arten der RNA. Bei der Translation, einem Teilvorgang der Proteinbiosynthese, wird die Aminosäuresequenz des betreffenden Proteins von der mRNA abgelesen. Die Proteine übernehmen im Körper jeweils spezifische Funktionen, mit denen sich die Merkmale ausprägen können. Der Aktivitätszustand eines Gens bzw. dessen Ausprägung, seine Expression, kann in einzelnen Zellen verschieden reguliert werden. ⓘ
Als Erbanlage oder Erbfaktor werden allgemein die nur elektronenmikroskopisch sichtbaren Gene auf spezifischen Plätzen in den Chromosomen bezeichnet, da sie die Träger von Erbinformation sind, die durch Reproduktion an Nachkommen weitergegeben wird. Die Erforschung des Aufbaus, der Funktion und Vererbung von Genen ist Gegenstand der Genetik. Die gesamte Erbinformation einer Zelle wird Genom genannt. ⓘ
Geschichte

Entdeckung der diskreten Vererbungseinheiten
Die Existenz diskreter Vererbungseinheiten wurde erstmals von Gregor Mendel (1822-1884) vorgeschlagen. Von 1857 bis 1864 untersuchte er in Brünn im österreichischen Kaiserreich (der heutigen Tschechischen Republik) die Vererbungsmuster von 8000 gewöhnlichen Erbsenpflanzen und verfolgte die Vererbung unterschiedlicher Merkmale von den Eltern auf die Nachkommen. Er beschrieb diese mathematisch als 2n-Kombinationen, wobei n die Anzahl der unterschiedlichen Merkmale in den ursprünglichen Erbsen ist. Obwohl er den Begriff "Gen" nicht verwendete, erläuterte er seine Ergebnisse in Form von diskreten vererbten Einheiten, die zu beobachtbaren physischen Merkmalen führen. Diese Beschreibung war eine Vorstufe zu Wilhelm Johannsens Unterscheidung zwischen Genotyp (dem genetischen Material eines Organismus) und Phänotyp (den beobachtbaren Merkmalen dieses Organismus). Mendel war auch der erste, der die unabhängige Selektion, die Unterscheidung zwischen dominanten und rezessiven Merkmalen, die Unterscheidung zwischen Heterozygoten und Homozygoten und das Phänomen der diskontinuierlichen Vererbung nachwies. ⓘ
Vor Mendels Arbeiten war die vorherrschende Vererbungstheorie die der Mischvererbung, die davon ausging, dass jeder Elternteil Flüssigkeiten zum Befruchtungsprozess beitrug und dass sich die Merkmale der Eltern vermischten, um die Nachkommenschaft hervorzubringen. Charles Darwin entwickelte eine Theorie der Vererbung, die er als Pangenese bezeichnete, abgeleitet von griechisch pan ("alles, ganz") und genesis ("Geburt") / genos ("Ursprung"). Darwin verwendete den Begriff Gemmule, um hypothetische Teilchen zu beschreiben, die sich bei der Fortpflanzung vermischen würden. ⓘ
Mendels Arbeit blieb nach ihrer Erstveröffentlichung im Jahr 1866 weitgehend unbeachtet, wurde aber im späten 19. Jahrhundert von Hugo de Vries, Carl Correns und Erich von Tschermak wiederentdeckt, die in ihren eigenen Forschungen zu ähnlichen Schlussfolgerungen gelangten (und dies auch behaupteten). Hugo de Vries veröffentlichte 1889 sein Buch "Intrazelluläre Pangenese", in dem er postulierte, dass verschiedene Merkmale individuelle Vererbungsträger haben und dass die Vererbung bestimmter Merkmale in Organismen in Partikeln erfolgt. De Vries nannte diese Einheiten in Anlehnung an Darwins Pangenese-Theorie von 1868 "Pangene" (Pangens). ⓘ
Zwanzig Jahre später, 1909, führte Wilhelm Johannsen den Begriff "Gen" und 1906 William Bateson den Begriff "Genetik" ein, während Eduard Strasburger und andere noch den Begriff "Pangene" für die grundlegende physikalische und funktionelle Einheit der Vererbung verwendeten: Vorwort des Übersetzers, viii ⓘ
Entdeckung der DNA
Im 20. Jahrhundert wurden weitere Fortschritte beim Verständnis von Genen und Vererbung erzielt. Die Desoxyribonukleinsäure (DNS) wurde in den 1940er bis 1950er Jahren durch Experimente als molekularer Träger der genetischen Information nachgewiesen. Die Struktur der DNA wurde von Rosalind Franklin und Maurice Wilkins mit Hilfe der Röntgenkristallographie untersucht, was James D. Watson und Francis Crick dazu veranlasste, ein Modell des doppelsträngigen DNA-Moleküls zu veröffentlichen, dessen gepaarte Nukleotidbasen eine zwingende Hypothese für den Mechanismus der genetischen Replikation darstellten. ⓘ
In den frühen 1950er Jahren herrschte die Ansicht vor, dass die Gene in einem Chromosom wie diskrete Einheiten agierten, die durch Rekombination unteilbar und wie Perlen auf einer Schnur angeordnet waren. Die Experimente von Benzer mit Mutanten, die in der rII-Region des Bakteriophagen T4 defekt sind (1955-1959), zeigten, dass einzelne Gene eine einfache lineare Struktur haben und wahrscheinlich einem linearen Abschnitt der DNA entsprechen. ⓘ
Insgesamt begründeten diese Forschungsarbeiten das zentrale Dogma der Molekularbiologie, wonach Proteine aus RNA übersetzt werden, die wiederum von der DNA transkribiert wird. Seither hat sich gezeigt, dass es Ausnahmen von diesem Dogma gibt, wie z. B. die reverse Transkription bei Retroviren. Das moderne Studium der Genetik auf der Ebene der DNA wird als Molekulargenetik bezeichnet. ⓘ
Walter Fiers und sein Team waren 1972 die ersten, die die Sequenz eines Gens bestimmten: die des Hüllproteins des Bakteriophagen MS2. Die anschließende Entwicklung der DNA-Sequenzierung mit Kettenabbruch im Jahr 1977 durch Frederick Sanger verbesserte die Effizienz der Sequenzierung und machte sie zu einem routinemäßigen Laborinstrument. Eine automatisierte Version der Sanger-Methode wurde in frühen Phasen des Humangenomprojekts eingesetzt. ⓘ
Die moderne Synthese und ihre Nachfolger
Die zu Beginn des 20. Jahrhunderts entwickelten Theorien zur Integration der Mendelschen Genetik mit der Darwinschen Evolution werden als moderne Synthese bezeichnet, ein Begriff, der von Julian Huxley eingeführt wurde. ⓘ
Evolutionsbiologen haben dieses Konzept in der Folgezeit modifiziert, wie z. B. George C. Williams' gen-zentrierte Sicht der Evolution. Er schlug ein evolutionäres Konzept des Gens als Einheit der natürlichen Selektion mit der Definition vor: "das, was sich mit nennenswerter Häufigkeit abspaltet und rekombiniert": 24 In dieser Sichtweise wird das molekulare Gen als eine Einheit transkribiert, und das evolutionäre Gen vererbt sich als eine Einheit. Ähnliche Ideen, die die zentrale Rolle von Genen in der Evolution betonen, wurden von Richard Dawkins popularisiert. ⓘ
Molekulare Basis

DNA
Die überwiegende Mehrheit der Organismen verschlüsselt ihre Gene in langen DNA-Strängen (Desoxyribonukleinsäure). Die DNA besteht aus einer Kette aus vier Arten von Nukleotiduntereinheiten, die jeweils aus einem Fünf-Kohlenstoff-Zucker (2-Desoxyribose), einer Phosphatgruppe und einer der vier Basen Adenin, Cytosin, Guanin und Thymin bestehen: 2.1 ⓘ
Zwei DNA-Ketten winden sich umeinander, um eine DNA-Doppelhelix zu bilden, wobei sich das Phosphat-Zucker-Grundgerüst spiralförmig um die Außenseite windet und die Basen nach innen zeigen, wobei Adenin eine Basenpaarung mit Thymin und Guanin mit Cytosin bildet. Die Spezifität der Basenpaarung kommt dadurch zustande, dass Adenin und Thymin sich so ausrichten, dass sie zwei Wasserstoffbrücken bilden, während Cytosin und Guanin drei Wasserstoffbrücken bilden. Die beiden Stränge einer Doppelhelix müssen daher komplementär sein, d. h. ihre Basenabfolge muss so übereinstimmen, dass die Adenine des einen Strangs mit den Thyminen des anderen Strangs gepaart sind, und so weiter: 4.1 ⓘ
Aufgrund der chemischen Zusammensetzung der Pentosereste der Basen haben die DNA-Stränge eine Richtungsabhängigkeit. Ein Ende eines DNA-Polymers enthält eine freiliegende Hydroxylgruppe an der Desoxyribose; dies wird als das 3'-Ende des Moleküls bezeichnet. Das andere Ende enthält eine freiliegende Phosphatgruppe; dies ist das 5'-Ende. Die beiden Stränge einer Doppelhelix verlaufen in entgegengesetzter Richtung. Die Nukleinsäuresynthese, einschließlich der DNA-Replikation und Transkription, erfolgt in der 5'→3'-Richtung, da neue Nukleotide durch eine Dehydratisierungsreaktion hinzugefügt werden, die das freiliegende 3'-Hydroxyl als Nukleophil verwendet: 27.2 ⓘ
Die Expression von Genen, die in der DNA kodiert sind, beginnt mit der Transkription des Gens in die RNA, eine zweite Art von Nukleinsäure, die der DNA sehr ähnlich ist, deren Monomere jedoch den Zucker Ribose und nicht Desoxyribose enthalten. Außerdem enthält die RNA anstelle von Thymin die Base Uracil. RNA-Moleküle sind weniger stabil als DNA und bestehen in der Regel aus einem einzigen Strang. Gene, die für Proteine kodieren, bestehen aus einer Reihe von Drei-Nukleotid-Sequenzen, den so genannten Codons, die als "Wörter" in der genetischen "Sprache" dienen. Der genetische Code legt die Entsprechung zwischen Codons und Aminosäuren bei der Proteinübersetzung fest. Der genetische Code ist für alle bekannten Organismen nahezu identisch: 4.1 ⓘ
Chromosomen
Die Gesamtheit der Gene in einem Organismus oder einer Zelle wird als sein Genom bezeichnet, das auf einem oder mehreren Chromosomen gespeichert sein kann. Ein Chromosom besteht aus einer einzigen, sehr langen DNA-Helix, auf der Tausende von Genen codiert sind: 4.2 Der Bereich des Chromosoms, in dem sich ein bestimmtes Gen befindet, wird als sein Locus bezeichnet. Jeder Locus enthält ein Allel eines Gens; die Mitglieder einer Population können jedoch verschiedene Allele an diesem Locus haben, die jeweils eine leicht unterschiedliche Gensequenz aufweisen. ⓘ
Die meisten eukaryotischen Gene sind auf einem Satz großer, linearer Chromosomen gespeichert. Die Chromosomen sind im Zellkern zusammen mit Speicherproteinen, den Histonen, zu einer Einheit, dem Nukleosom, verpackt. Die auf diese Weise verpackte und kondensierte DNA wird als Chromatin bezeichnet: 4.2 Die Art und Weise, wie die DNA auf den Histonen gespeichert ist, sowie chemische Modifikationen des Histons selbst bestimmen, ob ein bestimmter Bereich der DNA für die Genexpression zugänglich ist. Neben den Genen enthalten eukaryotische Chromosomen auch Sequenzen, die dafür sorgen, dass die DNA ohne Abbau von Endbereichen kopiert und während der Zellteilung in die Tochterzellen sortiert wird: Replikationsursprünge, Telomere und das Zentromer: 4.2 Replikationsursprünge sind die Sequenzregionen, an denen die DNA-Replikation eingeleitet wird, um zwei Kopien des Chromosoms herzustellen. Telomere sind lange Abschnitte mit sich wiederholenden Sequenzen, die die Enden der linearen Chromosomen abdecken und den Abbau der kodierenden und regulatorischen Regionen während der DNA-Replikation verhindern. Die Länge der Telomere nimmt bei jeder Replikation des Genoms ab und wird mit dem Alterungsprozess in Verbindung gebracht. Das Zentromer ist für die Bindung der Spindelfasern erforderlich, um die Schwesterchromatiden während der Zellteilung in Tochterzellen zu trennen: 18.2 ⓘ
Prokaryoten (Bakterien und Archaeen) speichern ihre Genome in der Regel auf einem einzigen großen, kreisförmigen Chromosom. Auch einige eukaryontische Organellen enthalten ein zirkuläres Restchromosom mit einer geringen Anzahl von Genen: 14.4 Prokaryoten ergänzen ihr Chromosom manchmal mit zusätzlichen kleinen DNA-Kreisen, den so genannten Plasmiden, die in der Regel nur für einige wenige Gene kodieren und zwischen Individuen übertragbar sind. So sind beispielsweise die Gene für Antibiotikaresistenzen in der Regel auf bakteriellen Plasmiden kodiert und können durch horizontalen Gentransfer zwischen einzelnen Zellen, auch solchen verschiedener Arten, weitergegeben werden. ⓘ
Während die Chromosomen von Prokaryonten eine relativ hohe Gendichte aufweisen, enthalten die Chromosomen von Eukaryonten oft DNA-Bereiche, die keine offensichtliche Funktion haben. Einfache einzellige Eukaryoten haben relativ geringe Mengen solcher DNA, während die Genome komplexer mehrzelliger Organismen, einschließlich des Menschen, die absolute Mehrheit an DNA ohne erkennbare Funktion enthalten. Diese DNA wurde oft als "Junk-DNA" bezeichnet. Neuere Analysen deuten jedoch darauf hin, dass, obwohl die proteinkodierende DNA nur knapp 2 % des menschlichen Genoms ausmacht, etwa 80 % der Basen im Genom exprimiert werden können, so dass der Begriff "Junk-DNA" möglicherweise eine falsche Bezeichnung ist. ⓘ
Auf molekularer Ebene besteht ein Gen aus zwei unterschiedlichen Bereichen:
- Einem DNA-Abschnitt, von dem durch Transkription eine einzelsträngige RNA-Kopie hergestellt wird.
- Allen zusätzlichen DNA-Abschnitten, die an der Regulation dieses Kopiervorgangs beteiligt sind. ⓘ
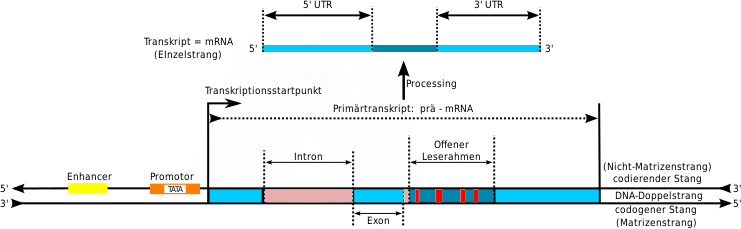
Es gibt verschiedene Besonderheiten im Aufbau von Genen verschiedener Lebewesen. In der Zeichnung wird der Aufbau eines typischen eukaryotischen Gens dargestellt, das ein Protein codiert. ⓘ
Struktur und Funktion
Struktur
|
|
Die Struktur eines Gens besteht aus vielen Elementen, von denen die eigentliche Protein kodierende Sequenz oft nur ein kleiner Teil ist. Dazu gehören DNA-Regionen, die nicht transkribiert werden, sowie untranslatierte Regionen der RNA. ⓘ
Flankierend zum offenen Leseraster enthalten Gene eine regulatorische Sequenz, die für ihre Expression erforderlich ist. Zunächst benötigen die Gene eine Promotorsequenz. Der Promotor wird von Transkriptionsfaktoren erkannt und gebunden, die die RNA-Polymerase rekrutieren und ihr helfen, an die Region zu binden, um die Transkription einzuleiten: 7.1 Die Erkennung erfolgt in der Regel in Form einer Konsenssequenz wie der TATA-Box. Ein Gen kann mehr als einen Promotor haben, was zu Boten-RNAs (mRNA) führt, die sich darin unterscheiden, wie weit sie am 5'-Ende reichen. Gene mit hoher Transkriptionsrate haben "starke" Promotorsequenzen, die mit Transkriptionsfaktoren assoziiert sind und dadurch die Transkription mit hoher Geschwindigkeit einleiten. Andere Gene haben "schwache" Promotoren, die schwache Verbindungen mit Transkriptionsfaktoren eingehen und die Transkription seltener auslösen: 7.2 Eukaryotische Promotorregionen sind viel komplexer und schwieriger zu identifizieren als prokaryotische Promotoren: 7.3 ⓘ
Darüber hinaus können Gene regulatorische Regionen aufweisen, die viele Kilobasen vor oder nach dem offenen Leseraster liegen und die Expression verändern. Diese wirken durch Bindung an Transkriptionsfaktoren, die dann eine Schleife in der DNA verursachen, so dass die regulatorische Sequenz (und der gebundene Transkriptionsfaktor) in die Nähe der RNA-Polymerase-Bindungsstelle gelangen. Enhancer beispielsweise erhöhen die Transkription durch Bindung eines Aktivatorproteins, das dann dazu beiträgt, die RNA-Polymerase an den Promotor zu rekrutieren; umgekehrt binden Silencer Repressorproteine und machen die DNA für die RNA-Polymerase weniger verfügbar. ⓘ
Die transkribierte prä-mRNA enthält an beiden Enden untranslatierte Regionen, die Bindungsstellen für Ribosomen, RNA-bindende Proteine, miRNA sowie Terminatoren und Start- und Stoppcodons enthalten. Darüber hinaus enthalten die meisten offenen Leserahmen von Eukaryonten untranslatierte Introns, die entfernt werden, und Exons, die in einem als RNA-Spleißen bekannten Prozess miteinander verbunden werden. Schließlich werden die Enden der Gentranskripte durch Spaltungs- und Polyadenylierungsstellen (CPA) definiert, an denen neu produzierte pre-mRNA gespalten und eine Kette von ~200 Adenosinmonophosphaten am 3'-Ende angehängt wird. Der Poly(A)-Schwanz schützt die reife mRNA vor dem Abbau und hat weitere Funktionen, die die Translation, die Lokalisierung und den Transport des Transkripts aus dem Zellkern beeinflussen. Durch Spleißen, gefolgt von CPA, entsteht die endgültige reife mRNA, die für das Protein oder RNA-Produkt kodiert. Obwohl die allgemeinen Mechanismen, die die Lokalisierung der menschlichen Gene bestimmen, bekannt sind, ist die Identifizierung der genauen Faktoren, die diese zellulären Prozesse regulieren, ein aktives Forschungsgebiet. Die bekannten Sequenzmerkmale in der 3'-UTR können zum Beispiel nur die Hälfte aller menschlichen Genenden erklären. ⓘ
Viele prokaryotische Gene sind in Operonen organisiert, die mehrere proteinkodierende Sequenzen enthalten, die als Einheit transkribiert werden. Die Gene in einem Operon werden als kontinuierliche Boten-RNA transkribiert, die als polyzistronische mRNA bezeichnet wird. Der Begriff Cistron ist in diesem Zusammenhang gleichbedeutend mit Gen. Die Transkription der mRNA eines Operons wird häufig durch einen Repressor gesteuert, der je nach Vorhandensein bestimmter Metaboliten in einem aktiven oder inaktiven Zustand vorliegen kann. Im aktiven Zustand bindet der Repressor an eine DNA-Sequenz am Anfang des Operons, die so genannte Operatorregion, und unterdrückt die Transkription des Operons; ist der Repressor inaktiv, kann die Transkription des Operons stattfinden (siehe z. B. Lac-Operon). Die Produkte von Operon-Genen haben in der Regel verwandte Funktionen und sind in dasselbe Regulationsnetzwerk eingebunden: 7.3 ⓘ
Funktionelle Definitionen
Regulatorische Regionen eines Gens, wie z. B. Enhancer, müssen nicht unbedingt in der Nähe der kodierenden Sequenz auf dem linearen Molekül liegen, da die dazwischen liegende DNA in einer Schleife angeordnet werden kann, um das Gen und seine regulatorische Region in die Nähe zu bringen. In ähnlicher Weise können die Introns eines Gens viel größer sein als seine Exons. Regulatorische Regionen können sich sogar auf völlig unterschiedlichen Chromosomen befinden und in Transregionen arbeiten, damit regulatorische Regionen auf einem Chromosom mit Zielgenen auf einem anderen Chromosom in Kontakt kommen können. ⓘ
Frühe Arbeiten in der Molekulargenetik legten das Konzept nahe, dass ein Gen ein Protein herstellt. Dieses Konzept (ursprünglich als "Ein-Gen-ein-Enzym-Hypothese" bezeichnet) geht auf eine einflussreiche Arbeit von George Beadle und Edward Tatum aus dem Jahr 1941 über Experimente mit Mutanten des Pilzes Neurospora crassa zurück. Norman Horowitz, ein früher Kollege in der Neurospora-Forschung, erinnerte sich 2004 daran, dass "diese Experimente die Wissenschaft dessen begründeten, was Beadle und Tatum biochemische Genetik nannten. In Wirklichkeit waren sie der Startschuss für die Molekulargenetik und alle darauf folgenden Entwicklungen". Das Konzept "ein Gen - ein Protein" wurde seit der Entdeckung von Genen, die durch alternatives Spleißen für mehrere Proteine kodieren können, und von kodierenden Sequenzen, die in kurzen Abschnitten im Genom aufgespalten sind und deren mRNAs durch Trans-Splicing verkettet werden, verfeinert. ⓘ
Um der Komplexität dieser verschiedenen Phänomene gerecht zu werden, wird manchmal eine weit gefasste operationelle Definition verwendet, bei der ein Gen als eine Vereinigung genomischer Sequenzen definiert wird, die für eine kohärente Reihe potenziell überlappender funktioneller Produkte kodieren. Bei dieser Definition werden Gene nach ihren funktionellen Produkten (Proteine oder RNA) und nicht nach ihren spezifischen DNA-Loci kategorisiert, wobei regulatorische Elemente als genassoziierte Regionen eingestuft werden. ⓘ
Überschneidungen zwischen Genen
Es ist auch möglich, dass Gene dieselbe DNA-Sequenz überlappen und als unterschiedliche, aber überlappende Gene betrachtet werden. Die derzeitige Definition eines überlappenden Gens ist bei Eukaryoten, Prokaryoten und Viren unterschiedlich. Bei Eukaryoten wurden sie kürzlich definiert als "wenn mindestens ein Nukleotid zwischen den äußersten Grenzen der primären Transkripte von zwei oder mehr Genen liegt, so dass eine DNA-Basenmutation an der Überlappungsstelle die Transkripte aller an der Überlappung beteiligten Gene beeinträchtigen würde". Bei Prokaryoten und Viren wurden sie kürzlich definiert als "wenn die kodierenden Sequenzen zweier Gene ein Nukleotid entweder auf demselben oder auf entgegengesetzten Strängen teilen". ⓘ
Genexpression
In allen Organismen sind zwei Schritte erforderlich, um die in der DNA eines Gens kodierte Information abzulesen und das von ihr spezifizierte Protein herzustellen. Zunächst wird die DNA des Gens in Boten-RNA (mRNA) umgeschrieben: 6.1 Zweitens wird diese mRNA in Protein übersetzt: 6.2 RNA-kodierende Gene müssen noch den ersten Schritt durchlaufen, werden aber nicht in Protein übersetzt. Der Prozess der Herstellung eines biologisch funktionsfähigen RNA- oder Proteinmoleküls wird als Genexpression bezeichnet, und das resultierende Molekül wird als Genprodukt bezeichnet. ⓘ
Genetischer Code

Die Nukleotidsequenz der DNA eines Gens spezifiziert die Aminosäuresequenz eines Proteins durch den genetischen Code. Sätze von drei Nukleotiden, so genannte Codons, entsprechen jeweils einer bestimmten Aminosäure: 6 Das Prinzip, dass drei aufeinander folgende Basen der DNA für jede Aminosäure kodieren, wurde 1961 anhand von Frameshift-Mutationen im rIIB-Gen des Bakteriophagen T4 nachgewiesen (siehe Experiment von Crick, Brenner et al.). ⓘ
Zusätzlich zeigen ein "Startcodon" und drei "Stoppcodons" den Beginn und das Ende der Proteincodierungsregion an. Es gibt 64 mögliche Codons (vier mögliche Nukleotide an jeder der drei Positionen, also 43 mögliche Codons) und nur 20 Standardaminosäuren; daher ist der Code redundant und mehrere Codons können dieselbe Aminosäure spezifizieren. Die Entsprechung zwischen Codons und Aminosäuren ist bei allen bekannten Lebewesen nahezu universell. ⓘ
Transkription
Bei der Transkription entsteht ein einzelsträngiges RNA-Molekül, die so genannte Boten-RNA, deren Nukleotidsequenz komplementär zur DNA ist, von der sie transkribiert wurde: 6.1 Die mRNA fungiert als Zwischenglied zwischen dem DNA-Gen und seinem endgültigen Proteinprodukt. Die DNA des Gens wird als Vorlage verwendet, um eine komplementäre mRNA zu erzeugen. Die mRNA entspricht der Sequenz des kodierenden DNA-Strangs des Gens, da sie als Komplement des Vorlagenstrangs synthetisiert wird. Die Transkription wird von einem Enzym namens RNA-Polymerase durchgeführt, das den Vorlagenstrang in der Richtung von 3' nach 5' liest und die RNA von 5' nach 3' synthetisiert. Um die Transkription einzuleiten, erkennt und bindet die Polymerase zunächst eine Promotorregion des Gens. Ein wichtiger Mechanismus der Genregulation ist daher die Blockierung oder Sequestrierung der Promotorregion, entweder durch enge Bindung durch Repressormoleküle, die die Polymerase physisch blockieren, oder durch die Organisation der DNA, so dass die Promotorregion nicht zugänglich ist..: 7 ⓘ
Bei Prokaryonten erfolgt die Transkription im Zytoplasma; bei sehr langen Transkripten kann die Translation am 5'-Ende der RNA beginnen, während das 3'-Ende noch transkribiert wird. Bei Eukaryonten findet die Transkription im Zellkern statt, wo die DNA der Zelle gespeichert ist. Das von der Polymerase produzierte RNA-Molekül wird als primäres Transkript bezeichnet und erfährt posttranskriptionelle Modifikationen, bevor es zur Translation in das Zytoplasma exportiert wird. Eine dieser Modifikationen ist das Spleißen von Introns, d. h. von Sequenzen in der transkribierten Region, die nicht für ein Protein kodieren. Alternative Spleißmechanismen können dazu führen, dass reife Transkripte desselben Gens unterschiedliche Sequenzen aufweisen und somit für unterschiedliche Proteine kodieren. Dies ist eine wichtige Form der Regulierung in eukaryontischen Zellen und kommt auch in einigen Prokaryonten vor: 7.5 ⓘ
Übersetzung
Die Translation ist der Prozess, bei dem ein reifes mRNA-Molekül als Vorlage für die Synthese eines neuen Proteins verwendet wird: 6.2 Die Translation wird von Ribosomen durchgeführt, großen Komplexen aus RNA und Protein, die für die Durchführung der chemischen Reaktionen verantwortlich sind, mit denen neue Aminosäuren durch die Bildung von Peptidbindungen an eine wachsende Polypeptidkette angehängt werden. Der genetische Code wird in Einheiten, die Codons genannt werden, über Interaktionen mit spezialisierten RNA-Molekülen, der so genannten Transfer-RNA (tRNA), drei Nukleotide auf einmal gelesen. Jede tRNA hat drei ungepaarte Basen, die als Anticodon bezeichnet werden und komplementär zu dem Codon sind, das sie auf der mRNA liest. Die tRNA ist außerdem kovalent an die durch das komplementäre Codon spezifizierte Aminosäure gebunden. Wenn die tRNA an ihr komplementäres Codon in einem mRNA-Strang bindet, fügt das Ribosom seine Aminosäurefracht an die neue Polypeptidkette an, die vom Aminoterminus zum Carboxylterminus synthetisiert wird. Während und nach der Synthese müssen sich die meisten neuen Proteine zu ihrer aktiven dreidimensionalen Struktur falten, bevor sie ihre zellulären Funktionen ausüben können: 3 ⓘ
Regulierung
Gene werden so reguliert, dass sie nur dann exprimiert werden, wenn das Produkt benötigt wird, da die Expression auf begrenzte Ressourcen zurückgreift: 7 Eine Zelle reguliert ihre Genexpression in Abhängigkeit von ihrer äußeren Umgebung (z. B. verfügbare Nährstoffe, Temperatur und andere Stressfaktoren), ihrer inneren Umgebung (z. B. Zellteilungszyklus, Stoffwechsel, Infektionsstatus) und ihrer spezifischen Rolle in einem mehrzelligen Organismus. Die Genexpression kann auf jeder Stufe reguliert werden: von der Transkriptionsinitiierung über die RNA-Prozessierung bis hin zur posttranslationalen Modifikation des Proteins. Die Regulierung von Genen des Laktosestoffwechsels in E. coli (lac-Operon) war der erste derartige Mechanismus, der 1961 beschrieben wurde. ⓘ
RNA-Gene
Ein typisches proteinkodierendes Gen wird zunächst in RNA kopiert, die als Zwischenprodukt bei der Herstellung des endgültigen Proteinprodukts dient: 6.1 In anderen Fällen sind die RNA-Moleküle die eigentlichen funktionellen Produkte, wie bei der Synthese der ribosomalen RNA und der Transfer-RNA. Einige als Ribozyme bezeichnete RNAs sind in der Lage, eine enzymatische Funktion zu erfüllen, und die microRNA hat eine regulatorische Funktion. Die DNA-Sequenzen, von denen solche RNAs transkribiert werden, werden als nicht-kodierende RNA-Gene bezeichnet. ⓘ
Einige Viren speichern ihr gesamtes Genom in Form von RNA und enthalten überhaupt keine DNA. Da sie ihre Gene in Form von RNA speichern, können ihre zellulären Wirte ihre Proteine synthetisieren, sobald sie infiziert sind, ohne auf die Transkription warten zu müssen. RNA-Retroviren, wie HIV, benötigen dagegen eine reverse Transkription ihres Genoms von RNA in DNA, bevor ihre Proteine synthetisiert werden können. Die RNA-vermittelte epigenetische Vererbung wurde auch bei Pflanzen und sehr selten bei Tieren beobachtet. ⓘ
Vererbung
Organismen erben ihre Gene von ihren Eltern. Ungeschlechtliche Organismen erben einfach eine vollständige Kopie des Genoms ihrer Eltern. Sexuelle Organismen haben zwei Kopien von jedem Chromosom, weil sie von jedem Elternteil einen vollständigen Satz erben: 1 ⓘ
Mendelsche Vererbung
Nach der Mendelschen Vererbung sind Variationen des Phänotyps (beobachtbare physische und Verhaltensmerkmale) eines Organismus zum Teil auf Variationen seines Genotyps (bestimmter Satz von Genen) zurückzuführen. Jedes Gen spezifiziert ein bestimmtes Merkmal, wobei eine unterschiedliche Sequenz eines Gens (Allele) zu unterschiedlichen Phänotypen führt. Die meisten eukaryontischen Organismen (wie die Erbsenpflanzen, an denen Mendel arbeitete) haben zwei Allele für jedes Merkmal, eines von jedem Elternteil geerbt: 20 ⓘ
Allele an einem Locus können dominant oder rezessiv sein; dominante Allele führen zu den entsprechenden Phänotypen, wenn sie mit einem beliebigen anderen Allel für dasselbe Merkmal gepaart sind, während rezessive Allele nur dann zu dem entsprechenden Phänotyp führen, wenn sie mit einer anderen Kopie desselben Allels gepaart sind. Wenn Sie die Genotypen der Organismen kennen, können Sie feststellen, welche Allele dominant und welche rezessiv sind. Wenn beispielsweise das Allel, das bei Erbsenpflanzen für hohe Stängel verantwortlich ist, gegenüber dem Allel, das für kurze Stängel verantwortlich ist, dominant ist, dann werden Erbsenpflanzen, die ein hohes Allel von einem Elternteil und ein kurzes Allel vom anderen Elternteil erben, auch hohe Stängel haben. Mendels Arbeit zeigte, dass sich Allele bei der Produktion von Keimzellen unabhängig voneinander vervielfältigen und so für Variation in der nächsten Generation sorgen. Obwohl die Mendelsche Vererbung nach wie vor ein gutes Modell für viele Merkmale ist, die von einzelnen Genen bestimmt werden (einschließlich einer Reihe bekannter genetischer Störungen), umfasst sie nicht die physikalischen Prozesse der DNA-Replikation und Zellteilung. ⓘ
DNA-Replikation und Zellteilung
Wachstum, Entwicklung und Fortpflanzung von Organismen beruhen auf der Zellteilung, dem Prozess, bei dem sich eine einzelne Zelle in zwei normalerweise identische Tochterzellen teilt. Dazu muss zunächst ein Duplikat jedes Gens im Genom in einem als DNA-Replikation bezeichneten Prozess erstellt werden: 5.2 Die Kopien werden von speziellen Enzymen, den so genannten DNA-Polymerasen, hergestellt, die einen Strang der doppelhelicalen DNA, den so genannten Vorlagenstrang, "ablesen" und einen neuen komplementären Strang synthetisieren. Da die DNA-Doppelhelix durch Basenpaarung zusammengehalten wird, legt die Sequenz eines Strangs die Sequenz des Komplementärstrangs vollständig fest; daher muss nur ein Strang von dem Enzym gelesen werden, um eine originalgetreue Kopie zu erzeugen. Der Prozess der DNA-Replikation ist semikonservativ, d. h. die Kopie des Genoms, die jede Tochterzelle erbt, enthält einen ursprünglichen und einen neu synthetisierten DNA-Strang: 5.2 ⓘ
Die Geschwindigkeit der DNA-Replikation in lebenden Zellen wurde erstmals anhand der Verlängerungsrate der Phagen-T4-DNA in phageninfizierten E. coli gemessen und erwies sich als beeindruckend schnell. Während des exponentiellen DNA-Anstiegs bei 37 °C betrug die Elongationsrate 749 Nukleotide pro Sekunde. ⓘ
Nach Abschluss der DNA-Replikation muss die Zelle die beiden Kopien des Genoms physisch trennen und sich in zwei verschiedene membrangebundene Zellen teilen: 18.2 Bei Prokaryonten (Bakterien und Archaeen) geschieht dies in der Regel durch einen relativ einfachen Prozess, die so genannte binäre Spaltung, bei der sich jedes zirkuläre Genom an die Zellmembran anlagert und in die Tochterzellen getrennt wird, während die Membran invaginiert, um das Zytoplasma in zwei membrangebundene Teile zu teilen. Die binäre Spaltung ist im Vergleich zu den Zellteilungsgeschwindigkeiten bei Eukaryonten extrem schnell. Die DNA-Replikation findet in einer Phase dieses Zyklus statt, die als S-Phase bezeichnet wird, während der Prozess der Chromosomensegregation und der Teilung des Zytoplasmas in der M-Phase stattfindet: 18.1 ⓘ
Molekulare Vererbung
Die Vervielfältigung und Weitergabe des genetischen Materials von einer Zellgeneration zur nächsten ist die Grundlage für die molekulare Vererbung und das Bindeglied zwischen dem klassischen und dem molekularen Bild von Genen. Organismen erben die Eigenschaften ihrer Eltern, weil die Zellen der Nachkommen Kopien der Gene in den Zellen ihrer Eltern enthalten. Bei sich ungeschlechtlich fortpflanzenden Organismen ist der Nachkomme eine genetische Kopie oder ein Klon des Elternorganismus. Bei sich sexuell fortpflanzenden Organismen werden durch eine spezielle Form der Zellteilung, die Meiose, Zellen erzeugt, die als Keimzellen bezeichnet werden und haploid sind oder nur eine Kopie jedes Gens enthalten. 20.2 Die von den Weibchen erzeugten Keimzellen werden Eizellen oder Eizellen genannt, die von den Männchen erzeugten Keimzellen werden Spermien genannt. Zwei Keimzellen verschmelzen zu einer diploiden befruchteten Eizelle, einer einzigen Zelle, die zwei Gensätze enthält, wobei eine Kopie jedes Gens von der Mutter und eine vom Vater stammt.: 20 ⓘ
Während der meiotischen Zellteilung kann es manchmal zu einem Ereignis kommen, das als genetische Rekombination oder Crossing-over bezeichnet wird und bei dem eine DNA-Länge auf einem Chromatid mit einer DNA-Länge auf dem entsprechenden homologen Nicht-Schwesterchromatid vertauscht wird. Dies kann zu einem Reassortment von ansonsten verbundenen Allelen führen: 5.5 Das Mendelsche Prinzip der unabhängigen Sortierung besagt, dass jedes der beiden Gene eines Elternteils für jedes Merkmal unabhängig in die Gameten sortiert wird; welches Allel ein Organismus für ein Merkmal erbt, ist unabhängig davon, welches Allel er für ein anderes Merkmal erbt. Dies gilt allerdings nur für Gene, die nicht auf demselben Chromosom liegen oder die auf demselben Chromosom sehr weit voneinander entfernt sind. Je näher zwei Gene auf demselben Chromosom liegen, desto enger sind sie in den Keimzellen miteinander verbunden und desto häufiger treten sie gemeinsam auf (so genannte genetische Kopplung). Gene, die sehr nahe beieinander liegen, werden im Grunde nie getrennt, da es äußerst unwahrscheinlich ist, dass ein Kreuzungspunkt zwischen ihnen auftritt. ⓘ
Molekulare Entwicklung
Mutation
Die DNA-Replikation ist im Großen und Ganzen sehr genau, aber es treten auch Fehler (Mutationen) auf: 7.6 Die Fehlerrate in eukaryontischen Zellen kann bis zu 10-8 pro Nukleotid und Replikation betragen, während sie bei einigen RNA-Viren bis zu 10-3 betragen kann. Das bedeutet, dass jedes menschliche Genom in jeder Generation 1-2 neue Mutationen anhäuft. Kleine Mutationen können durch die DNA-Replikation und die Folgen von DNA-Schäden verursacht werden und umfassen Punktmutationen, bei denen eine einzelne Base verändert wird, und Frameshift-Mutationen, bei denen eine einzelne Base eingefügt oder gelöscht wird. Jede dieser Mutationen kann das Gen durch Missense (Änderung eines Codons, um eine andere Aminosäure zu kodieren) oder Nonsense (ein vorzeitiges Stoppcodon) verändern. Größere Mutationen können durch Fehler bei der Rekombination verursacht werden, die zu Chromosomenanomalien führen, einschließlich der Duplikation, Deletion, Umordnung oder Inversion großer Abschnitte eines Chromosoms. Darüber hinaus können die DNA-Reparaturmechanismen bei der Behebung von physischen Schäden am Molekül Mutationsfehler verursachen. Die Reparatur ist, selbst bei Mutationen, für das Überleben wichtiger als die Wiederherstellung einer exakten Kopie, beispielsweise bei der Reparatur von Doppelstrangbrüchen: 5.4 ⓘ
Wenn in der Population einer Art mehrere unterschiedliche Allele für ein Gen vorhanden sind, wird es als polymorph bezeichnet. Die meisten verschiedenen Allele sind funktionell gleichwertig, einige Allele können jedoch unterschiedliche phänotypische Merkmale hervorrufen. Das häufigste Allel eines Gens wird als Wildtyp bezeichnet, während seltene Allele als Mutanten bezeichnet werden. Die genetische Variation der relativen Häufigkeit verschiedener Allele in einer Population ist sowohl auf natürliche Selektion als auch auf genetische Drift zurückzuführen. Das Wildtyp-Allel ist nicht notwendigerweise der Vorfahre der selteneren Allele, und es ist auch nicht unbedingt fitter. ⓘ
Die meisten Mutationen innerhalb von Genen sind neutral und haben keine Auswirkungen auf den Phänotyp des Organismus (stille Mutationen). Einige Mutationen verändern die Aminosäuresequenz nicht, weil mehrere Codons für dieselbe Aminosäure kodieren (synonyme Mutationen). Andere Mutationen können neutral sein, wenn sie zu einer Änderung der Aminosäuresequenz führen, das Protein aber auch mit der neuen Aminosäure noch ähnlich funktioniert (z. B. konservative Mutationen). Viele Mutationen sind jedoch schädlich oder sogar tödlich und werden durch natürliche Selektion aus den Populationen entfernt. Genetische Störungen sind das Ergebnis schädlicher Mutationen und können auf eine spontane Mutation im betroffenen Individuum zurückzuführen sein oder vererbt werden. Ein kleiner Teil der Mutationen schließlich ist vorteilhaft, verbessert die Fitness des Organismus und ist für die Evolution äußerst wichtig, da ihre gerichtete Auswahl zu einer adaptiven Evolution führt: 7.6 ⓘ
Sequenzhomologie

Gene mit einem jüngsten gemeinsamen Vorfahren, die also eine gemeinsame evolutionäre Abstammung haben, werden als Homologe bezeichnet. Diese Gene entstehen entweder durch Genduplikation innerhalb des Genoms eines Organismus (paraloge Gene) oder sind das Ergebnis einer Divergenz der Gene nach einem Speziationsereignis (orthologe Gene) und erfüllen in verwandten Organismen oft die gleichen oder ähnliche Funktionen. Es wird oft angenommen, dass die Funktionen orthologer Gene ähnlicher sind als die paraloger Gene, obwohl der Unterschied minimal ist. ⓘ
Die Verwandtschaft zwischen Genen kann durch den Vergleich der Sequenzabgleiche ihrer DNA gemessen werden: 7.6 Der Grad der Sequenzähnlichkeit zwischen homologen Genen wird als konservierte Sequenz bezeichnet. Die meisten Änderungen an der Sequenz eines Gens haben keine Auswirkungen auf seine Funktion, so dass Gene im Laufe der Zeit durch neutrale molekulare Evolution Mutationen ansammeln. Darüber hinaus führt jede Selektion auf ein Gen dazu, dass seine Sequenz mit einer anderen Geschwindigkeit divergiert. Gene, die einer stabilisierenden Selektion unterworfen sind, verändern sich langsamer, während Gene, die einer gerichteten Selektion unterliegen, ihre Sequenz schneller verändern. Die Sequenzunterschiede zwischen Genen können für phylogenetische Analysen verwendet werden, um zu untersuchen, wie sich diese Gene entwickelt haben und wie die Organismen, von denen sie stammen, miteinander verwandt sind. ⓘ
Der Ursprung neuer Gene

Die häufigste Quelle neuer Gene in eukaryontischen Abstammungslinien ist die Genduplikation, bei der eine Variation der Kopienzahl eines bestehenden Gens im Genom entsteht. Die daraus resultierenden Gene (Paraloge) können dann in Sequenz und Funktion voneinander abweichen. Die auf diese Weise gebildeten Gengruppen bilden eine Genfamilie. Genduplikationen und -verluste innerhalb einer Familie sind häufig und stellen eine wichtige Quelle der evolutionären Artenvielfalt dar. Manchmal kann eine Genduplikation zu einer nicht funktionsfähigen Kopie eines Gens führen, oder eine funktionsfähige Kopie kann Mutationen ausgesetzt sein, die zu einem Funktionsverlust führen; solche nicht funktionsfähigen Gene werden als Pseudogene bezeichnet: 7.6 ⓘ
"Orphan"-Gene, deren Sequenz keine Ähnlichkeit mit bestehenden Genen aufweist, sind weniger häufig als Genduplikate. Das menschliche Genom enthält schätzungsweise 18 bis 60 Gene, die keine erkennbaren Homologe außerhalb des Menschen aufweisen. Orphan-Gene entstehen in erster Linie entweder 'de novo' aus einer zuvor nicht kodierenden Sequenz oder durch Genduplikation, gefolgt von einer so schnellen Sequenzänderung, dass die ursprüngliche Verwandtschaft nicht mehr nachweisbar ist. De-novo-Gene sind in der Regel kürzer und einfacher strukturiert als die meisten eukaryontischen Gene und weisen nur wenige oder gar keine Introns auf. Über lange evolutionäre Zeiträume hinweg kann die Entstehung von De-novo-Genen für einen erheblichen Teil der taxonomisch begrenzten Genfamilien verantwortlich sein. ⓘ
Der horizontale Gentransfer bezieht sich auf die Übertragung von genetischem Material durch einen anderen Mechanismus als die Fortpflanzung. Dieser Mechanismus ist eine häufige Quelle neuer Gene bei Prokaryonten, und manchmal wird angenommen, dass er mehr zur genetischen Variation beiträgt als die Genduplikation. Er ist ein gängiges Mittel zur Verbreitung von Antibiotikaresistenz, Virulenz und adaptiven Stoffwechselfunktionen. Obwohl der horizontale Gentransfer bei Eukaryonten selten ist, wurden wahrscheinlich Beispiele von Protisten- und Algengenomen identifiziert, die Gene bakteriellen Ursprungs enthalten. ⓘ
Genom
Das Genom ist das gesamte genetische Material eines Organismus und umfasst sowohl die Gene als auch die nicht codierenden Sequenzen. Eukaryotengene können mit FINDER annotiert werden. ⓘ
Anzahl von Genen

Die Größe des Genoms und die Anzahl der darin kodierten Gene ist von Organismus zu Organismus sehr unterschiedlich. Die kleinsten Genome kommen bei Viren und Viroiden vor (die als ein einziges nicht codierendes RNA-Gen fungieren). Umgekehrt können Pflanzen extrem große Genome haben, wie z. B. Reis mit mehr als 46.000 proteinkodierenden Genen. Die Gesamtzahl der proteinkodierenden Gene (das Proteom der Erde) wird auf 5 Millionen Sequenzen geschätzt. ⓘ
Obwohl die Anzahl der Basenpaare der DNA im menschlichen Genom seit den 1960er Jahren bekannt ist, hat sich die geschätzte Anzahl der Gene im Laufe der Zeit geändert, da die Definitionen von Genen und die Methoden zu deren Nachweis verfeinert wurden. Anfängliche theoretische Vorhersagen über die Anzahl der menschlichen Gene beliefen sich auf bis zu 2.000.000. Frühe experimentelle Messungen ergaben, dass es 50.000-100.000 transkribierte Gene (expressed sequence tags) gibt. Später zeigte die Sequenzierung im Rahmen des Humangenomprojekts, dass es sich bei vielen dieser Transkripte um alternative Varianten derselben Gene handelte, und die Gesamtzahl der proteinkodierenden Gene wurde auf etwa 20.000 reduziert, wobei 13 Gene im mitochondrialen Genom kodiert werden. Mit dem GENCODE Annotationsprojekt ist diese Schätzung weiter auf 19.000 gesunken. Nur 1 bis 2 % des menschlichen Genoms bestehen aus proteinkodierenden Sequenzen, der Rest ist "nicht-kodierende" DNA wie Introns, Retrotransposons und nicht-kodierende RNAs. Jeder vielzellige Organismus hat alle seine Gene in jeder Zelle seines Körpers, aber nicht jedes Gen funktioniert in jeder Zelle. ⓘ
In der nachfolgenden Tabelle sind einige Proteine und das jeweils codierende Gen aufgeführt. ⓘ
| Protein | Anzahl der Aminosäuren |
Gen | Anzahl der Basenpaare |
Anzahl codierender Basenpaare |
Anteil codierender Sequenz |
Referenz ⓘ |
|---|---|---|---|---|---|---|
| Dystrophin | 3685 | DMD | 2.500.000 | 11.055 | 0,44 % | |
| FOXP2 | 715 | FOXP2 | 603.000 | 2145 | 0,36 % | |
| Neurofibromin | 2838 | NF1 | 280.000 | 8514 | 3,0 % | |
| BRCA2 | 3418 | BRCA2 | 84.000 | 10.254 | 12,2 % | |
| BRCA1 | 1863 | BRCA1 | 81.000 | 5589 | 6,9 % | |
| Survivin | 142 | BIRC5 | 15.000 | 426 | 2,9 % |
Wesentliche Gene
Als essentielle Gene werden die Gene bezeichnet, die für das Überleben eines Organismus als entscheidend gelten. Bei dieser Definition wird davon ausgegangen, dass alle relevanten Nährstoffe im Überfluss vorhanden sind und es keinen Umweltstress gibt. Nur ein kleiner Teil der Gene eines Organismus ist essenziell. Bei Bakterien sind schätzungsweise 250-400 Gene für Escherichia coli und Bacillus subtilis essenziell, das sind weniger als 10 % ihrer Gene. Die Hälfte dieser Gene sind Orthologe in beiden Organismen und sind größtenteils an der Proteinsynthese beteiligt. Bei der Knospenhefe Saccharomyces cerevisiae ist die Zahl der essenziellen Gene mit 1000 Genen (~20 % der Gene) etwas höher. Bei höheren Eukaryonten ist die Zahl zwar schwieriger zu messen, aber bei Mäusen und Menschen schätzt man die Zahl der essenziellen Gene auf etwa 2000 (~10 % ihrer Gene). Der synthetische Organismus Syn 3 hat ein minimales Genom mit 473 essenziellen Genen und quasi-essenziellen Genen (die für ein schnelles Wachstum notwendig sind), wobei 149 eine unbekannte Funktion haben. ⓘ
Zu den essenziellen Genen gehören Housekeeping-Gene (die für die grundlegenden Zellfunktionen entscheidend sind) sowie Gene, die zu verschiedenen Zeiten in der Entwicklung oder im Lebenszyklus des Organismus exprimiert werden. Housekeeping-Gene werden bei der Analyse der Genexpression als experimentelle Kontrolle verwendet, da sie konstitutiv auf einem relativ konstanten Niveau exprimiert werden. ⓘ
Genetische und genomische Nomenklatur
Die Gennomenklatur wurde vom HUGO Gene Nomenclature Committee (HGNC), einem Ausschuss der Human Genome Organisation, für jedes bekannte menschliche Gen in Form eines genehmigten Gennamens und eines Symbols (Abkürzung) festgelegt, die über eine vom HGNC geführte Datenbank abgerufen werden können. Die Symbole werden so gewählt, dass sie eindeutig sind, und jedes Gen hat nur ein Symbol (obwohl sich die genehmigten Symbole manchmal ändern). Die Symbole werden vorzugsweise mit anderen Mitgliedern einer Genfamilie und mit Homologen in anderen Spezies übereinstimmen, insbesondere mit der Maus, da sie ein häufiger Modellorganismus ist. ⓘ
Gentechnik

Gentechnik ist die Veränderung des Genoms eines Organismus durch Biotechnologie. Seit den 1970er Jahren wurde eine Vielzahl von Techniken entwickelt, um gezielt Gene in einem Organismus hinzuzufügen, zu entfernen oder zu verändern. Bei neueren Genom-Engineering-Techniken werden künstlich hergestellte Nuklease-Enzyme eingesetzt, um gezielte DNA-Reparaturen in einem Chromosom vorzunehmen und dabei ein Gen entweder zu unterbrechen oder zu verändern, wenn der Bruch repariert wird. Der verwandte Begriff der synthetischen Biologie wird manchmal verwendet, um eine umfassende gentechnische Veränderung eines Organismus zu bezeichnen. ⓘ
Die Gentechnik ist heute ein Routinewerkzeug für die Forschung mit Modellorganismen. So lassen sich z. B. leicht Gene in Bakterien einfügen, und zur Untersuchung der Funktion eines bestimmten Gens werden Linien von Knockout-Mäusen verwendet, bei denen die Funktion des Gens gestört ist. Viele Organismen sind für Anwendungen in der Landwirtschaft, der industriellen Biotechnologie und der Medizin gentechnisch verändert worden. ⓘ
Bei mehrzelligen Organismen wird in der Regel der Embryo gentechnisch verändert, der zum erwachsenen gentechnisch veränderten Organismus heranwächst. Die Genome von Zellen in einem erwachsenen Organismus können jedoch mit gentherapeutischen Techniken bearbeitet werden, um genetische Krankheiten zu behandeln. ⓘ
Besondere Gene
Springende Gene
Sie werden auch als Transposons bezeichnet und sind mobile Erbgutabschnitte, die sich innerhalb der DNA einer Zelle frei bewegen können. Aus ihrem angestammten Ort im Erbgut schneiden sie sich selbst aus und fügen sich an einer beliebig anderen Stelle wieder ein. Biologen um Fred Gage vom Salk Institute for Biological Studies in La Jolla (USA) haben nachgewiesen, dass diese springenden Gene nicht nur wie bislang angenommen in den Zellen der Keimbahn vorkommen, sondern auch in Nerven-Vorläuferzellen aktiv sind. Forschungsergebnisse von Eric Lander et al. (2007) zeigen, dass Transposons eine wichtige Funktion haben, indem sie als kreativer Faktor im Genom wichtige genetische Innovationen rasch im Erbgut verbreiten können. ⓘ
Orphangene
Orphan-Gene sind Gene ohne nachweisbare Homologie in anderen Linien. Sie werden auch ORFans genannt, insbesondere in der mikrobiellen Literatur (mit ORF als Akronym für englisch open reading frame ‚offener Leserahmen‘). Orphan-Gene sind eine Teilmenge von taxonomisch eingeschränkten Genen, die auf einer bestimmten taxonomischen Ebene (z. B. pflanzenspezifisch) einzigartig sind. Sie gelten in der Regel als einzigartig für ein sehr schmales Taxon, sogar für eine Art (Spezies). Orphan-Gene unterscheiden sich dadurch, dass sie linienspezifisch sind und keine bekannte Geschichte der gemeinsamen Verdoppelung und Neuordnung außerhalb ihrer spezifischen Spezies oder Gruppe haben. In Menschen gibt es beispielsweise 634 Gene, die dem Schimpansen fehlen. Umgekehrt fehlen dem Menschen 780 Schimpansen-Gene. ⓘ
Typische Genomgrößen und Genanzahl
| Organismus / Biologisches System | Anzahl der Gene | Basenpaare insgesamt ⓘ |
|---|---|---|
| Gemeiner Wasserfloh | 30.907 | 2·108 |
| Acker-Schmalwand (Arabidopsis thaliana, Modellpflanze) | >25.000 | 108–1011 |
| Mensch | ~22.500 | 3·109 |
| Drosophila melanogaster (Fliege) | 12.000 | 1,6·108 |
| Backhefe (Saccharomyces cerevisiae) | 6.000 | 1,3·107 |
| Bakterium | 180–7.000 | 105−107 |
| Escherichia coli | ~5.000 | 4,65·106 |
| Carsonella ruddii | 182 | 160.000 |
| DNA-Virus | 10–300 | 5.000–200.000 |
| RNA-Virus | 1–25 | 1.000–23.000 |
| Viroid | 0 | 246–401 |