MRNA
In der Molekularbiologie ist die Boten-Ribonukleinsäure (mRNA) ein einzelsträngiges RNA-Molekül, das der genetischen Sequenz eines Gens entspricht und von einem Ribosom bei der Synthese eines Proteins abgelesen wird. ⓘ
Die mRNA entsteht bei der Transkription, bei der ein Enzym (RNA-Polymerase) das Gen in eine primäre mRNA-Transkription (auch pre-mRNA genannt) umwandelt. Diese prä-mRNA enthält in der Regel noch Introns, d. h. Regionen, die nicht für die endgültige Aminosäuresequenz kodieren. Diese werden beim Spleißen der RNA entfernt, so dass nur Exons übrig bleiben, also Regionen, die für das Protein kodieren. Diese Exon-Sequenz bildet die reife mRNA. Die reife mRNA wird dann vom Ribosom gelesen, und unter Verwendung der von der Transfer-RNA (tRNA) transportierten Aminosäuren bildet das Ribosom das Protein. Dieser Vorgang wird als Translation bezeichnet. Alle diese Prozesse sind Teil des zentralen Dogmas der Molekularbiologie, das den Fluss der genetischen Information in einem biologischen System beschreibt. ⓘ
Wie bei der DNA ist die genetische Information in der mRNA in der Abfolge der Nukleotide enthalten, die in Codons angeordnet sind, die jeweils aus drei Ribonukleotiden bestehen. Jedes Codon kodiert für eine bestimmte Aminosäure, mit Ausnahme der Stoppcodons, die die Proteinsynthese beenden. Für die Übersetzung der Codons in Aminosäuren sind zwei weitere RNA-Typen erforderlich: die Transfer-RNA, die das Codon erkennt und die entsprechende Aminosäure bereitstellt, und die ribosomale RNA (rRNA), die zentrale Komponente der Proteinproduktionsmaschinerie des Ribosoms. ⓘ
Die Idee der mRNA wurde erstmals von Sydney Brenner und Francis Crick am 15. April 1960 im King's College in Cambridge entwickelt, als François Jacob ihnen von einem Experiment berichtete, das Arthur Pardee und Jacques Monod kürzlich durchgeführt hatten. Mit Cricks Ermutigung machten sich Brenner und Jacob sofort daran, diese neue Hypothese zu testen, und sie wandten sich an Matthew Meselson vom California Institute of Technology. Im Sommer 1960 führten Brenner, Jacob und Meselson in Meselsons Labor am Caltech ein Experiment durch, das die Existenz von mRNA nachwies. Im Herbst desselben Jahres prägten Jacob und Monod den Namen "Boten-RNA" und entwickelten den ersten theoretischen Rahmen zur Erklärung ihrer Funktion. Im Februar 1961 enthüllte James Watson, dass seine Forschungsgruppe mit einem ähnlichen Experiment, das in etwa in die gleiche Richtung ging, unmittelbar hinter ihnen stand; Brenner und die anderen stimmten der Bitte von Watson zu, die Veröffentlichung ihrer Forschungsergebnisse zu verschieben. Infolgedessen wurden die Artikel von Brenner und Watson gleichzeitig in derselben Ausgabe von Nature im Mai 1961 veröffentlicht, während Jacob und Monod im selben Monat ihr theoretisches Gerüst für mRNA im Journal of Molecular Biology veröffentlichten. ⓘ
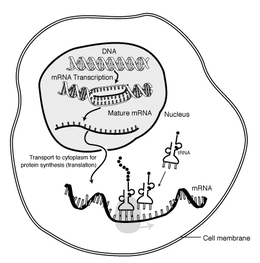
Zellen verändern Gestalt und Vorgänge durch ihre Proteine, die aus verschieden langen Ketten von bestimmten Aminosäuren in unterschiedlicher Reihenfolge bestehen. Die jeweilige Reihenfolge von Aminosäuren ist nicht zufällig, sondern wird als Bauplan vorgegeben. Die entsprechenden Vorlagen sind ein wesentlicher Teil der Erbinformation und liegen gespeichert in der Form von Desoxyribonukleinsäure (DNA) vor, jeweils in einem bestimmten DNA-Abschnitt (Gen) als Nukleotidsequenz. Beim Zugriff auf diese Information wird eine DNA-Sequenz abgelesen und in eine RNA-Sequenz umgeschrieben. Ein fertiges mRNA-Molekül trägt damit die Botschaft von der DNA – die bei Eukaryoten im Zellkern liegt – für den Proteinaufbau im Zellplasma und dient an den Ribosomen jeweils als Bauanleitung für ein bestimmtes Protein. ⓘ
Grundlegend für solche Bauanweisungen ist die Reihenfolge von Nukleinbasen (Basensequenz) in den Genabschnitten der DNA-Moleküle. Damit die Information zum Ausdruck kommen kann (Genexpression), muss sie in die Sequenz der Nukleotide von mRNA-Molekülen umgeschrieben werden (Transkription). Hierbei wird anhand der DNA-Matrize ein RNA-Strang erstellt; dies geschieht unter enzymatischer Wirkung einer RNA-Polymerase. Als Botschaft einer mRNA kann die nun in deren Basensequenz enthaltene Information an Ribosomen abgelesen und mithilfe von tRNA-Molekülen übersetzt werden (Translation). Bei dieser ribosomalen Proteinbiosynthese gibt die codierende Basensequenz der mRNA dann die Reihenfolge von Aminosäuren (Aminosäurensequenz) in der neuaufgebauten Polypeptidkette vor – und damit die Primärstruktur eines bestimmten Proteins. ⓘ
Bei Medikamenten, die auf mRNA basieren, produzieren Zellen nach der hierdurch eingebrachten Information dann das jeweils wirksame Protein. Dieses kann zum Beispiel bei mRNA-Impfstoffen als Antigen wirken. ⓘ
Synthese, Verarbeitung und Funktion

Die kurze Existenz eines mRNA-Moleküls beginnt mit der Transkription und endet schließlich mit dem Abbau. Im Laufe seines Lebens kann ein mRNA-Molekül auch verarbeitet, bearbeitet und transportiert werden, bevor es übersetzt wird. Eukaryotische mRNA-Moleküle erfordern häufig eine umfangreiche Verarbeitung und einen umfangreichen Transport, während dies bei prokaryotischen mRNA-Molekülen nicht der Fall ist. Ein Molekül eukaryontischer mRNA und die es umgebenden Proteine werden zusammen als Boten-RNP bezeichnet. ⓘ
Transkription
Bei der Transkription wird die RNA von der DNA kopiert. Während der Transkription erstellt die RNA-Polymerase je nach Bedarf eine Kopie eines Gens von der DNA zur mRNA. Dieser Prozess unterscheidet sich bei Eukaryoten und Prokaryoten geringfügig. Ein bemerkenswerter Unterschied besteht darin, dass die prokaryotische RNA-Polymerase während der Transkription mit DNA-verarbeitenden Enzymen assoziiert, so dass die Verarbeitung während der Transkription stattfinden kann. Dies führt dazu, dass der neue mRNA-Strang doppelsträngig wird, indem ein komplementärer Strang, der so genannte tRNA-Strang, gebildet wird, der in seiner Kombination nicht in der Lage ist, Strukturen aus Basenpaarungen zu bilden. Außerdem ist die Vorlage für die mRNA der komplementäre tRNA-Strang, der in seiner Sequenz mit der Anticodon-Sequenz identisch ist, an die die DNA bindet. Das kurzlebige, unverarbeitete oder teilweise verarbeitete Produkt wird als Vorläufer-mRNA oder prä-mRNA bezeichnet; nach vollständiger Verarbeitung wird es als reife mRNA bezeichnet. ⓘ
Eukaryotische prä-mRNA-Verarbeitung

Die Verarbeitung von mRNA ist bei Eukaryoten, Bakterien und Archaeen sehr unterschiedlich. Nicht-eukaryotische mRNA ist im Wesentlichen bei der Transkription ausgereift und bedarf, außer in seltenen Fällen, keiner Verarbeitung. Eukaryontische prä-mRNA hingegen erfordert mehrere Verarbeitungsschritte, bevor sie in das Zytoplasma transportiert und vom Ribosom übersetzt wird. ⓘ
Spleißen
Die umfangreiche Verarbeitung der eukaryotischen prä-mRNA, die zur reifen mRNA führt, ist das RNA-Spleißen, ein Mechanismus, bei dem Introns oder Outrons (nicht codierende Regionen) entfernt und Exons (codierende Regionen) zusammengefügt werden. ⓘ
5'-Cap-Zusatz

Eine 5'-Kappe (auch als RNA-Kappe, RNA-7-Methylguanosin-Kappe oder RNA-m7G-Kappe bezeichnet) ist ein modifiziertes Guanin-Nukleotid, das kurz nach Beginn der Transkription an das "vordere" oder 5'-Ende einer eukaryotischen Boten-RNA angefügt wurde. Die 5'-Kappe besteht aus einem endständigen 7-Methylguanosin-Rest, der über eine 5'-5'-Triphosphatbindung mit dem ersten transkribierten Nukleotid verbunden ist. Ihr Vorhandensein ist entscheidend für die Erkennung durch das Ribosom und den Schutz vor RNasen. ⓘ
Die Cap-Addition ist an die Transkription gekoppelt und erfolgt kotranskriptiv, d. h., beide beeinflussen sich gegenseitig. Kurz nach Beginn der Transkription wird das 5'-Ende der zu synthetisierenden mRNA von einem mit der RNA-Polymerase assoziierten Cap-Synthesekomplex gebunden. Dieser enzymatische Komplex katalysiert die chemischen Reaktionen, die für die Verkappung der mRNA erforderlich sind. Die Synthese verläuft als mehrstufige biochemische Reaktion. ⓘ
Bearbeitung
In einigen Fällen wird eine mRNA editiert, wobei die Nukleotidzusammensetzung dieser mRNA verändert wird. Ein Beispiel beim Menschen ist die Apolipoprotein-B-mRNA, die in einigen Geweben editiert wird, in anderen jedoch nicht. Durch die Editierung entsteht ein frühes Stoppcodon, das bei der Translation ein kürzeres Protein erzeugt. ⓘ
Polyadenylierung

Polyadenylierung ist die kovalente Verknüpfung eines Polyadenylyl-Anteils mit einem Boten-RNA-Molekül. In eukaryotischen Organismen sind die meisten Boten-RNA-Moleküle (mRNA) am 3'-Ende polyadenyliert, aber neuere Studien haben gezeigt, dass auch kurze Abschnitte mit Uridin (Oligouridylierung) üblich sind. Der Poly(A)-Schwanz und das daran gebundene Protein tragen dazu bei, die mRNA vor dem Abbau durch Exonukleasen zu schützen. Die Polyadenylierung ist auch für die Beendigung der Transkription, den Export der mRNA aus dem Zellkern und die Translation wichtig. Auch in prokaryotischen Organismen kann mRNA polyadenyliert werden, wobei die Poly(A)-Schwänze den exonukleolytischen Abbau eher erleichtern als behindern. ⓘ
Die Polyadenylierung erfolgt während und/oder unmittelbar nach der Transkription der DNA in RNA. Nach Beendigung der Transkription wird die mRNA-Kette durch einen mit der RNA-Polymerase assoziierten Endonuklease-Komplex gespalten. Nach der Spaltung der mRNA werden etwa 250 Adenosinreste an das freie 3'-Ende an der Spaltstelle angefügt. Diese Reaktion wird von der Polyadenylat-Polymerase katalysiert. Genau wie beim alternativen Spleißen kann es mehr als eine Polyadenylierungsvariante einer mRNA geben. ⓘ
Auch Mutationen der Polyadenylierungsstelle kommen vor. Das primäre RNA-Transkript eines Gens wird an der Poly-A-Additionsstelle gespalten, und 100-200 A's werden an das 3'-Ende der RNA angefügt. Wenn diese Stelle verändert ist, entsteht ein abnorm langes und instabiles mRNA-Konstrukt. ⓘ
Transport
Ein weiterer Unterschied zwischen Eukaryoten und Prokaryoten ist der mRNA-Transport. Da bei Eukaryoten Transkription und Translation kompartimentell getrennt sind, müssen eukaryotische mRNAs aus dem Zellkern in das Zytoplasma exportiert werden - ein Prozess, der durch verschiedene Signalwege reguliert werden kann. Reife mRNAs werden an ihren prozessierten Modifikationen erkannt und dann durch die Kernpore exportiert, indem sie an die cap-bindenden Proteine CBP20 und CBP80 sowie an den Transkriptions-/Exportkomplex (TREX) gebunden werden. In Eukaryonten sind mehrere mRNA-Exportwege identifiziert worden. ⓘ
In räumlich komplexen Zellen werden einige mRNAs zu bestimmten subzellulären Zielen transportiert. In reifen Neuronen werden bestimmte mRNA vom Soma zu den Dendriten transportiert. Ein Ort der mRNA-Translation befindet sich an Polyribosomen, die selektiv unterhalb von Synapsen lokalisiert sind. Die mRNA für Arc/Arg3.1 wird durch synaptische Aktivität induziert und lokalisiert sich selektiv in der Nähe aktiver Synapsen auf der Grundlage von Signalen, die von NMDA-Rezeptoren erzeugt werden. Auch andere mRNAs wandern als Reaktion auf äußere Reize in die Dendriten, wie z. B. β-Actin-mRNA. Nach dem Export aus dem Zellkern assoziiert die Aktin-mRNA mit ZBP1 und der 40S-Untereinheit. Der Komplex wird von einem Motorprotein gebunden und entlang des Zytoskeletts zum Zielort (Neuritenverlängerung) transportiert. Schließlich wird ZBP1 von Src phosphoryliert, damit die Translation eingeleitet werden kann. In sich entwickelnden Neuronen werden mRNAs auch in wachsende Axone und insbesondere Wachstumskegel transportiert. Viele mRNAs sind mit sogenannten "Zip-Codes" markiert, die ihren Transport an einen bestimmten Ort lenken. ⓘ
Übersetzung

Da prokaryotische mRNA nicht verarbeitet oder transportiert werden muss, kann die Übersetzung durch das Ribosom unmittelbar nach dem Ende der Transkription beginnen. Daher kann man sagen, dass die prokaryotische Translation an die Transkription gekoppelt ist und kotranskriptiv erfolgt. ⓘ
Eukaryotische mRNA, die verarbeitet und ins Zytoplasma transportiert wurde (d. h. reife mRNA), kann dann vom Ribosom translatiert werden. Die Translation kann an Ribosomen erfolgen, die frei im Zytoplasma schwimmen, oder durch das Signalerkennungspartikel zum endoplasmatischen Retikulum geleitet werden. Anders als bei Prokaryonten ist die Translation bei Eukaryonten also nicht direkt an die Transkription gekoppelt. In manchen Zusammenhängen ist es sogar möglich, dass verringerte mRNA-Spiegel mit erhöhten Proteinspiegeln einhergehen, wie dies für die mRNA/Proteinspiegel von EEF1A1 bei Brustkrebs beobachtet wurde. ⓘ
Struktur

Kodierende Regionen
Kodierende Regionen bestehen aus Codons, die vom Ribosom entschlüsselt und in Proteine übersetzt werden; bei Eukaryoten in der Regel in ein Codon und bei Prokaryoten in mehrere. Kodierende Regionen beginnen mit dem Startcodon und enden mit einem Stoppcodon. Im Allgemeinen ist das Startcodon ein AUG-Triplett und das Stoppcodon ist UAG ("Amber"), UAA ("Ocker") oder UGA ("Opal"). Die kodierenden Regionen werden in der Regel durch interne Basenpaare stabilisiert; dies erschwert den Abbau. Zusätzlich zur Proteincodierung können Teile der kodierenden Regionen als regulatorische Sequenzen in der prä-mRNA als exonische Spleißverstärker oder exonische Spleißsilencer dienen. ⓘ
Untranslatierte Regionen

Untranslatierte Regionen (UTRs) sind Abschnitte der mRNA vor dem Startcodon und nach dem Stoppcodon, die nicht translatiert werden. Sie werden als five prime untranslated region (5' UTR) bzw. three prime untranslated region (3' UTR) bezeichnet. Diese Regionen werden zusammen mit der kodierenden Region transkribiert und sind somit exonisch, da sie in der reifen mRNA vorhanden sind. Den untranslatierten Regionen werden mehrere Funktionen bei der Genexpression zugeschrieben, darunter die Stabilität der mRNA, die Lokalisierung der mRNA und die Effizienz der Translation. Die Fähigkeit einer UTR, diese Funktionen zu erfüllen, hängt von der Sequenz der UTR ab und kann sich zwischen verschiedenen mRNAs unterscheiden. Genetische Varianten in der 3'-UTR wurden auch mit Krankheitsanfälligkeit in Verbindung gebracht, da sie die RNA-Struktur und die Protein-Translation verändern. ⓘ
Die Stabilität von mRNAs kann durch den 5'-UTR und/oder den 3'-UTR aufgrund der unterschiedlichen Affinität für RNA-abbauende Enzyme, so genannte Ribonukleasen, und für zusätzliche Proteine, die den RNA-Abbau fördern oder hemmen können, kontrolliert werden. (Siehe auch: C-reiches Stabilitätselement.) ⓘ
Die Translationseffizienz, manchmal auch die vollständige Hemmung der Translation, kann durch UTRs gesteuert werden. Proteine, die entweder an den 3'- oder 5'-UTR binden, können die Translation beeinflussen, indem sie die Fähigkeit des Ribosoms, an die mRNA zu binden, beeinträchtigen. MikroRNAs, die an den 3'-UTR gebunden sind, können ebenfalls die Translationseffizienz oder die mRNA-Stabilität beeinflussen. ⓘ
Es wird angenommen, dass die zytoplasmatische Lokalisierung der mRNA eine Funktion der 3'-UTR ist. Proteine, die in einer bestimmten Region der Zelle benötigt werden, können auch dort translatiert werden; in einem solchen Fall kann die 3'-UTR Sequenzen enthalten, die es ermöglichen, dass das Transkript zur Translation in dieser Region lokalisiert wird. ⓘ
Einige der in den untranslatierten Regionen enthaltenen Elemente bilden bei der Transkription in RNA eine charakteristische Sekundärstruktur. Diese strukturellen mRNA-Elemente sind an der Regulierung der mRNA beteiligt. Einige, wie das SECIS-Element, sind Ziele für die Bindung von Proteinen. Eine Klasse von mRNA-Elementen, die Riboschalter, binden direkt kleine Moleküle und verändern deren Faltung, um die Transkriptions- oder Translationsraten zu verändern. In diesen Fällen reguliert sich die mRNA selbst. ⓘ
Poly(A)-Schwanz
Der 3'-Poly(A)-Schwanz ist eine lange Sequenz von Adenin-Nukleotiden (oft mehrere hundert), die an das 3'-Ende der prä-mRNA angehängt ist. Dieser Schwanz fördert den Export aus dem Zellkern und die Translation und schützt die mRNA vor dem Abbau. ⓘ
Monocistronische versus polycistronische mRNA
Ein mRNA-Molekül wird als monokistron bezeichnet, wenn es die genetische Information zur Übersetzung nur einer einzigen Proteinkette (Polypeptid) enthält. Dies ist bei den meisten eukaryotischen mRNAs der Fall. Polycistronische mRNA hingegen enthält mehrere offene Leserahmen (ORFs), von denen jeder in ein Polypeptid übersetzt wird. Diese Polypeptide haben in der Regel eine verwandte Funktion (sie sind oft die Untereinheiten, aus denen sich ein komplexes Endprotein zusammensetzt), und ihre kodierende Sequenz wird in einer regulatorischen Region gruppiert und reguliert, die einen Promotor und einen Operator enthält. Die meisten der in Bakterien und Archaeen vorkommenden mRNAs sind polycistronisch, so auch das menschliche mitochondriale Genom. Dicistronische oder bicistronische mRNA kodiert nur zwei Proteine. ⓘ
mRNA-Zirkularisierung

In Eukaryonten bilden mRNA-Moleküle zirkuläre Strukturen aufgrund einer Interaktion zwischen dem eIF4E und dem Poly(A)-bindenden Protein, die beide an eIF4G binden und eine mRNA-Protein-mRNA-Brücke bilden. Man geht davon aus, dass die Zirkularisierung den Zyklus der Ribosomen auf der mRNA fördert, was zu einer zeiteffizienten Translation führt, und möglicherweise auch dafür sorgt, dass nur intakte mRNA translatiert werden (teilweise abgebaute mRNA haben typischerweise keine m7G-Kappe oder keinen Poly-A-Schwanz). ⓘ
Es gibt noch andere Mechanismen für die Zirkularisierung, insbesondere bei Virus-mRNA. Die mRNA des Poliovirus verwendet einen Kleeblattabschnitt an ihrem 5'-Ende, um PCBP2 zu binden, das wiederum Poly(A)-bindendes Protein bindet, wodurch der bekannte mRNA-Protein-mRNA-Kreis entsteht. Das Gerstengelbverzwergungsvirus besitzt Bindungen zwischen mRNA-Segmenten an seinem 5'- und 3'-Ende (so genannte kissing stem loops), die die mRNA ohne Beteiligung von Proteinen zirkulieren lassen. ⓘ
Auch die Genome von RNA-Viren (deren + Stränge als mRNA übersetzt werden) sind häufig zirkularisiert. Während der Genomreplikation erhöht die Zirkularisierung die Geschwindigkeit der Genomreplikation, indem sie die virale RNA-abhängige RNA-Polymerase ähnlich wie das Ribosom in Bewegung hält. ⓘ
Abbau
Verschiedene mRNAs innerhalb derselben Zelle haben unterschiedliche Lebensdauern (Stabilitäten). In Bakterienzellen können einzelne mRNAs von Sekunden bis zu mehr als einer Stunde überleben. Die durchschnittliche Lebensdauer liegt jedoch zwischen 1 und 3 Minuten, wodurch bakterielle mRNA viel weniger stabil ist als eukaryotische mRNA. In Säugetierzellen reicht die Lebensdauer der mRNA von einigen Minuten bis zu Tagen. Je größer die Stabilität einer mRNA ist, desto mehr Protein kann von dieser mRNA produziert werden. Die begrenzte Lebensdauer der mRNA ermöglicht es einer Zelle, die Proteinsynthese als Reaktion auf ihre wechselnden Bedürfnisse schnell zu verändern. Es gibt viele Mechanismen, die zur Zerstörung einer mRNA führen, von denen einige im Folgenden beschrieben werden. ⓘ
Prokaryotischer mRNA-Abbau
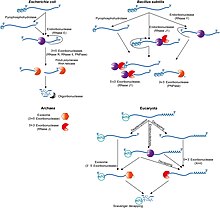
Im Allgemeinen ist die Lebensdauer von mRNA in Prokaryonten viel kürzer als in Eukaryonten. Prokaryonten bauen Nachrichten durch eine Kombination von Ribonukleasen, einschließlich Endonukleasen, 3'-Exonukleasen und 5'-Exonukleasen ab. In einigen Fällen können kleine RNA-Moleküle (sRNA) mit einer Länge von zehn bis hunderten von Nukleotiden den Abbau spezifischer mRNAs stimulieren, indem sie Basenpaare mit komplementären Sequenzen bilden und die Ribonuklease-Spaltung durch RNase III erleichtern. Kürzlich wurde gezeigt, dass Bakterien auch eine Art 5'-Kappe besitzen, die aus einem Triphosphat am 5'-Ende besteht. Durch die Entfernung von zwei der Phosphate bleibt ein 5'-Monophosphat übrig, wodurch die Botschaft durch die Exonuklease RNase J zerstört wird, die 5' zu 3' abbaut. ⓘ
Eukaryotischer mRNA-Umsatz
In eukaryotischen Zellen besteht ein Gleichgewicht zwischen den Prozessen der Translation und des mRNA-Zerfalls. Nachrichten, die aktiv übersetzt werden, werden von Ribosomen, den eukaryotischen Initiationsfaktoren eIF-4E und eIF-4G und dem Poly(A)-bindenden Protein gebunden. eIF-4E und eIF-4G blockieren das Decapping-Enzym (DCP2), und das Poly(A)-bindende Protein blockiert den Exosomenkomplex und schützt so die Enden der Nachricht. Das Gleichgewicht zwischen Translation und Zerfall spiegelt sich in der Größe und Häufigkeit der zytoplasmatischen Strukturen wider, die als P-Körper bekannt sind. Der Poly(A)-Schwanz der mRNA wird durch spezialisierte Exonukleasen verkürzt, die durch eine Kombination von cis-regulierenden Sequenzen auf der RNA und trans-wirkenden RNA-bindenden Proteinen auf bestimmte Boten-RNAs ausgerichtet sind. Es wird angenommen, dass die Entfernung des Poly(A)-Schwanzes die zirkuläre Struktur der Nachricht stört und den Cap-Bindungskomplex destabilisiert. Die Nachricht wird dann entweder durch den Exosom-Komplex oder den Decapping-Komplex abgebaut. Auf diese Weise können translatorisch inaktive Botschaften schnell zerstört werden, während aktive Botschaften intakt bleiben. Der Mechanismus, durch den die Translation gestoppt und die Nachricht an die Zerfallskomplexe weitergegeben wird, ist nicht im Detail bekannt. ⓘ
Zerfall von AU-reichen Elementen
Das Vorhandensein von AU-reichen Elementen in einigen mRNAs von Säugetieren destabilisiert diese Transkripte durch die Wirkung von zellulären Proteinen, die diese Sequenzen binden und die Entfernung des Poly(A)-Schwanzes anregen. Es wird angenommen, dass der Verlust des Poly(A)-Schwanzes den mRNA-Abbau fördert, indem er den Angriff sowohl durch den Exosomenkomplex als auch durch den Decapping-Komplex erleichtert. Der rasche mRNA-Abbau über AU-reiche Elemente ist ein entscheidender Mechanismus zur Verhinderung der Überproduktion potenter Zytokine wie Tumor-Nekrose-Faktor (TNF) und Granulozyten-Makrophagen-Kolonie-stimulierender Faktor (GM-CSF). AU-reiche Elemente regulieren auch die Biosynthese von proto-onkogenen Transkriptionsfaktoren wie c-Jun und c-Fos. ⓘ
Nonsense-vermittelter Zerfall
Eukaryotische Nachrichten unterliegen der Überwachung durch den Nonsense-vermittelten Zerfall (NMD), der das Vorhandensein von vorzeitigen Stoppcodons (Nonsense-Codons) in der Nachricht überprüft. Diese können durch unvollständiges Spleißen, V(D)J-Rekombination im adaptiven Immunsystem, Mutationen in der DNA, Transkriptionsfehler, undichtes Scannen durch das Ribosom, das eine Rahmenverschiebung verursacht, und andere Ursachen entstehen. Die Entdeckung eines vorzeitigen Stoppcodons löst den Abbau der mRNA durch 5'-Entkappung, Entfernung des 3'-Poly(A)-Schwanzes oder endonukleolytische Spaltung aus. ⓘ
Kleine interferierende RNA (siRNA)
In Metazoen werden kleine interferierende RNAs (siRNAs), die von Dicer verarbeitet werden, in einen Komplex eingebaut, der als RNA-induzierter Silencing-Komplex oder RISC bekannt ist. Dieser Komplex enthält eine Endonuklease, die perfekt komplementäre Botschaften spaltet, an die die siRNA bindet. Die so entstandenen mRNA-Fragmente werden dann durch Exonukleasen zerstört. siRNA wird häufig in Labors verwendet, um die Funktion von Genen in Zellkulturen zu blockieren. Man geht davon aus, dass sie Teil des angeborenen Immunsystems zur Abwehr von doppelsträngigen RNA-Viren ist. ⓘ
MikroRNA (miRNA)
MicroRNAs (miRNAs) sind kleine RNAs, die in der Regel teilweise komplementär zu Sequenzen in Boten-RNAs von Metazoen sind. Die Bindung einer miRNA an eine Nachricht kann die Übersetzung dieser Nachricht unterdrücken und die Entfernung des Poly(A)-Schwanzes beschleunigen, wodurch der mRNA-Abbau beschleunigt wird. Der Wirkmechanismus von miRNAs ist Gegenstand aktiver Forschung. ⓘ
Andere Zerfallsmechanismen
Es gibt noch andere Wege, auf denen Botschaften abgebaut werden können, darunter der Non-Stop-Zerfall und das Silencing durch Piwi-interacting RNA (piRNA), um nur einige zu nennen. ⓘ
Anwendungen
Die Verabreichung einer nukleosidmodifizierten Boten-RNA-Sequenz kann eine Zelle dazu veranlassen, ein Protein zu produzieren, das wiederum direkt eine Krankheit behandeln oder als Impfstoff dienen könnte; indirekter könnte das Protein eine körpereigene Stammzelle dazu bringen, sich in der gewünschten Weise zu differenzieren. ⓘ
Die größte Herausforderung bei der RNA-Therapie besteht darin, die RNA zu den entsprechenden Zellen zu bringen. Zu den Herausforderungen gehört die Tatsache, dass nackte RNA-Sequenzen nach ihrer Herstellung auf natürliche Weise abgebaut werden; sie können das körpereigene Immunsystem dazu veranlassen, sie als Eindringling anzugreifen; und sie sind für die Zellmembran undurchlässig. Sobald sie in der Zelle sind, müssen sie den Transportmechanismus der Zelle verlassen, um im Zytoplasma zu wirken, in dem sich die notwendigen Ribosomen befinden. ⓘ
Um diese Herausforderungen zu überwinden, wurde mRNA als Therapeutikum erstmals 1989 vorgeschlagen, "nachdem eine breit anwendbare In-vitro-Transfektionstechnik entwickelt worden war". In den 1990er Jahren wurden mRNA-Impfstoffe für personalisierte Krebserkrankungen entwickelt, die auf nicht nukleosidisch modifizierter mRNA beruhen. mRNA-basierte Therapien werden weiterhin als Behandlungs- oder Therapiemethode sowohl für Krebs als auch für Autoimmun-, Stoffwechsel- und Entzündungskrankheiten der Atemwege untersucht. Auch Gene-Editing-Therapien wie CRISPR können von der Verwendung von mRNA profitieren, um Zellen zur Herstellung des gewünschten Cas-Proteins zu veranlassen. ⓘ
Seit den 2010er Jahren gelten RNA-Impfstoffe und andere RNA-Therapeutika als "eine neue Klasse von Medikamenten". Die ersten mRNA-basierten Impfstoffe erhielten eine eingeschränkte Zulassung und wurden beispielsweise während der COVID-19-Pandemie von Pfizer-BioNTech COVID-19-Impfstoff und Moderna weltweit eingeführt. ⓘ
Translation
Bei der Translation wird die codierende Sequenz von Nukleinbasen der mRNA-Nukleotide in die Aminosäuresequenz der Polypeptidkette eines Proteins übersetzt (translatiert). Die Nukleotidsequenz eines Offenen Leserahmens wird hierbei in Dreierschritten abgelesen und jedem Basentriplett mittels tRNA-Molekülen jeweils eine bestimmte Aminosäure zugeordnet sowie diese mit der vorangegangenen über eine Peptidbindung verknüpft. Dieser Prozess findet an den Ribosomen im Cytoplasma statt und stellt die eigentliche Proteinbiosynthese dar. Bei eukaryotischen Zellen können die Ribosomen frei vorliegen oder sich der Membran des Endoplasmatischen Retikulums anheften. ⓘ
Jedes Ribosom, das eine mRNA erkennt und an diese bindet, translatiert dann die codierende Nukleotidsequenz der mRNA gemäß dem genetischen Code in die entsprechende Aminosäurensequenz eines Proteins. Die hierfür nötigen Aminosäuremoleküle werden von tRNA-Molekülen herangetragen und dem Cytoplasma der Zelle entnommen. Während eine prokaryotische mRNA oft mehrere codierende Abschnitte enthält (polygenische mRNA), sind eukaryotische mRNAs gewöhnlich monocistronisch und enthalten so nur einen Abschnitt mit codierender Sequenz. ⓘ
Ein Ribosom übersetzt jeweils nur eine mRNA und baut nach deren Vorgabe das codierte Polypeptid einmal auf. Danach löst sich das Ribosom von der mRNA. An eine mRNA können sich aber mehrere Ribosomen zugleich anlagern und jeweils eine Polypeptidkette synthetisieren. Ebenso kann eine mRNA mehrmals nacheinander ribosomal abgelesen werden. Die Anzahl der gebildeten Proteinmoleküle hängt also von der Zahl an durchgeführten Translationsvorgängen ab. Je länger ein mRNA-Molekül existiert, desto häufiger kann es hierfür benutzt werden. ⓘ
Degradation
Später wird die mRNA durch eine Ribonuklease (RNase) enzymatisch abgebaut und in ihre Nukleotide zerlegt, die dann wieder zum Aufbau neuer RNA-Moleküle genutzt werden können. Mit dieser Zersetzung, auch Degradation genannt, endet die Lebensdauer eines mRNA-Moleküls. Die Dauer der Verfügbarkeit bis zum Abbau durch Nukleasen in der Zelle kann jedoch verschieden sein und ist für eine Regulation der Proteinbiosynthese bedeutsam. Der Abbauprozess findet in eukaryotischen Zellen oft in spezifischen Strukturen statt, den sogenannten P-bodies im Cytoplasma. Da hier mRNA auch – statt abgebaut zu werden – temporär zwischengelagert werden kann für eine erneute Translation, sind damit zusätzliche Regulationsweisen möglich. ⓘ
Reinigung und Nachweis
RNA kann durch eine RNA-Reinigung isoliert werden, z. B. bei RNA mit einem Poly-A-Schwanz durch Verwendung einer Oligo-dT-Säule. Der Nachweis erfolgt per Northern Blot oder nach einer RT-PCR durch eine qPCR oder eine DNA-Sequenzierung. ⓘ
mRNA-Impfstoffe
Da mRNA bereits in kurzer Zeit in den Wirtszellen enzymatisch abgebaut wird und außerdem über die Aktivierung von Toll-like-Rezeptoren zu einer Zytokin-Ausschüttung mit unerwünschten Wirkungen führen kann, wurde zunächst nur zögerlich an Medikamenten auf RNA-Basis geforscht. ⓘ
Bei der Anwendung als Impfstoff wird eine mRNA verabreicht, die ein ausgewähltes Protein codiert. In den Zellen des Impflings wird anhand der aufgenommenen mRNA dieses Protein ribosomal synthetisiert. Anschließend kann es auf der Zelloberfläche extrazellulär dem Immunsystem präsentiert werden und als Antigen wirken. In der Folge können darüber unterschiedliche Immunantworten ausgelöst werden. Dieses Verfahren kann für verschiedene Zwecke eingesetzt werden, zum Beispiel in der Krebstherapie sowie für Influenzaimpfstoffe und für Tollwutimpfstoffe. ⓘ
Verwendet man statt normaler mRNA hingegen modRNA (nukleosid-modifizierte mRNA), lassen sich der rasche enzymatische Abbau und die Aktivierung von Toll-like-Rezeptoren verlangsamen bzw. verhindern, was die Entwicklung von Impfstoffen auf mRNA-Basis massiv beschleunigte. ⓘ
Siehe auch
Literatur
- James E. Darnell, Harvey Lodish, David Baltimore: Molekulare Zellbiologie. de Gruyter, Berlin u. a. 1993, ISBN 3-11-011934-X (4. Auflage. Harvey Lodish: Molekulare Zellbiologie. Spektrum Akademischer Verlag, Heidelberg u. a. 2001, ISBN 3-8274-1077-0).
- Benjamin Lewin: Molekularbiologie der Gene. Spektrum Akademischer Verlag, Heidelberg u. a. 1998, ISBN 3-8274-0234-4.
- William S. Klug, Michael R. Cummings, Charlotte A. Spencer: Genetik. 8., aktualisierte Auflage 2007, ISBN 978-3-8273-7247-5.
- G. Haimovich, D. A. Medina u. a.: Gene expression is circular: factors for mRNA degradation also foster mRNA synthesis. In: Cell. Band 153, Nummer 5, Mai 2013, S. 1000–1011, doi:10.1016/j.cell.2013.05.012. PMID 23706738.
- Moritz Schmidt, Florian Kluge, Felix Sandmeir, Uwe Kühn, Peter Schäfer, Christian Tüting, Christian Ihling, Elena Conti, Elmar Wahle: Reconstitution of 3′ end processing of mammalian pre-mRNA reveals a central role of RBBP6. In: CSH Press: Genes & Development, Band 36, Nr. 5–6, 1. März 2022, S. 195–209; doi:10.1101/gad.349217.121, Epub 17. Februar 2022. Dazu:
Bauanleitung für Proteine: Wie die mRNA ihre finale Form bekommt. Auf: EurekAlert! vom 21. Februar 2022. Quelle: Martin-Luther-Universität Halle-Wittenberg. ⓘ
Dokumentationen
- mRNA − Hype oder Hoffnung? (Wissen Hoch 2) TV-Dokumentation in HD von Larissa Klinker und Tina Roth, ZDF/ 3sat, D 2022 (Auf: youtube.com). ⓘ