DNA-Sequenzierung
| Teil einer Serie über ⓘ |
| Genetik |
|---|
 |
|
Unter DNA-Sequenzierung versteht man den Prozess der Bestimmung der Nukleinsäuresequenz - der Reihenfolge der Nukleotide in der DNA. Sie umfasst jede Methode oder Technologie, die zur Bestimmung der Reihenfolge der vier Basen Adenin, Guanin, Cytosin und Thymin verwendet wird. Das Aufkommen von schnellen DNA-Sequenzierungsmethoden hat die biologische und medizinische Forschung und Entdeckung stark beschleunigt. ⓘ
Die Kenntnis von DNA-Sequenzen ist für die biologische Grundlagenforschung, für DNA-genografische Projekte und in zahlreichen Anwendungsbereichen wie medizinische Diagnose, Biotechnologie, forensische Biologie, Virologie und biologische Systematik unverzichtbar geworden. Durch den Vergleich gesunder und mutierter DNA-Sequenzen können verschiedene Krankheiten, einschließlich verschiedener Krebsarten, diagnostiziert, das Antikörperrepertoire charakterisiert und die Behandlung von Patienten gesteuert werden. Die Möglichkeit, DNA-Sequenzen schnell zu sequenzieren, ermöglicht eine schnellere und individuellere medizinische Versorgung und die Identifizierung und Katalogisierung von mehr Organismen. ⓘ
Die mit der modernen DNA-Sequenzierungstechnologie erreichte hohe Sequenziergeschwindigkeit hat zur Sequenzierung vollständiger DNA-Sequenzen oder Genome zahlreicher Lebensformen und -arten beigetragen, darunter das menschliche Genom und andere vollständige DNA-Sequenzen vieler Tier-, Pflanzen- und Mikrobenarten.

Die ersten DNA-Sequenzen wurden in den frühen 1970er Jahren von akademischen Forschern mit mühsamen Methoden auf der Grundlage der zweidimensionalen Chromatographie gewonnen. Seit der Entwicklung fluoreszenzbasierter Sequenzierungsmethoden mit einem DNA-Sequenzer ist die DNA-Sequenzierung einfacher und um Größenordnungen schneller geworden. ⓘ
Anwendungen
Die DNA-Sequenzierung kann zur Bestimmung der Sequenz einzelner Gene, größerer genetischer Regionen (d. h. Gencluster oder Operons), ganzer Chromosomen oder ganzer Genome eines beliebigen Organismus verwendet werden. Die DNA-Sequenzierung ist auch die effizienteste Methode zur indirekten Sequenzierung von RNA oder Proteinen (über deren offene Leserahmen). Die DNA-Sequenzierung ist zu einer Schlüsseltechnologie in vielen Bereichen der Biologie und anderer Wissenschaften wie Medizin, Forensik und Anthropologie geworden. ⓘ
Molekularbiologie
Die Sequenzierung wird in der Molekularbiologie eingesetzt, um Genome und die von ihnen kodierten Proteine zu untersuchen. Die mit Hilfe der Sequenzierung gewonnenen Informationen ermöglichen es den Forschern, Veränderungen in Genen und Zusammenhänge mit Krankheiten und Phänotypen zu erkennen und potenzielle Angriffspunkte für Medikamente zu identifizieren. ⓘ
Evolutionsbiologie
Da es sich bei der DNA um ein informatives Makromolekül handelt, das von einer Generation zur nächsten weitergegeben werden kann, wird die DNA-Sequenzierung in der Evolutionsbiologie eingesetzt, um zu untersuchen, wie verschiedene Organismen miteinander verwandt sind und wie sie sich entwickelt haben. Im Februar 2021 berichteten Wissenschaftler erstmals über die Sequenzierung von DNA aus tierischen Überresten, in diesem Fall von einem Mammut, das über eine Million Jahre alt ist - die älteste bisher sequenzierte DNA. ⓘ
Metagenomik
Die Metagenomik befasst sich mit der Identifizierung von Organismen in Gewässern, Abwässern, Schmutz, aus der Luft gefilterten Rückständen oder Tupferproben von Organismen. Zu wissen, welche Organismen in einer bestimmten Umgebung vorkommen, ist entscheidend für die Forschung in den Bereichen Ökologie, Epidemiologie, Mikrobiologie und anderen Bereichen. Mit Hilfe der Sequenzierung können Forscher zum Beispiel feststellen, welche Arten von Mikroben in einem Mikrobiom vorhanden sind. ⓘ
Virologie
Da die meisten Viren zu klein sind, um mit einem Lichtmikroskop gesehen zu werden, ist die Sequenzierung eines der wichtigsten Instrumente in der Virologie zur Identifizierung und Untersuchung des Virus. Virale Genome können auf DNA oder RNA basieren. RNA-Viren sind für die Genomsequenzierung zeitempfindlicher, da sie in klinischen Proben schneller abgebaut werden. Die herkömmliche Sanger-Sequenzierung und die Sequenzierung der nächsten Generation werden für die Sequenzierung von Viren in der Grundlagen- und klinischen Forschung sowie für die Diagnose neu auftretender Virusinfektionen, die molekulare Epidemiologie viraler Krankheitserreger und die Prüfung auf Arzneimittelresistenz eingesetzt. Es gibt mehr als 2,3 Millionen einzigartige virale Sequenzen in GenBank. In letzter Zeit hat NGS die traditionelle Sanger-Methode als beliebteste Methode zur Erzeugung von Virusgenomen abgelöst. ⓘ
Während des Ausbruchs der Geflügelpest im Jahr 1990 wurde durch Sequenzierung des Virus festgestellt, dass der Influenza-Subtyp durch Reassortierung zwischen Wachteln und Geflügel entstanden ist. Dies führte in Hongkong zu einer Gesetzgebung, die den gemeinsamen Verkauf von lebenden Wachteln und Geflügel auf dem Markt verbot. Die Virensequenzierung kann auch dazu verwendet werden, den Zeitpunkt des Beginns eines Virusausbruchs mit Hilfe einer molekularen Uhr abzuschätzen. ⓘ
Medizin
Medizintechniker können Gene (oder theoretisch ganze Genome) von Patienten sequenzieren, um festzustellen, ob ein Risiko für genetische Krankheiten besteht. Dies ist eine Form von Gentests, auch wenn einige Gentests keine DNA-Sequenzierung beinhalten. ⓘ
Die DNA-Sequenzierung wird auch zunehmend zur Diagnose und Behandlung seltener Krankheiten eingesetzt. Da immer mehr Gene identifiziert werden, die seltene genetische Krankheiten verursachen, werden molekulare Diagnosen für Patienten immer wichtiger. Mit Hilfe der DNA-Sequenzierung können Kliniker genetisch bedingte Krankheiten erkennen, das Krankheitsmanagement verbessern, Fortpflanzungsberatung anbieten und wirksamere Therapien entwickeln. ⓘ
Außerdem kann die DNA-Sequenzierung bei der Bestimmung spezifischer Bakterien nützlich sein, um eine präzisere Antibiotikabehandlung zu ermöglichen und so das Risiko der Entstehung einer Antibiotikaresistenz in Bakterienpopulationen zu verringern. ⓘ
Forensische Untersuchung
Die DNA-Sequenzierung kann zusammen mit DNA-Profilierungsmethoden für die forensische Identifizierung und Vaterschaftstests verwendet werden. Die DNA-Tests haben sich in den letzten Jahrzehnten enorm weiterentwickelt, um einen DNA-Abdruck mit dem zu untersuchenden Objekt in Verbindung zu bringen. Die DNA-Muster in Fingerabdrücken, Speichel, Haarfollikeln usw. unterscheiden jeden lebenden Organismus auf einzigartige Weise von anderen. Der DNA-Test ist eine Technik, die spezifische Genome in einem DNA-Strang nachweisen kann, um ein einzigartiges und individuelles Muster zu erzeugen. ⓘ
Die vier kanonischen Basen
Die kanonische Struktur der DNA hat vier Basen: Thymin (T), Adenin (A), Cytosin (C) und Guanin (G). Bei der DNA-Sequenzierung wird die physikalische Reihenfolge dieser Basen in einem DNA-Molekül bestimmt. Es gibt jedoch noch viele andere Basen, die in einem Molekül vorhanden sein können. In einigen Viren (insbesondere Bakteriophagen) kann Cytosin durch Hydroxymethyl- oder Hydroxymethylglucosecytosin ersetzt sein. In der DNA von Säugetieren können Basenvarianten mit Methylgruppen oder Phosphosulfat vorkommen. Je nach Sequenzierungstechnik kann eine bestimmte Modifikation, z. B. das beim Menschen übliche 5mC (5-Methyl-Cytosin), nachgewiesen werden oder auch nicht. ⓘ
Geschichte
Entdeckung der DNA-Struktur und -Funktion
Die Desoxyribonukleinsäure (DNS) wurde erstmals 1869 von Friedrich Miescher entdeckt und isoliert, blieb jedoch viele Jahrzehnte lang unerkannt, da man davon ausging, dass Proteine und nicht die DNS den genetischen Bauplan des Lebens enthalten. Dies änderte sich nach 1944, als Oswald Avery, Colin MacLeod und Maclyn McCarty in einigen Experimenten nachwiesen, dass gereinigte DNA einen Bakterienstamm in einen anderen verwandeln kann. Dies war das erste Mal, dass DNA nachweislich in der Lage war, die Eigenschaften von Zellen zu verändern. ⓘ
1953 stellten James Watson und Francis Crick ihr Doppelhelix-Modell der DNA vor, das auf kristallisierten Röntgenstrukturen basierte, die von Rosalind Franklin untersucht wurden. Nach diesem Modell besteht die DNS aus zwei umeinander gewundenen Strängen von Nukleotiden, die durch Wasserstoffbrückenbindungen miteinander verbunden sind und in entgegengesetzte Richtungen verlaufen. Jeder Strang besteht aus vier komplementären Nukleotiden - Adenin (A), Cytosin (C), Guanin (G) und Thymin (T) -, wobei ein A auf dem einen Strang immer mit einem T auf dem anderen Strang gepaart ist und ein C immer mit einem G. Sie schlugen vor, dass eine solche Struktur es ermöglicht, jeden Strang zu verwenden, um den anderen zu rekonstruieren, eine Idee, die für die Weitergabe von Erbinformationen zwischen Generationen von zentraler Bedeutung ist. ⓘ

Der Grundstein für die Sequenzierung von Proteinen wurde durch die Arbeit von Frederick Sanger gelegt, der 1955 die Sequenz aller Aminosäuren des Insulins, eines kleinen Proteins der Bauchspeicheldrüse, fertiggestellt hatte. Dies lieferte den ersten schlüssigen Beweis dafür, dass Proteine chemische Einheiten mit einem spezifischen molekularen Muster sind und nicht eine zufällige Mischung von in Flüssigkeit suspendiertem Material. Sangers Erfolg bei der Sequenzierung von Insulin spornte die Röntgenkristallographen an, darunter auch Watson und Crick, die nun versuchten zu verstehen, wie die DNA die Bildung von Proteinen in einer Zelle steuert. Schon bald nach dem Besuch einer Vortragsreihe von Frederick Sanger im Oktober 1954 begann Crick mit der Entwicklung einer Theorie, die besagte, dass die Anordnung der Nukleotide in der DNA die Reihenfolge der Aminosäuren in den Proteinen bestimmt, was wiederum die Funktion eines Proteins mitbestimmt. Er veröffentlichte diese Theorie im Jahr 1958. ⓘ
RNA-Sequenzierung
Die RNA-Sequenzierung war eine der frühesten Formen der Nukleotidsequenzierung. Der wichtigste Meilenstein der RNA-Sequenzierung ist die Sequenzierung des ersten vollständigen Gens und des vollständigen Genoms des Bakteriophagen MS2, die 1972 und 1976 von Walter Fiers und seinen Mitarbeitern an der Universität Gent (Gent, Belgien) identifiziert und veröffentlicht wurden. Traditionelle RNA-Sequenzierungsmethoden erfordern die Herstellung eines cDNA-Moleküls, das sequenziert werden muss. ⓘ
Frühe DNA-Sequenzierungsmethoden
Die erste Methode zur Bestimmung von DNA-Sequenzen bestand in einer ortsspezifischen Primer-Verlängerungsstrategie, die 1970 von Ray Wu an der Cornell University entwickelt wurde. Zur Sequenzierung der kohäsiven Enden der Lambda-Phagen-DNA wurden die DNA-Polymerase-Katalyse und die spezifische Nukleotidmarkierung verwendet, die beide in den heutigen Sequenzierungsverfahren eine wichtige Rolle spielen. Zwischen 1970 und 1973 demonstrierten Wu, R. Padmanabhan und Kollegen, dass diese Methode zur Bestimmung jeder beliebigen DNA-Sequenz unter Verwendung synthetischer ortsspezifischer Primer eingesetzt werden kann. Frederick Sanger übernahm diese Primer-Verlängerungsstrategie, um am MRC Centre in Cambridge, UK, schnellere DNA-Sequenzierungsmethoden zu entwickeln, und veröffentlichte 1977 eine Methode zur "DNA-Sequenzierung mit kettenabschließenden Inhibitoren". Walter Gilbert und Allan Maxam in Harvard entwickelten ebenfalls Sequenzierungsmethoden, darunter eine Methode zur "DNA-Sequenzierung durch chemischen Abbau". 1973 berichteten Gilbert und Maxam über die Sequenzierung von 24 Basenpaaren mit Hilfe einer Methode, die als "wandering-spot analysis" bekannt ist. Die Fortschritte bei der Sequenzierung wurden durch die gleichzeitige Entwicklung der rekombinanten DNA-Technologie begünstigt, die es ermöglichte, DNA-Proben aus anderen Quellen als Viren zu isolieren. ⓘ
Sequenzierung vollständiger Genome

Das erste vollständige DNA-Genom, das sequenziert wurde, war das des Bakteriophagen φX174 im Jahr 1977. Wissenschaftler des Medical Research Council entschlüsselten 1984 die vollständige DNA-Sequenz des Epstein-Barr-Virus und fanden heraus, dass sie 172 282 Nukleotide umfasst. Die Entschlüsselung der Sequenz markierte einen bedeutenden Wendepunkt in der DNA-Sequenzierung, da sie ohne vorherige Kenntnis des genetischen Profils des Virus erfolgte. ⓘ
Anfang der 1980er Jahre entwickelten Herbert Pohl und seine Mitarbeiter eine nicht-radioaktive Methode, um die DNA-Moleküle von Sequenzierreaktionsmischungen während der Elektrophorese auf eine immobilisierende Matrix zu übertragen. Es folgte die Kommerzialisierung des DNA-Sequenzierers "Direct-Blotting-Electrophoresis-System GATC 1500" durch die Firma GATC Biotech, der im Rahmen des EU-Genom-Sequenzierprogramms, der vollständigen DNA-Sequenz der Hefe Saccharomyces cerevisiae Chromosom II, intensiv genutzt wurde. Das Labor von Leroy E. Hood am California Institute of Technology stellte 1986 die erste halbautomatische DNA-Sequenzierungsmaschine vor. Es folgten 1987 das erste vollautomatische Sequenziergerät ABI 370 von Applied Biosystems und das Genesis 2000 von Dupont, das eine neuartige Fluoreszenzmarkierungstechnik verwendete, mit der alle vier Dideoxynukleotide in einer einzigen Spur identifiziert werden konnten. 1990 begannen die U.S. National Institutes of Health (NIH) mit groß angelegten Sequenzierungsversuchen an Mycoplasma capricolum, Escherichia coli, Caenorhabditis elegans und Saccharomyces cerevisiae zu einem Preis von 0,75 US$ pro Base. In der Zwischenzeit wurde im Labor von Craig Venter mit der Sequenzierung menschlicher cDNA-Sequenzen, den so genannten Expressed Sequence Tags, begonnen, ein Versuch, den kodierenden Teil des menschlichen Genoms zu erfassen. 1995 veröffentlichten Venter, Hamilton Smith und Kollegen vom Institute for Genomic Research (TIGR) das erste vollständige Genom eines frei lebenden Organismus, des Bakteriums Haemophilus influenzae. Das zirkuläre Chromosom enthält 1.830.137 Basen, und die Veröffentlichung in der Zeitschrift Science markierte den ersten veröffentlichten Einsatz der Shotgun-Sequenzierung des gesamten Genoms, wodurch die Notwendigkeit einer anfänglichen Kartierung entfiel. ⓘ
Im Jahr 2001 wurde mit Hilfe von Shotgun-Sequenzierungsmethoden ein Entwurf des menschlichen Genoms erstellt. ⓘ
Hochdurchsatz-Sequenzierungsmethoden (HTS)

Mitte bis Ende der 1990er Jahre wurden mehrere neue Methoden für die DNA-Sequenzierung entwickelt, die bis zum Jahr 2000 in kommerzielle DNA-Sequenziergeräte integriert wurden. Diese wurden als "Next-Generation"- oder "Second-Generation"-Sequenzierungsmethoden (NGS) bezeichnet, um sie von den früheren Methoden, einschließlich der Sanger-Sequenzierung, zu unterscheiden. Im Gegensatz zur ersten Generation der Sequenzierung zeichnet sich die NGS-Technologie in der Regel durch eine hohe Skalierbarkeit aus, so dass das gesamte Genom auf einmal sequenziert werden kann. In der Regel wird dies erreicht, indem das Genom in kleine Teile fragmentiert wird, ein Fragment nach dem Zufallsprinzip entnommen und mit einer der verschiedenen Technologien, wie den unten beschriebenen, sequenziert wird. Ein ganzes Genom ist möglich, weil mehrere Fragmente gleichzeitig sequenziert werden (daher der Name "massiv parallele" Sequenzierung), und zwar in einem automatisierten Prozess. ⓘ
Die NGS-Technologie hat es Forschern ermöglicht, Erkenntnisse über die Gesundheit zu gewinnen, Anthropologen die Herkunft des Menschen zu erforschen und die Bewegung der "personalisierten Medizin" voranzutreiben. Allerdings hat sie auch die Tür zu mehr Raum für Fehler geöffnet. Es gibt viele Software-Tools für die computergestützte Analyse von NGS-Daten, die oft auf Online-Plattformen wie dem CSI NGS Portal zusammengestellt werden, und jedes hat seinen eigenen Algorithmus. Selbst die Parameter innerhalb eines Softwarepakets können das Ergebnis der Analyse verändern. Darüber hinaus haben die großen Datenmengen, die bei der DNA-Sequenzierung anfallen, auch die Entwicklung neuer Methoden und Programme für die Sequenzanalyse erforderlich gemacht. Um diese Herausforderungen zu bewältigen, wurden mehrere Versuche unternommen, Standards im Bereich der NGS zu entwickeln, wobei es sich zumeist um kleinere Bemühungen einzelner Labors handelte. In jüngster Zeit hat eine große, organisierte und von der FDA finanzierte Anstrengung im BioCompute-Standard ihren Höhepunkt gefunden. ⓘ
Am 26. Oktober 1990 meldeten Roger Tsien, Pepi Ross, Margaret Fahnestock und Allan J. Johnston ein Patent an, das die schrittweise ("base-by-base") Sequenzierung mit entfernbaren 3'-Blockern auf DNA-Arrays (Blots und einzelne DNA-Moleküle) beschreibt. 1996 veröffentlichten Pål Nyrén und sein Student Mostafa Ronaghi vom Royal Institute of Technology in Stockholm ihre Methode der Pyrosequenzierung. ⓘ
Am 1. April 1997 reichten Pascal Mayer [fr] und Laurent Farinelli bei der Weltorganisation für geistiges Eigentum Patente ein, die die DNA-Kolonie-Sequenzierung beschreiben. Die in diesem Patent beschriebene Methode der DNA-Probenvorbereitung und der zufälligen Oberflächen-Polymerase-Kettenreaktion (PCR) in Verbindung mit der "Base-by-Base"-Sequenzierungsmethode von Roger Tsien et al. wird jetzt in den Hi-Seq-Genomsequenzierern von Illumina eingesetzt. ⓘ
1998 beschrieben Phil Green und Brent Ewing von der University of Washington ihren phred quality score für die Analyse von Sequenzierdaten, eine bahnbrechende Analysetechnik, die weit verbreitet wurde und immer noch die gängigste Metrik zur Bewertung der Genauigkeit einer Sequenzierplattform ist. ⓘ
Lynx Therapeutics veröffentlichte und vermarktete im Jahr 2000 das Massively Parallel Signature Sequencing (MPSS). Diese Methode umfasste eine parallelisierte, durch Adapter/Ligation vermittelte Sequenzierungstechnologie auf Bead-Basis und diente als erste kommerziell verfügbare "Next-Generation"-Sequenzierungsmethode, obwohl keine DNA-Sequenzer an unabhängige Labors verkauft wurden. ⓘ
Grundlegende Methoden
Maxam-Gilbert-Sequenzierung
Allan Maxam und Walter Gilbert veröffentlichten 1977 eine DNA-Sequenzierungsmethode, die auf der chemischen Modifizierung der DNA und der anschließenden Spaltung an bestimmten Basen beruht. Diese auch als chemische Sequenzierung bezeichnete Methode ermöglichte die Verwendung gereinigter Proben doppelsträngiger DNA ohne weitere Klonierung. Der Einsatz radioaktiver Markierungen und die technische Komplexität dieser Methode haben nach der Weiterentwicklung der Sanger-Methode von einer breiten Anwendung abgehalten. ⓘ
Die Maxam-Gilbert-Sequenzierung erfordert eine radioaktive Markierung an einem 5'-Ende der DNA und die Reinigung des zu sequenzierenden DNA-Fragments. Durch eine chemische Behandlung werden dann in jeder der vier Reaktionen (G, A+G, C, C+T) Brüche an einem kleinen Teil von einer oder zwei der vier Nukleotidbasen erzeugt. Die Konzentration der modifizierenden Chemikalien wird so gesteuert, dass im Durchschnitt eine Modifikation pro DNA-Molekül erzeugt wird. Auf diese Weise entsteht eine Reihe von markierten Fragmenten, vom radiomarkierten Ende bis zur ersten "Schnitt"-Stelle in jedem Molekül. Die Fragmente der vier Reaktionen werden zur Größentrennung nebeneinander in denaturierenden Acrylamidgelen elektrophoretisiert. Um die Fragmente sichtbar zu machen, wird das Gel für die Autoradiographie einem Röntgenfilm ausgesetzt, der eine Reihe von dunklen Banden zeigt, die jeweils einem radioaktiv markierten DNA-Fragment entsprechen und aus denen die Sequenz abgeleitet werden kann. ⓘ
Methoden des Kettenabbruchs
Die von Frederick Sanger und seinen Mitarbeitern 1977 entwickelte Kettenterminationsmethode wurde aufgrund ihrer relativen Einfachheit und Zuverlässigkeit bald zur Methode der Wahl. Bei ihrer Erfindung wurden bei der Kettenabbruchmethode weniger giftige Chemikalien und geringere Mengen an Radioaktivität verwendet als bei der Methode von Maxam und Gilbert. Aufgrund ihrer vergleichsweise einfachen Handhabung wurde die Sanger-Methode bald automatisiert und in der ersten Generation von DNA-Sequenzierern eingesetzt. ⓘ
Die Sanger-Sequenzierung ist die Methode, die von den 1980er Jahren bis Mitte der 2000er Jahre vorherrschte. In diesem Zeitraum wurden große Fortschritte in der Technik gemacht, wie z. B. die Fluoreszenzmarkierung, die Kapillarelektrophorese und die allgemeine Automatisierung. Diese Entwicklungen ermöglichten eine wesentlich effizientere Sequenzierung, was zu niedrigeren Kosten führte. Die Sanger-Methode in Form einer Massenproduktion ist die Technologie, mit der 2001 das erste menschliche Genom erstellt wurde und die das Zeitalter der Genomik einleitete. Im weiteren Verlauf des Jahrzehnts kamen jedoch radikal andere Ansätze auf den Markt, die die Kosten pro Genom von 100 Millionen Dollar im Jahr 2001 auf 10.000 Dollar im Jahr 2011 sinken ließen. ⓘ
Sequenzierung im großen Maßstab und de novo-Sequenzierung
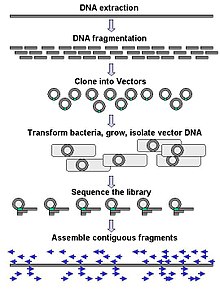
Die Sequenzierung im großen Maßstab zielt häufig auf die Sequenzierung sehr langer DNA-Stücke ab, wie z. B. ganzer Chromosomen, obwohl die Sequenzierung im großen Maßstab auch dazu verwendet werden kann, eine sehr große Anzahl kurzer Sequenzen zu erzeugen, wie z. B. beim Phagen-Display. Bei längeren Zielobjekten wie Chromosomen bestehen die üblichen Ansätze darin, große DNA-Fragmente (mit Restriktionsenzymen) zu schneiden oder (mit mechanischen Kräften) in kürzere DNA-Fragmente zu scheren. Die fragmentierte DNA kann dann in einen DNA-Vektor kloniert und in einem bakteriellen Wirt wie Escherichia coli amplifiziert werden. Kurze, aus einzelnen Bakterienkolonien gereinigte DNA-Fragmente werden einzeln sequenziert und elektronisch zu einer langen, zusammenhängenden Sequenz zusammengesetzt. Studien haben gezeigt, dass das Hinzufügen eines Größenauswahlschritts zum Sammeln von DNA-Fragmenten einheitlicher Größe die Sequenziereffizienz und die Genauigkeit der Genomassemblierung verbessern kann. In diesen Studien hat sich die automatische Größenselektion als reproduzierbarer und präziser erwiesen als die manuelle Größenselektion im Gel. ⓘ
Der Begriff "de novo-Sequenzierung" bezieht sich speziell auf Methoden zur Bestimmung der Sequenz von DNA, deren Sequenz zuvor nicht bekannt war. De novo bedeutet aus dem Lateinischen übersetzt "von Anfang an". Lücken in der assemblierten Sequenz können durch Primer-Walking geschlossen werden. Shotgun-Methoden werden häufig für die Sequenzierung großer Genome verwendet, aber die Assemblierung ist komplex und schwierig, vor allem weil Sequenzwiederholungen häufig Lücken in der Genomassemblierung verursachen. ⓘ
Die meisten Sequenzierungsverfahren verwenden einen In-vitro-Klonierungsschritt, um einzelne DNA-Moleküle zu amplifizieren, da ihre molekularen Nachweismethoden nicht empfindlich genug für die Sequenzierung einzelner Moleküle sind. Bei der Emulsions-PCR werden einzelne DNA-Moleküle zusammen mit Primer-beschichteten Kügelchen in wässrigen Tröpfchen innerhalb einer Ölphase isoliert. Durch eine Polymerase-Kettenreaktion (PCR) wird dann jedes Bead mit klonalen Kopien des DNA-Moleküls beschichtet und anschließend für die spätere Sequenzierung immobilisiert. Die Emulsions-PCR wird in den von Marguilis et al. (von 454 Life Sciences vermarktet), Shendure und Porreca et al. (auch bekannt als "Polony Sequencing") und SOLiD Sequencing (entwickelt von Agencourt, später Applied Biosystems, jetzt Life Technologies) entwickelten Methoden eingesetzt. Emulsions-PCR wird auch in den von 10x Genomics entwickelten Plattformen GemCode und Chromium verwendet. ⓘ
Shotgun-Sequenzierung
Die Shotgun-Sequenzierung ist eine Sequenzierungsmethode für die Analyse von DNA-Sequenzen, die länger als 1000 Basenpaare sind, bis hin zu ganzen Chromosomen. Bei dieser Methode muss die Ziel-DNA in zufällige Fragmente zerlegt werden. Nach der Sequenzierung der einzelnen Fragmente mit Hilfe der Kettenterminationsmethode können die Sequenzen anhand ihrer überlappenden Bereiche wieder zusammengesetzt werden. ⓘ
Hochdurchsatz-Methoden

Die Hochdurchsatz-Sequenzierung, zu der die "Short-Read"- und "Long-Read"-Sequenzierungsverfahren der nächsten und dritten Generation gehören, wird für die Exom-Sequenzierung, Genom-Sequenzierung, Genom-Resequenzierung, Transkriptom-Profilierung (RNA-Seq), DNA-Protein-Interaktionen (ChIP-Sequenzierung) und Epigenom-Charakterisierung eingesetzt. ⓘ
Die hohe Nachfrage nach kostengünstiger Sequenzierung hat die Entwicklung von Hochdurchsatz-Sequenzierungstechnologien vorangetrieben, die den Sequenzierungsprozess parallelisieren und Tausende oder Millionen von Sequenzen gleichzeitig produzieren. Hochdurchsatz-Sequenzierungstechnologien sollen die Kosten der DNA-Sequenzierung über das hinaus senken, was mit Standard-Farbstoffterminator-Methoden möglich ist. Bei der Ultra-Hochdurchsatz-Sequenzierung können bis zu 500.000 Sequenziervorgänge durch Synthese parallel durchgeführt werden. Solche Technologien haben dazu geführt, dass ein ganzes menschliches Genom in nur einem Tag sequenziert werden kann. Zu den führenden Unternehmen bei der Entwicklung von Hochdurchsatz-Sequenzierungsprodukten gehören ab 2019 Illumina, Qiagen und ThermoFisher Scientific. ⓘ
| Methode | Leselänge | Genauigkeit (einzelner Read, nicht Konsens) | Reads pro Lauf | Zeit pro Durchlauf | Kosten pro 1 Milliarde Basen (in US$) | Vorteile | Nachteile ⓘ |
|---|---|---|---|---|---|---|---|
| Einzelmolekül-Echtzeit-Sequenzierung (Pacific Biosciences) | 30.000 bp (N50);
maximale Leselänge >100.000 Basen |
87% Roh-Lesegenauigkeit | 4.000.000 pro Sequel 2 SMRT-Zelle, 100-200 Gigabasen | 30 Minuten bis 20 Stunden | $7.2-$43.3 | Schnell. Erfasst 4mC, 5mC, 6mA. | Mäßiger Durchsatz. Die Ausrüstung kann sehr teuer sein. |
| Ionen-Halbleiter (Ion Torrent Sequenzierung) | bis zu 600 bp | 99.6% | bis zu 80 Millionen | 2 Stunden | $66.8-$950 | Weniger teure Ausrüstung. Schnell. | Homopolymer-Fehler. |
| Pyrosequenzierung (454) | 700 bp | 99.9% | 1 Million | 24 Stunden | $10,000 | Lange Lesegröße. Schnell. | Läufe sind teuer. Homopolymer-Fehler. |
| Sequenzierung durch Synthese (Illumina) | MiniSeq, NextSeq: 75-300 bp;
MiSeq: 50-600 bp; HiSeq 2500: 50-500 bp; HiSeq 3/4000: 50-300 bp; HiSeq X: 300 bp |
99,9 % (Phred30) | MiniSeq/MiSeq: 1-25 Millionen;
NextSeq: 130-00 Millionen; HiSeq 2500: 300 Millionen - 2 Milliarden; HiSeq 3/4000: 2,5 Milliarden; HiSeq X: 3 Milliarden |
1 bis 11 Tage, je nach Sequenzer und angegebener Leselänge | $5 bis $150 | Potenzial für hohe Sequenzausbeute, je nach Sequenzierermodell und gewünschter Anwendung. | Die Ausrüstung kann sehr teuer sein. Erfordert hohe Konzentrationen von DNA. |
| Kombinatorische Sondenankersynthese (cPAS- BGI/MGI) | BGISEQ-50: 35-50bp;
MGISEQ 200: 50-200bp; BGISEQ-500, MGISEQ-2000: 50-300bp |
99,9 % (Phred30) | BGISEQ-50: 160M;
MGISEQ 200: 300M; BGISEQ-500: 1300M pro Durchflusszelle; MGISEQ-2000: 375M FCS-Fließzelle, 1500M FCL-Fließzelle pro Fließzelle. |
1 bis 9 Tage je nach Gerät, Leselänge und Anzahl der gleichzeitig ausgeführten Fließzellen. | $5– $120 | ||
| Sequenzierung durch Ligation (SOLiD-Sequenzierung) | 50+35 oder 50+50 bp | 99.9% | 1,2 bis 1,4 Milliarden | 1 bis 2 Wochen | $60–130 | Niedrige Kosten pro Base. | Langsamer als andere Methoden. Hat Probleme bei der Sequenzierung von palindromischen Sequenzen. |
| Nanopore-Sequenzierung | Abhängig von der Bibliotheksvorbereitung, nicht vom Gerät, daher wählt der Benutzer die Leselänge (bis zu 2.272.580 bp berichtet). | ~92-97% Single-Read | abhängig von der vom Benutzer gewählten Leselänge | Daten werden in Echtzeit gestreamt. Wählen Sie zwischen 1 Minute und 48 Stunden | $7–100 | Längste einzelne Reads. Zugängliche Benutzergemeinschaft. Tragbar (in Palm-Größe). | Geringerer Durchsatz als andere Geräte, Einzel-Lesegenauigkeit in den 90ern. |
| GenapSys Sequenzierung | Rund 150 bp single-end | 99,9 % (Phred30) | 1 bis 16 Millionen | Etwa 24 Stunden | $667 | Geringe Kosten für das Gerät ($10.000) | |
| Kettenabbruch (Sanger-Sequenzierung) | 400 bis 900 bp | 99.9% | K.A. | 20 Minuten bis 3 Stunden | $2,400,000 | Nützlich für viele Anwendungen. | Teurer und unpraktisch für größere Sequenzierungsprojekte. Diese Methode erfordert außerdem den zeitaufwändigen Schritt der Plasmidklonierung oder PCR. |
Long-read Sequenziermethoden
Einzelmolekül-Echtzeit-Sequenzierung (SMRT)
Die SMRT-Sequenzierung basiert auf dem Ansatz der Sequenzierung durch Synthese. Die DNA wird in Zero-Mode-Wave-Guides (ZMWs) synthetisiert, d. h. in kleinen, wannenartigen Behältern, in denen sich die Aufnahmewerkzeuge am Boden der Wanne befinden. Die Sequenzierung erfolgt unter Verwendung von unmodifizierter Polymerase (die am Boden des ZMW angebracht ist) und fluoreszenzmarkierten Nukleotiden, die frei in der Lösung fließen. Die Vertiefungen sind so konstruiert, dass nur die am Boden der Vertiefung auftretende Fluoreszenz erfasst wird. Die Fluoreszenzmarkierung wird nach dem Einbau in den DNA-Strang vom Nukleotid abgelöst, so dass ein unveränderter DNA-Strang zurückbleibt. Nach Angaben von Pacific Biosciences (PacBio), dem Entwickler der SMRT-Technologie, ermöglicht diese Methode den Nachweis von Nukleotidveränderungen (z. B. Cytosinmethylierung). Dies geschieht durch die Beobachtung der Polymerasekinetik. Dieser Ansatz ermöglicht Lesevorgänge von 20.000 Nukleotiden oder mehr mit einer durchschnittlichen Leselänge von 5 Kilobasen. Im Jahr 2015 kündigte Pacific Biosciences die Einführung eines neuen Sequenziergeräts namens Sequel System an, das 1 Million ZMWs im Vergleich zu 150.000 ZMWs im PacBio RS II-Gerät bietet. Die SMRT-Sequenzierung wird als "dritte Generation" oder "Long-Read"-Sequenzierung bezeichnet. ⓘ
Nanopore-DNA-Sequenzierung
Die DNA, die die Nanopore durchläuft, verändert ihren Ionenstrom. Diese Veränderung ist abhängig von der Form, Größe und Länge der DNA-Sequenz. Jede Art von Nukleotid blockiert den Ionenfluss durch die Pore für eine unterschiedliche Zeitspanne. Die Methode erfordert keine modifizierten Nukleotide und wird in Echtzeit durchgeführt. Die Nanopore-Sequenzierung wird zusammen mit der SMRT-Sequenzierung als "Sequenzierung der dritten Generation" oder "Long-Read-Sequenzierung" bezeichnet. ⓘ
Die frühe industrielle Forschung zu dieser Methode basierte auf einer Technik namens "Exonuklease-Sequenzierung", bei der das Auslesen elektrischer Signale erfolgte, wenn Nukleotide durch kovalent mit Cyclodextrin gebundene alpha(α)-Hämolysin-Poren passierten. Die spätere kommerzielle Methode, die "Strangsequenzierung", sequenziert dagegen DNA-Basen in einem intakten Strang. ⓘ
Zwei Hauptbereiche der Nanoporen-Sequenzierung, die sich in der Entwicklung befinden, sind die Festkörper-Nanoporen-Sequenzierung und die proteinbasierte Nanoporen-Sequenzierung. Bei der Protein-Nanoporen-Sequenzierung werden Membranproteinkomplexe wie α-Hämolysin, MspA (Mycobacterium smegmatis Porin A) oder CssG verwendet, die aufgrund ihrer Fähigkeit, zwischen einzelnen Nukleotiden und Nukleotidgruppen zu unterscheiden, sehr vielversprechend sind. Im Gegensatz dazu werden bei der Festkörper-Nanoporen-Sequenzierung synthetische Materialien wie Siliziumnitrid und Aluminiumoxid verwendet, die aufgrund ihrer überlegenen mechanischen Eigenschaften und ihrer thermischen und chemischen Stabilität bevorzugt werden. Die Herstellungsmethode ist für diese Art der Sequenzierung von entscheidender Bedeutung, da die Nanoporenanordnung Hunderte von Poren mit Durchmessern von weniger als acht Nanometern enthalten kann. ⓘ
Das Konzept geht auf die Idee zurück, dass einzelsträngige DNA- oder RNA-Moleküle elektrophoretisch in einer strikt linearen Abfolge durch eine biologische Pore von weniger als acht Nanometern getrieben und nachgewiesen werden können, da die Moleküle bei ihrer Bewegung durch die Pore einen Ionenstrom abgeben. Die Pore enthält eine Detektionsregion, die in der Lage ist, verschiedene Basen zu erkennen, wobei jede Base beim Durchqueren der Pore verschiedene zeitlich spezifische Signale erzeugt, die der Sequenz der Basen entsprechen und dann ausgewertet werden. Die genaue Kontrolle des DNA-Transports durch die Pore ist entscheidend für den Erfolg. Verschiedene Enzyme wie Exonukleasen und Polymerasen wurden eingesetzt, um diesen Prozess zu moderieren, indem sie in der Nähe des Poreneingangs positioniert wurden. ⓘ
Bei der Einzelstrangsequenzierung wird doppelsträngiger DNA durch eine Helikase getrennt und in die Nanopore eingeführt. Im Falle einer MspA-Pore befinden sich gleichzeitig vier Nukleotide der DNA innerhalb der Pore. Die Durchtrittsgeschwindigkeit ist unter anderem von der pH-Wert-Differenz beidseitig der Membran abhängig. Durch die spezifischen Ionenstromänderungen für jedes der vier Nukleotide lässt sich aus dem erhaltenen Datensatz die Sequenz ablesen. Eine Auswertung erfolgt z. B. mit der Software Poretools. Der Vorteil der Methode besteht in ihrer Geschwindigkeit und in der gleichbleibenden Genauigkeit auch bei langen DNA-Strängen. ⓘ
Ira Deveson organisierte eine Untersuchung des ganzen RNA-Genoms von SARS-CoV-2 aus 157 infizierten Patienten. Ein überraschendes Ergebnis war, wie sehr die Virenproben variierten. Die Methode wird nicht nur für Nukleinsäuren, sondern auch, mit Abwandlung, zur Proteinsequenzierung verwendet. ⓘ
Short-read Sequenziermethoden
Massiv parallele Signatursequenzierung (MPSS)
Die erste der Hochdurchsatz-Sequenzierungstechnologien, die massiv-parallele Signatursequenzierung (MPSS), wurde in den 1990er Jahren von Lynx Therapeutics entwickelt, einem 1992 von Sydney Brenner und Sam Eletr gegründeten Unternehmen. MPSS war eine auf Beads basierende Methode, die einen komplexen Ansatz der Adapterligierung mit anschließender Adapterdecodierung verwendete, wobei die Sequenz in Schritten von vier Nukleotiden gelesen wurde. Diese Methode machte sie anfällig für sequenzspezifische Verzerrungen oder den Verlust bestimmter Sequenzen. Da die Technologie so komplex war, wurde MPSS nur intern von Lynx Therapeutics durchgeführt und es wurden keine DNA-Sequenziergeräte an unabhängige Labors verkauft. Lynx Therapeutics fusionierte 2004 mit Solexa (später von Illumina übernommen), was zur Entwicklung der Sequenzierung durch Synthese führte, einem einfacheren Ansatz, der von Manteia Predictive Medicine übernommen wurde und MPSS obsolet machte. Die wesentlichen Eigenschaften der MPSS-Ausgabe waren jedoch typisch für spätere Hochdurchsatz-Datentypen, einschließlich Hunderttausender kurzer DNA-Sequenzen. Im Falle von MPSS wurden diese typischerweise für die Sequenzierung von cDNA zur Messung der Genexpressionswerte verwendet. ⓘ
Polony-Sequenzierung
Die Polony-Sequenzierungsmethode, die im Labor von George M. Church in Harvard entwickelt wurde, gehörte zu den ersten Hochdurchsatz-Sequenzierungssystemen und wurde 2005 zur Sequenzierung eines vollständigen E. coli-Genoms eingesetzt. Dabei wurde eine in vitro-Bibliothek mit gepaarten Markierungen mit Emulsions-PCR, einem automatisierten Mikroskop und ligationsbasierter Sequenzierungschemie kombiniert, um ein E. coli-Genom mit einer Genauigkeit von mehr als 99,9999 % und zu Kosten zu sequenzieren, die etwa 1/9 der Kosten der Sanger-Sequenzierung betragen. Die Technologie wurde an Agencourt Biosciences lizenziert, anschließend in Agencourt Personal Genomics ausgegliedert und schließlich in die SOLiD-Plattform von Applied Biosystems integriert. Applied Biosystems wurde später von Life Technologies, jetzt Teil von Thermo Fisher Scientific, übernommen. ⓘ
454 Pyrosequenzierung
Die Pyrosequenzierung nutzt wie die Sanger-Sequenzierung eine DNA-Polymerase zur Synthese des DNA-Gegenstranges, wobei der Typ der DNA-Polymerase durchaus noch unterschiedlich sein kann. Die DNA-Mischung wird mit einem DNA-Adapter ligiert und über eine komplementäre Adaptersequenz an beads gekoppelt. Die mit DNA beladenen beads werden auf eine Platte mit Poren von der Größe eines beads gegeben, bei der unter jeder Pore ein Lichtleiter zu einem Detektor führt. Die DNA-Polymerase wird gewissermaßen „in Aktion“ beobachtet, wie sie nacheinander einzelne Nukleotide an einen neusynthetisierten DNA-Strang anhängt. Der erfolgreiche Einbau eines Nukleotids wird durch ein ausgeklügeltes Enzymsystem unter Beteiligung einer Luziferase in einen Lichtblitz umgesetzt und von einem Detektor erfasst. Die zu sequenzierende DNA dient als Matrizenstrang und liegt einzelsträngig vor. Ausgehend von einem Primer erfolgt die Strangverlängerung, Nukleotid um Nukleotid, durch Zugabe von jeweils einer der vier Arten der Desoxynukleosidtriphosphate (dNTP). Bei Zugabe des passenden (komplementären) Nukleotids erhält man ein Signal. Wurde ein an dieser Stelle nicht passendes NTP zugegeben, bleibt der Lichtblitz aus. Danach werden die vorhandenen NTP zerstört, und eine andere Art wird zugesetzt; dies wird fortgesetzt, bis sich wieder eine Reaktion zeigt; spätestens nach der vierten Zugabe zeigt sich eine Reaktion, da dann alle Arten von NTP durchprobiert wurden. ⓘ
Bei Einbau eines komplementären Nukleotids durch die DNA-Polymerase wird Pyrophosphat (PPi) freigesetzt. Das Pyrophosphat wird durch die ATP-Sulfurylase zu Adenosintriphosphat (ATP) umgesetzt. Das ATP treibt die Luziferase-Reaktion an, wodurch Luziferin in Oxyluziferin umgesetzt wird. Dies resultiert wiederum in einem detektierbaren Lichtsignal – dessen Stärke proportional zum verbrauchten ATP ist. ⓘ
Die Pyrosequenzierung wird beispielsweise zur Bestimmung der Häufigkeit von bestimmten Genmutationen (SNPs, engl. Single Nucleotide Polymorphism), z. B. bei der Untersuchung von Erbkrankheiten eingesetzt. Die Pyrosequenzierung ist gut automatisierbar und eignet sich zur hochparallelen Analyse von DNA-Proben. ⓘ
Pyrosequenzierung wurde Mitte der 1990er Jahre von Mathias Uhlén, Mostafa Ronaghi und Pål Nyrén entwickelt (Nyrén erhielt dafür 2013 den Europäischen Erfinderpreis) und ab 1999 von Jonathan Rothberg in der 454 GS FLX der Firma 454 Life Sciences mit Chip-Technologie umgesetzt (siehe Ionen-Halbleiter-DNA-Sequenzierungssystem), die 2005 als erste Next Generation Plattform auf den Markt kam (aufgekauft von Roche Diagnostics 2007). Mit der 454 GS FLX gelang es 2007 das Genom von James Watson, der die Doppelhelixstruktur der DNA 1953 mit Francis Crick entdeckte, in nur 2 Monaten zu sequenzieren, während das 2003 abgeschlossene erste Human Genome Project noch 13 Jahren benötigte. ⓘ
Illumina (Solexa) Sequenzierung
Solexa, jetzt Teil von Illumina, wurde 1998 von Shankar Balasubramanian und David Klenerman gegründet und entwickelte eine Sequenzierungsmethode, die auf der Technologie der reversiblen Farbstoffterminatoren und speziell entwickelten Polymerasen basiert. Das Konzept der reversiblen Terminatorchemie wurde von Bruno Canard und Simon Sarfati am Institut Pasteur in Paris erfunden. Es wurde intern bei Solexa von den in den entsprechenden Patenten genannten Personen entwickelt. 2004 erwarb Solexa das Unternehmen Manteia Predictive Medicine, um eine 1997 von Pascal Mayer [fr] und Laurent Farinelli erfundene Technologie zur massiv parallelen Sequenzierung zu erwerben. Sie basiert auf "DNA-Clustern" oder "DNA-Kolonien", die die klonale Vervielfältigung von DNA auf einer Oberfläche beinhalten. Die Clustertechnologie wurde gemeinsam mit Lynx Therapeutics aus Kalifornien erworben. Solexa Ltd. fusionierte später mit Lynx und gründete Solexa Inc. ⓘ


Bei dieser Methode werden zunächst DNA-Moleküle und Primer auf einem Objektträger oder einer Fließzelle angebracht und mit Polymerase vervielfältigt, so dass sich lokale klonale DNA-Kolonien, später als "DNA-Cluster" bezeichnet, bilden. Um die Sequenz zu bestimmen, werden vier Arten von reversiblen Terminatorbasen (RT-Basen) hinzugefügt und nicht eingebaute Nukleotide weggewaschen. Eine Kamera nimmt Bilder von den fluoreszierend markierten Nukleotiden auf. Anschließend wird der Farbstoff zusammen mit dem terminalen 3'-Blocker chemisch von der DNA entfernt, so dass der nächste Zyklus beginnen kann. Anders als bei der Pyrosequenzierung werden die DNA-Ketten jeweils um ein Nukleotid verlängert, und die Bildaufnahme kann zu einem späteren Zeitpunkt erfolgen, so dass sehr große Arrays von DNA-Kolonien mit aufeinanderfolgenden Bildern von einer einzigen Kamera erfasst werden können. ⓘ

Die Entkopplung der enzymatischen Reaktion und der Bilderfassung ermöglicht einen optimalen Durchsatz und eine theoretisch unbegrenzte Sequenzierkapazität. Bei einer optimalen Konfiguration wird der letztlich erreichbare Gerätedurchsatz somit allein durch die Analog-Digital-Wandlungsrate der Kamera bestimmt, multipliziert mit der Anzahl der Kameras und dividiert durch die Anzahl der Pixel pro DNA-Kolonie, die für deren optimale Visualisierung erforderlich sind (etwa 10 Pixel/Kolonie). Im Jahr 2012 kann der Durchsatz mit Kameras, die mit einer A/D-Wandlungsrate von mehr als 10 MHz arbeiten, und mit der verfügbaren Optik, Fluidik und Enzymatik ein Vielfaches von 1 Million Nukleotiden/Sekunde betragen, was in etwa einem Äquivalent von 1 Humangenom bei 1-facher Abdeckung pro Stunde pro Gerät entspricht, und einem Humangenom, das (bei ca. 30-facher Abdeckung) pro Tag pro Gerät (ausgestattet mit einer einzigen Kamera) resequenziert wird. ⓘ
Kombinatorische Sondenankersynthese (cPAS)
Bei dieser Methode handelt es sich um eine verbesserte Modifikation der kombinatorischen Sondenanker-Ligations-Technologie (cPAL), die von Complete Genomics beschrieben wurde, das seit 2013 zum chinesischen Genomikunternehmen BGI gehört. Die beiden Unternehmen haben die Technologie verfeinert, um größere Leselängen, kürzere Reaktionszeiten und schnellere Ergebnisse zu ermöglichen. Darüber hinaus werden die Daten nun als zusammenhängende Voll-Leseabschnitte im Standard-FASTQ-Dateiformat generiert und können in den meisten Short-Read-basierten Bioinformatik-Pipelines unverändert verwendet werden. ⓘ
Die beiden Technologien, die die Grundlage für diese Hochdurchsatz-Sequenzierungstechnologie bilden, sind DNA-Nanokugeln (DNB) und strukturierte Arrays für die Befestigung der Nanokugeln an einer festen Oberfläche. DNS-Nanokugeln werden einfach durch Denaturierung doppelsträngiger, mit Adaptern ligierter Bibliotheken und Ligierung nur des Vorwärtsstrangs an ein Splint-Oligonukleotid zur Bildung eines ssDNA-Kreises gebildet. Treue Kopien der Kreise, die das DNA-Insert enthalten, werden mit Hilfe der Rolling-Circle-Amplifikation hergestellt, die etwa 300-500 Kopien erzeugt. Der lange Strang der ssDNA faltet sich um sich selbst, um eine dreidimensionale Nanoball-Struktur mit einem Durchmesser von etwa 220 nm zu erzeugen. Die Herstellung von DNBs ersetzt die Notwendigkeit, PCR-Kopien der Bibliothek auf der Fließzelle zu erzeugen, und kann so einen großen Anteil an doppelten Reads, Adapter-Adapter-Ligationen und PCR-bedingten Fehlern beseitigen. ⓘ

Die strukturierte Anordnung positiv geladener Spots wird durch Photolithographie und Ätztechniken hergestellt, gefolgt von einer chemischen Modifikation, um eine Sequenzierungsfließzelle zu erzeugen. Jeder Spot auf der Fließzelle hat einen Durchmesser von etwa 250 nm und einen Abstand von 700 nm (von Mitte zu Mitte) und ermöglicht die einfache Anbringung einer einzelnen negativ geladenen DNB an der Fließzelle, wodurch eine Unter- oder Überhäufung der Fließzelle vermieden wird. ⓘ
Die Sequenzierung wird dann durch Zugabe einer Oligonukleotid-Sonde durchgeführt, die sich in Kombination an spezifische Stellen innerhalb der DNB anlagert. Die Sonde fungiert als Anker, der dann die Bindung eines von vier einzelnen reversibel inaktivierten, markierten Nukleotiden ermöglicht, nachdem sie durch die Fließzelle geflossen ist. Ungebundene Nukleotide werden weggewaschen, bevor die Laseranregung der angehängten Markierungen Fluoreszenz ausstrahlt und das Signal von Kameras erfasst wird, die es in einen digitalen Ausgang für die Basenbestimmung umwandeln. Am Ende des Zyklus werden der Terminator und die Markierung der angehängten Base chemisch abgespalten. Der Zyklus wird mit einem weiteren Fluss freier, markierter Nukleotide durch die Fließzelle wiederholt, damit das nächste Nukleotid binden und sein Signal erfassen kann. Dieser Vorgang wird mehrere Male durchgeführt (in der Regel 50 bis 300 Mal), um die Sequenz des eingefügten DNA-Stücks mit einer Geschwindigkeit von etwa 40 Millionen Nukleotiden pro Sekunde (Stand 2018) zu bestimmen. ⓘ
SOLiD-Sequenzierung


Bei der SOLiD-Technologie von Applied Biosystems (jetzt eine Marke von Life Technologies) wird die Sequenzierung durch Ligation durchgeführt. Dabei wird ein Pool aller möglichen Oligonukleotide mit einer festen Länge entsprechend der sequenzierten Position markiert. Die Oligonukleotide werden aneinander gebunden und ligiert; die bevorzugte Ligation durch die DNA-Ligase für übereinstimmende Sequenzen führt zu einem Signal, das über das Nukleotid an dieser Position informiert. Jede Base in der Vorlage wird zweimal sequenziert, und die resultierenden Daten werden nach dem bei dieser Methode verwendeten 2-Basen-Kodierungsschema entschlüsselt. Vor der Sequenzierung wird die DNA durch Emulsions-PCR amplifiziert. Die so entstandenen Kügelchen, die jeweils einzelne Kopien desselben DNA-Moleküls enthalten, werden auf einem Objektträger abgelagert. Das Ergebnis sind Sequenzen in Mengen und Längen, die mit der Illumina-Sequenzierung vergleichbar sind. Es wurde berichtet, dass diese Sequenzierungsmethode durch Ligation einige Probleme bei der Sequenzierung palindromischer Sequenzen aufweist. ⓘ
Ion Torrent Halbleiter-Sequenzierung
Ion Torrent Systems Inc. (jetzt im Besitz von Life Technologies) hat ein System entwickelt, das auf der Verwendung von Standard-Sequenzierungschemie basiert, jedoch mit einem neuartigen, halbleiterbasierten Detektionssystem. Diese Sequenzierungsmethode basiert auf dem Nachweis von Wasserstoffionen, die während der Polymerisation der DNA freigesetzt werden, im Gegensatz zu den optischen Methoden, die in anderen Sequenzierungssystemen verwendet werden. Eine Mikrovertiefung, die einen zu sequenzierenden DNA-Strang enthält, wird mit einem einzigen Nukleotidtyp geflutet. Wenn das eingeführte Nukleotid komplementär zum führenden Template-Nukleotid ist, wird es in den wachsenden komplementären Strang eingebaut. Dies bewirkt die Freisetzung eines Wasserstoffions, das einen überempfindlichen Ionensensor auslöst, der anzeigt, dass eine Reaktion stattgefunden hat. Sind in der Matrizensequenz homopolymere Wiederholungen vorhanden, werden mehrere Nukleotide in einem einzigen Zyklus eingebaut. Dies führt zu einer entsprechenden Anzahl von freigesetzten Wasserstoffatomen und einem proportional höheren elektronischen Signal. ⓘ

DNA-Nanoball-Sequenzierung
Bei der DNA-Nanoball-Sequenzierung handelt es sich um eine Hochdurchsatz-Sequenzierungstechnologie, mit der die gesamte genomische Sequenz eines Organismus bestimmt werden kann. Das Unternehmen Complete Genomics nutzt diese Technologie zur Sequenzierung von Proben, die von unabhängigen Forschern eingereicht wurden. Die Methode nutzt die Rolling-Circle-Replikation, um kleine Fragmente genomischer DNA in DNA-Nanokugeln zu amplifizieren. Die Nukleotidsequenz wird dann durch Ligationssequenzierung ohne Verkettung bestimmt. Diese Methode der DNA-Sequenzierung ermöglicht die Sequenzierung einer großen Anzahl von DNA-Nanokugeln pro Durchlauf und im Vergleich zu anderen Hochdurchsatz-Sequenzierungsplattformen zu geringen Reagenzienkosten. Allerdings werden von jedem DNA-Nanoball nur kurze DNA-Sequenzen bestimmt, was die Zuordnung der kurzen Reads zu einem Referenzgenom erschwert. Diese Technologie wurde bereits für mehrere Genomsequenzierungsprojekte eingesetzt und soll für weitere Projekte genutzt werden. ⓘ
Heliscope Einzelmolekül-Sequenzierung
Die Heliskop-Sequenzierung ist eine von Helicos Biosciences entwickelte Methode der Einzelmolekül-Sequenzierung. Sie verwendet DNA-Fragmente mit angefügten Poly-A-Schwanzadaptern, die an der Oberfläche der Fließzelle angebracht werden. Die nächsten Schritte umfassen die verlängerungsbasierte Sequenzierung mit zyklischen Waschvorgängen der Fließzelle mit fluoreszenzmarkierten Nukleotiden (ein Nukleotidtyp nach dem anderen, wie bei der Sanger-Methode). Die Reads werden mit dem Heliscope-Sequenzer durchgeführt. Die Reads sind kurz, im Durchschnitt 35 bp. Das Besondere an dieser Technologie war, dass sie die erste ihrer Klasse war, die nicht-amplifizierte DNA sequenzierte und damit Lesefehler im Zusammenhang mit Amplifikationsschritten verhinderte. Im Jahr 2009 wurde ein menschliches Genom mit dem Heliscope sequenziert, doch 2012 ging das Unternehmen in Konkurs. ⓘ
Mikrofluidische Systeme
Es gibt zwei wichtige mikrofluidische Systeme, die für die DNA-Sequenzierung verwendet werden: tröpfchenbasierte Mikrofluidik und digitale Mikrofluidik. Mit mikrofluidischen Geräten lassen sich viele der derzeitigen Einschränkungen der aktuellen Sequenzierungsarrays überwinden. ⓘ
Abate et al. untersuchten die Verwendung von Mikrofluidikgeräten auf Tröpfchenbasis für die DNA-Sequenzierung. Diese Geräte sind in der Lage, Tröpfchen von Pikolitergröße mit einer Geschwindigkeit von Tausenden pro Sekunde zu bilden und zu verarbeiten. Die Geräte wurden aus Polydimethylsiloxan (PDMS) hergestellt und nutzten den Forster-Resonanzenergietransfer (FRET), um die in den Tröpfchen enthaltenen DNA-Sequenzen zu lesen. Jede Position auf dem Array wurde auf eine bestimmte 15-Basen-Sequenz getestet. ⓘ
Fair et al. verwendeten digitale Mikrofluidikgeräte zur Untersuchung der DNA-Pyrosequenzierung. Zu den wesentlichen Vorteilen gehören die Tragbarkeit des Geräts, das Reagenzvolumen, die Geschwindigkeit der Analyse, die Möglichkeit der Massenproduktion und der hohe Durchsatz. Diese Studie lieferte einen Konzeptnachweis, der zeigt, dass digitale Geräte für die Pyrosequenzierung verwendet werden können; die Studie umfasste die Verwendung der Synthese, die die Verlängerung der Enzyme und die Zugabe von markierten Nukleotiden beinhaltet. ⓘ
Boles et al. untersuchten auch die Pyrosequenzierung auf digitalen Mikrofluidikgeräten. Sie verwendeten ein Elektrobenetzungsgerät, um Tröpfchen zu erzeugen, zu mischen und zu teilen. Bei der Sequenzierung werden ein Drei-Enzym-Protokoll und mit Magnetkügelchen verankerte DNA-Vorlagen verwendet. Das Gerät wurde mit zwei Protokollen getestet und ergab eine Genauigkeit von 100 % auf der Grundlage der Rohpyrogrammwerte. Zu den Vorteilen dieser digitalen mikrofluidischen Geräte gehören die Größe, die Kosten und das erreichbare Niveau der funktionalen Integration. ⓘ
Die Forschung im Bereich der DNA-Sequenzierung mit Hilfe der Mikrofluidik kann auch auf die Sequenzierung von RNA angewendet werden, wobei ähnliche mikrofluidische Tröpfchentechniken wie die Methode inDrops zum Einsatz kommen. Dies zeigt, dass viele dieser DNA-Sequenzierungstechniken weiter angewendet werden können, um mehr über Genome und Transkriptome zu erfahren. ⓘ
Methoden in der Entwicklung
Zu den derzeit in der Entwicklung befindlichen DNA-Sequenzierungsmethoden gehören das Ablesen der Sequenz beim Durchgang eines DNA-Strangs durch Nanoporen (eine Methode, die inzwischen kommerziell ist, aber nachfolgende Generationen wie Festkörper-Nanoporen befinden sich noch in der Entwicklung) und auf Mikroskopie basierende Techniken wie die Rasterkraftmikroskopie oder die Transmissionselektronenmikroskopie, die zur Identifizierung der Positionen einzelner Nukleotide innerhalb langer DNA-Fragmente (>5.000 bp) durch Nukleotidmarkierung mit schwereren Elementen (z. B. Halogenen) zur visuellen Erkennung und Aufzeichnung eingesetzt werden. Die Technologien der dritten Generation zielen darauf ab, den Durchsatz zu erhöhen, die Zeit bis zum Ergebnis zu verkürzen und die Kosten zu senken, indem sie den Bedarf an überflüssigen Reagenzien eliminieren und die Prozessivität der DNA-Polymerase nutzbar machen. ⓘ
DNA-Sequenzierung durch Tunnelströme
Ein weiterer Ansatz besteht in der Messung der elektrischen Tunnelströme durch die einzelsträngige DNA, während sie sich durch einen Kanal bewegt. Je nach ihrer elektronischen Struktur beeinflusst jede Base den Tunnelstrom unterschiedlich, was eine Unterscheidung zwischen verschiedenen Basen ermöglicht. ⓘ
Die Verwendung von Tunnelströmen hat das Potenzial, die Sequenzierung um Größenordnungen schneller durchzuführen als Ionenstrommethoden, und die Sequenzierung mehrerer DNA-Oligomere und Mikro-RNA ist bereits gelungen. ⓘ
Sequenzierung durch Hybridisierung
Die Sequenzierung durch Hybridisierung ist eine nicht-enzymatische Methode, bei der ein DNA-Mikroarray verwendet wird. Ein einzelner DNA-Pool, dessen Sequenz bestimmt werden soll, wird fluoreszierend markiert und an einen Array mit bekannten Sequenzen hybridisiert. Starke Hybridisierungssignale von einem bestimmten Punkt auf dem Array identifizieren dessen Sequenz in der zu sequenzierenden DNA. ⓘ
Bei dieser Methode der Sequenzierung werden die Bindungseigenschaften einer Bibliothek kurzer einzelsträngiger DNA-Moleküle (Oligonukleotide), auch DNA-Sonden genannt, genutzt, um eine Ziel-DNA-Sequenz zu rekonstruieren. Unspezifische Hybride werden durch Waschen entfernt und die Ziel-DNA wird eluiert. Die Hybride werden neu angeordnet, so dass die DNA-Sequenz rekonstruiert werden kann. Der Vorteil dieses Sequenzierungstyps ist die Fähigkeit, eine große Anzahl von Targets mit einer homogenen Abdeckung zu erfassen. In der Regel ist eine große Anzahl von Chemikalien und Ausgangs-DNA erforderlich. Mit dem Aufkommen der lösungsbasierten Hybridisierung sind jedoch viel weniger Geräte und Chemikalien erforderlich. ⓘ
Sequenzierung mit Massenspektrometrie
Die Massenspektrometrie kann zur Bestimmung von DNA-Sequenzen eingesetzt werden. Die MALDI-TOF-Massenspektrometrie (Matrix-Assisted Laser Desorption Ionization Time-of-Flight MS) wurde speziell als alternative Methode zur Gelelektrophorese für die Visualisierung von DNA-Fragmenten untersucht. Bei dieser Methode werden DNA-Fragmente, die durch Sequenzierungsreaktionen mit Kettenabbruch erzeugt wurden, nach ihrer Masse und nicht nach ihrer Größe verglichen. Die Masse jedes Nukleotids unterscheidet sich von den anderen, und dieser Unterschied ist durch Massenspektrometrie nachweisbar. Einzelne Nukleotidmutationen in einem Fragment lassen sich mit MS leichter nachweisen als durch Gelelektrophorese allein. MALDI-TOF MS kann Unterschiede zwischen RNA-Fragmenten leichter nachweisen, so dass Forscher mit MS-basierten Methoden indirekt DNA sequenzieren können, indem sie sie zunächst in RNA umwandeln. ⓘ
Die höhere Auflösung von DNA-Fragmenten, die durch MS-basierte Methoden ermöglicht wird, ist für Forscher in der Forensik von besonderem Interesse, da sie möglicherweise Einzel-Nukleotid-Polymorphismen in menschlichen DNA-Proben finden möchten, um Personen zu identifizieren. Diese Proben können stark degradiert sein, so dass forensische Forscher oft mitochondriale DNA wegen ihrer höheren Stabilität und ihrer Anwendung für Abstammungsstudien bevorzugen. MS-basierte Sequenzierungsmethoden wurden eingesetzt, um die Sequenzen menschlicher mitochondrialer DNA aus Proben in einer Datenbank des Federal Bureau of Investigation und aus Knochen zu vergleichen, die in Massengräbern von Soldaten des Ersten Weltkriegs gefunden wurden. ⓘ
Frühe Kettenterminations- und TOF-MS-Methoden zeigten Leselängen von bis zu 100 Basenpaaren. Forscher waren nicht in der Lage, diese durchschnittliche Leselänge zu überschreiten; wie die Kettenterminationssequenzierung allein ist auch die MS-basierte DNA-Sequenzierung möglicherweise nicht für große de novo-Sequenzierungsprojekte geeignet. Dennoch wurden in einer kürzlich durchgeführten Studie die kurzen Sequenzlesungen und die Massenspektroskopie zum Vergleich von Einzelnukleotid-Polymorphismen in pathogenen Streptokokkenstämmen verwendet. ⓘ
Mikrofluidische Sanger-Sequenzierung
Bei der mikrofluidischen Sanger-Sequenzierung erfolgt die gesamte thermozyklische Amplifikation von DNA-Fragmenten sowie deren Auftrennung durch Elektrophorese auf einem einzigen Glaswafer (mit einem Durchmesser von etwa 10 cm), wodurch der Reagenzienverbrauch und die Kosten reduziert werden. In einigen Fällen haben Forscher gezeigt, dass sie den Durchsatz der herkömmlichen Sequenzierung durch den Einsatz von Mikrochips erhöhen können. Es muss noch geforscht werden, um diesen Technologieeinsatz effektiv zu gestalten. ⓘ
Mikroskopie-basierte Techniken
Bei diesem Ansatz wird die Sequenz von DNA-Molekülen mit Hilfe der Elektronenmikroskopie direkt sichtbar gemacht. Die erste Identifizierung von DNA-Basenpaaren innerhalb intakter DNA-Moleküle durch enzymatischen Einbau modifizierter Basen, die Atome mit erhöhter Ordnungszahl enthalten, sowie die direkte Visualisierung und Identifizierung individuell markierter Basen innerhalb eines synthetischen DNA-Moleküls mit 3.272 Basenpaaren und eines viralen Genoms mit 7.249 Basenpaaren wurde nachgewiesen. ⓘ
RNAP-Sequenzierung
Diese Methode basiert auf der Verwendung von RNA-Polymerase (RNAP), die an ein Polystyrol-Bead gebunden ist. Ein Ende der zu sequenzierenden DNA ist an einem anderen Kügelchen befestigt, wobei beide Kügelchen in optische Fallen gestellt werden. Durch die RNAP-Bewegung während der Transkription nähern sich die Kügelchen an und ihr relativer Abstand ändert sich, was dann mit einer Auflösung von einem Nukleotid aufgezeichnet werden kann. Ähnlich wie bei der Sanger-Methode wird die Sequenz auf der Grundlage der vier Ablesungen mit verringerten Konzentrationen jedes der vier Nukleotidtypen abgeleitet. Es wird ein Vergleich zwischen den Regionen durchgeführt, und die Sequenzinformationen werden durch den Vergleich der bekannten Sequenzregionen mit den unbekannten Sequenzregionen abgeleitet. ⓘ
In-vitro-Virus-Hochdurchsatz-Sequenzierung
Es wurde eine Methode entwickelt, um vollständige Sätze von Proteininteraktionen mit einer Kombination aus 454-Pyrosequenzierung und einer In-vitro-Virus-mRNA-Display-Methode zu analysieren. Bei dieser Methode werden die interessierenden Proteine kovalent an die mRNAs, die sie kodieren, gebunden, und die mRNA-Stücke werden dann mit Hilfe von reversen Transkriptions-PCRs nachgewiesen. Die mRNA kann dann amplifiziert und sequenziert werden. Die kombinierte Methode wurde als IVV-HiTSeq bezeichnet und kann unter zellfreien Bedingungen durchgeführt werden, obwohl ihre Ergebnisse möglicherweise nicht repräsentativ für In-vivo-Bedingungen sind. ⓘ
Probenvorbereitung
Der Erfolg eines jeden DNA-Sequenzierungsprotokolls hängt von der DNA- oder RNA-Probenextraktion und -vorbereitung aus dem biologischen Material von Interesse ab.
- Bei einer erfolgreichen DNA-Extraktion wird eine DNA-Probe mit langen, nicht abgebauten Strängen gewonnen.
- Eine erfolgreiche RNA-Extraktion ergibt eine RNA-Probe, die mit Hilfe der reversen Transkriptase in komplementäre DNA (cDNA) umgewandelt werden sollte - eine DNA-Polymerase, die eine komplementäre DNA auf der Grundlage der vorhandenen RNA-Stränge in einer PCR-ähnlichen Weise synthetisiert. Die komplementäre DNA kann dann auf die gleiche Weise verarbeitet werden wie genomische DNA. ⓘ
Je nach der zu verwendenden Sequenzierungstechnologie müssen die aus der DNA- oder RNA-Extraktion resultierenden Proben weiter aufbereitet werden. Für die Sanger-Sequenzierung sind entweder Klonierungsverfahren oder PCR vor der Sequenzierung erforderlich. Bei den Sequenzierungsmethoden der nächsten Generation ist vor der Verarbeitung eine Bibliotheksvorbereitung erforderlich. Die Bewertung der Qualität und Quantität der Nukleinsäuren sowohl nach der Extraktion als auch nach der Bibliotheksvorbereitung identifiziert degradierte, fragmentierte und niedrigreine Proben und liefert qualitativ hochwertige Sequenzierungsdaten. ⓘ
Der hohe Durchsatz der derzeitigen DNA/RNA-Sequenzierungstechnologien stellt eine Herausforderung für die Skalierung der Probenvorbereitungsmethode dar. Verschiedene Liquid-Handling-Instrumente werden für die Vorbereitung einer größeren Anzahl von Proben mit einem geringeren Zeitaufwand eingesetzt:
| Unternehmen | Flüssighandhabungsgeräte / Automatisierung | untere_marke_USD | obere_marke_USD | landen_url ⓘ |
| Opentrons | OffenTrons OT-2 | $5,750 | $20,000 | https://www.opentrons.com/</nowiki> |
| Gilson | Gilson Pipetmax | $20,000 | $40,000 | https://gb.gilson.com/GBSV/system-pipetmax.html</nowiki> |
| Neotec | Neotec EzMate | $25,000 | $45,000 | http://neotec.co.il/pipetting-device/</nowiki> |
| Formulatrix | Formulatrix Mantis | $40,000 | $60,000 | https://formulatrix.com/liquid-handling-systems/mantis-liquid-handler/</nowiki> |
| Hudson Robotertechnik | Hudson Robotertechnik SOLO | $40,000 | $50,000 | https://hudsonrobotics.com/products/applications/automated-solutions-next-generation-sequencing-ngs/</nowiki> |
| Hamilton | Hamilton Mikrolabor NIMBUS | $40,000 | $80,000 | https://www.hamiltoncompany.com/automated-liquid-handling/platforms/microlab-nimbus#specifications</nowiki> |
| TTP Labortechnik | TTP Labtech Stechmücke HV Genomics | $45,000 | $80,000 | https://www.sptlabtech.com/products/liquid-handling/mosquito-hv-genomics/</nowiki> |
| Beckman Coulter | Biomek 4000 | $50,000 | $65,000 | https://www.mybeckman.uk/liquid-handlers/biomek-4000/b22640</nowiki> |
| Hamilton | Hamilton Genomic STARlet | $50,000 | $100,000 | https://www.hamiltoncompany.com/automated-liquid-handling/assay-ready-workstations/genomic-starlet</nowiki> |
| Eppendorf | Eppendorf epMotion 5075t | $95,000 | $110,000 | https://www.eppendorf.com/epmotion/</nowiki> |
| Beckman Coulter | Beckman Coulter Biomek i5 | $100,000 | $150,000 | https://www.beckman.com/liquid-handlers/biomek-i5</nowiki> |
| Hamilton | Hamilton NGS STAR | $100,000 | $200,000 | http://www.hamiltonrobotics.com/</nowiki> |
| PerkinElmer | PerkinElmer Sciclone G3 NGS und NGSx Arbeitsstation | $150,000 | $220,000 | https://www.perkinelmer.com/uk/product/sciclone-g3-ngs-workstation-cls145321</nowiki> |
| Agilent | Agilent Bravo NGS | $170,000 | $290,000 | https://www.agilent.com/en/products/automated-liquid-handling/automated-liquid-handling-applications/bravo-ngs</nowiki> |
| Beckman Coulter | Beckman Coulter Biomek i7 | $200,000 | $250,000 | https://www.beckman.com/liquid-handlers/biomek-i7</nowiki> |
| Labcyte Echo 525 | Beckman Coulter Labcyte Echo 525 | $260,000 | $300,000 | https://www.labcyte.com/products/liquid-handling/echo-525-liquid-handler</nowiki> |
| Tecan | Tecan NGS | $270,000 | $350,000 | https://lifesciences.tecan.com/ngs-sample-preparation</nowiki> |
Entwicklungsinitiativen

Im Oktober 2006 gründete die X Prize Foundation eine Initiative zur Förderung der Entwicklung von Technologien für die vollständige Genomsequenzierung, den so genannten Archon X Prize, mit dem Ziel, 10 Millionen Dollar an "das erste Team zu vergeben, das ein Gerät bauen und verwenden kann, um 100 menschliche Genome innerhalb von 10 Tagen oder weniger zu sequenzieren, mit einer Genauigkeit von nicht mehr als einem Fehler pro 100.000 sequenzierten Basen, mit Sequenzen, die mindestens 98 % des Genoms genau abdecken, und mit wiederkehrenden Kosten von nicht mehr als 10.000 Dollar (US) pro Genom". ⓘ
Das National Human Genome Research Institute (NHGRI) vergibt jedes Jahr Zuschüsse für neue Forschungen und Entwicklungen in der Genomik. Zu den Zuschüssen für 2010 und den Kandidaten für 2011 gehört die Fortsetzung der Arbeit in den Bereichen Mikrofluidik, Polony und basenlastige Sequenzierungsmethoden. ⓘ
Computertechnische Herausforderungen
Die hier beschriebenen Sequenzierungstechnologien erzeugen Rohdaten, die zu längeren Sequenzen, wie z. B. vollständigen Genomen, zusammengesetzt werden müssen (Sequenzmontage). Um dies zu erreichen, gibt es viele rechnerische Herausforderungen, wie z. B. die Auswertung der Rohsequenzdaten, die von Programmen und Algorithmen wie Phred und Phrap vorgenommen wird. Eine weitere Herausforderung sind die sich wiederholenden Sequenzen, die häufig eine vollständige Genomassemblierung verhindern, da sie an vielen Stellen des Genoms vorkommen. Infolgedessen können viele Sequenzen nicht bestimmten Chromosomen zugeordnet werden. Die Produktion von Rohsequenzdaten ist erst der Anfang ihrer detaillierten bioinformatischen Analyse. Dennoch wurden neue Methoden zur Sequenzierung und Korrektur von Sequenzierungsfehlern entwickelt. ⓘ
Read Trimming
Manchmal sind die vom Sequenzer erzeugten Rohdaten nur in einem Bruchteil ihrer Länge korrekt und präzise. Die Verwendung des gesamten Reads kann zu Artefakten in den nachgelagerten Analysen wie Genomassemblierung, SNP-Calling oder Schätzung der Genexpression führen. Es wurden zwei Klassen von Trimmprogrammen eingeführt, die auf fensterbasierten oder auf Laufsummen-Algorithmen basieren. Im Folgenden finden Sie eine unvollständige Liste der derzeit verfügbaren Trimming-Algorithmen mit Angabe der Algorithmusklasse, zu der sie gehören:
| Name des Algorithmus | Typ des Algorithmus | Link ⓘ |
|---|---|---|
| Cutadapt | Laufende Summe | Cutadapt |
| ConDeTri | Fensterbasiert | ConDeTri |
| ERNE-FILTER | Laufende Summe | ERNE-FILTER |
| FASTX Qualitätstrimmer | Fensterbasiert | FASTX Qualitätstrimmer |
| PRINSEQ | Fensterbasiert | PRINSEQ |
| Trimmautomatik | Fensterbasiert | Trimmautomatik |
| SolexaQA | Fensterbasiert | SolexaQA |
| SolexaQA-BWA | Laufende Summe | SolexaQA-BWA |
| Sichel | Fensterbasiert | Sichel |
Ethische Fragen
Die Humangenetik gehört seit den frühen 1970er Jahren zum Bereich der Bioethik, und der zunehmende Einsatz der DNA-Sequenzierung (insbesondere der Hochdurchsatz-Sequenzierung) hat eine Reihe ethischer Fragen aufgeworfen. Eine zentrale Frage ist das Eigentum an der DNA einer Person und an den Daten, die bei der Sequenzierung dieser DNA entstehen. Was das DNA-Molekül selbst betrifft, so hat der führende Rechtsfall zu diesem Thema, Moore v. Regents of the University of California (1990), entschieden, dass Einzelpersonen keine Eigentumsrechte an weggeworfenen Zellen oder an den mit diesen Zellen erzielten Gewinnen (z. B. als patentierte Zelllinie) haben. Der Einzelne hat jedoch ein Recht auf eine informierte Zustimmung zur Entnahme und Verwendung von Zellen. Was die durch die DNA-Sequenzierung gewonnenen Daten angeht, so gibt Moore dem Einzelnen keine Rechte an den aus seiner DNA gewonnenen Informationen. ⓘ
Mit der zunehmenden Verbreitung der DNA-Sequenzierung hat auch die Speicherung, Sicherheit und Weitergabe von Genomdaten an Bedeutung gewonnen. Eine Befürchtung ist zum Beispiel, dass Versicherer die Genomdaten einer Person nutzen könnten, um ihr Angebot zu ändern, je nachdem, wie der zukünftige Gesundheitszustand der Person auf der Grundlage ihrer DNA eingeschätzt wird. Im Mai 2008 wurde in den Vereinigten Staaten der Genetic Information Nondiscrimination Act (GINA) unterzeichnet, der die Diskriminierung aufgrund von genetischen Informationen in der Krankenversicherung und bei der Beschäftigung verbietet. Im Jahr 2012 berichtete die US Presidential Commission for the Study of Bioethical Issues, dass die bestehenden Datenschutzgesetze für DNA-Sequenzierungsdaten wie GINA und der Health Insurance Portability and Accountability Act unzureichend seien, und wies darauf hin, dass Daten aus der Ganzgenomsequenzierung besonders sensibel seien, da sie dazu verwendet werden könnten, nicht nur die Person zu identifizieren, von der die Daten stammen, sondern auch deren Verwandte. ⓘ
In den meisten Bundesstaaten der Vereinigten Staaten darf "herrenlose" DNA, die z. B. auf einer abgeleckten Briefmarke oder einem Briefumschlag, einer Kaffeetasse, einer Zigarette, einem Kaugummi, im Hausmüll oder in Haaren, die auf einen öffentlichen Gehweg gefallen sind, gefunden wird, von jedermann legal gesammelt und sequenziert werden, einschließlich der Polizei, privater Ermittler, politischer Gegner oder Personen, die in Vaterschaftsstreitigkeiten verwickelt sind. Seit 2013 gibt es in elf Staaten Gesetze, die so ausgelegt werden können, dass sie den "DNA-Diebstahl" verbieten. ⓘ
Ethische Fragen wurden auch durch den zunehmenden Einsatz von Tests auf genetische Variationen aufgeworfen, sowohl bei Neugeborenen als auch bei Erwachsenen durch Unternehmen wie 23andMe. Es wurde behauptet, dass das Screening auf genetische Variationen schädlich sein kann und bei Personen, bei denen ein erhöhtes Krankheitsrisiko festgestellt wurde, Ängste auslöst. In einem Fall, über den in der Zeitschrift Time berichtet wurde, entschieden sich beispielsweise Ärzte, die ein krankes Baby auf genetische Varianten untersuchten, die Eltern nicht über eine nicht verwandte Variante zu informieren, die mit Demenz in Verbindung gebracht wurde, da dies den Eltern schaden würde. Eine Studie aus dem Jahr 2011, die im New England Journal of Medicine veröffentlicht wurde, hat jedoch gezeigt, dass Personen, die sich einem Krankheitsrisikoprofil unterzogen, keine erhöhten Angstzustände aufwiesen. ⓘ
Sequenzierungsmethoden

Es gibt heute mehrere Verfahren zum Ablesen der Sequenzinformation von einem DNA-Molekül. Lange Zeit waren überwiegend Weiterentwicklungen der Methode nach Frederick Sanger in Verwendung. Moderne Verfahren bieten Möglichkeiten der beschleunigten Sequenzierung durch hochparallelen Einsatz. Die nach der Sanger-Methode entwickelten Sequenzierungsverfahren werden häufig als Sequenzierung der nächsten Generation (engl. next generation sequencing) bezeichnet. ⓘ
Moderne Ansätze
Mit der zunehmenden Bedeutung der DNA-Sequenzierung in der Forschung und Diagnostik wurden Methoden entwickelt, die einen erhöhten Durchsatz erlauben. Damit ist es nun möglich, das komplette menschliche Genom in etwa 8 Tagen zu sequenzieren. Die entsprechenden Verfahren werden als Sequenzierung der zweiten Generation (engl. second generation sequencing) bezeichnet. Verschiedene Firmen haben Verfahren mit unterschiedlichen Vor- und Nachteilen entwickelt. Außer den hier aufgeführten gibt es noch weitere. Die DNA-Sequenzierung der zweiten Generation wurde von der Zeitschrift Nature Methods zur Methode des Jahres 2007 gekürt. ⓘ
Sequenzierung durch Hybridisierung
Zu diesem Zweck werden auf einem Glasträger (DNA-Chip oder Microarray) kurze DNA-Abschnitte (Oligonukleotide) in Reihen und Spalten fixiert. Die Fragmente der zu sequenzierenden DNA werden mit Farbstoffen markiert und das Fragmentgemisch wird auf der Oligonukleotidmatrix aufgebracht, so dass komplementäre fixierte und freie DNA-Abschnitte miteinander hybridisieren können. Nach dem Auswaschen ungebundener Fragmente lässt sich das Hybridisierungsmuster anhand der Farbmarkierungen und deren Stärke ablesen. Da die Sequenzen der fixierten Oligonukleotide und deren Überlappungsbereiche bekannt sind, kann man letztlich aus dem Farbmuster auf die zugrundeliegende Gesamtsequenz der unbekannten DNA rückschließen. ⓘ
Ionen-Halbleiter-DNA-Sequenzierungssystem
Dieses Verfahren von Ion Torrent nutzt Halbleiterverfahren, um mittels integrierter Schaltkreise eine unmittelbare nicht-optische Genom-Sequenzierung durchzuführen. Dabei werden die Sequenzierungsdaten direkt über die Halbleiterchip-Detektion von Ionen gewonnen, die von vorlageabhängigen DNA-Polymerasen produziert werden. Der dafür verwendete Chip besitzt ionensensitive Feldeffekttransistor-Sensoren, die in einem Raster von 1,2 Mio. Vertiefungen angeordnet sind, in denen die Polymerase-Reaktion stattfindet. Dieses Raster ermöglicht parallele und simultane Detektion unabhängiger Sequenzreaktionen. Dabei kommt die komplementäre Metalloxid-Halbleiter-Technologie (CMOS) zum Einsatz, die eine kostengünstige Reaktion in hoher Messpunkt-Dichte erlaubt. ⓘ
Ein erster solcher Chip wurde von Jonathan Rothberg entwickelt für die erste Next-Generation-Plattform, die 454 GS FlX, die Pyrosequenzierung benutzt (siehe oben). ⓘ
Sequenzierung mit gepaarten Enden
Ein eindeutig identifizierbares Signal erhält man auch über die Erzeugung von kurzen DNA-Stücken aus dem Anfang und Ende einer DNA-Sequenz (engl. Paired End Tag Sequencing, PETS), wenn das Genom bereits vollständig sequenziert wurde. ⓘ
Sequenzierung der dritten Generation
Die Sequenzierung der dritten Generation misst erstmals die Reaktion bei einzelnen Molekülen als Einzelmolekülexperiment, wodurch eine der Sequenzierung vorangehende Amplifikation per PCR entfällt. Dadurch wird die ungleichmäßige Amplifikation durch thermostabile DNA-Polymerasen vermieden, da Polymerasen manche DNA-Sequenzen bevorzugt binden und diese verstärkt replizieren (engl. polymerase bias). Dadurch können manche Sequenzen übersehen werden. Weiterhin kann das Genom einzelner Zellen untersucht werden. Das freigesetzte Signal wird in Echtzeit aufgenommen. Bei der DNA-Sequenzierung der dritten Generation werden, je nach Verfahren, zwei verschiedene Signale aufgezeichnet: Freigesetzte Protonen (als Variante der Halbleitersequenzierung) oder Fluorophore (mit Fluoreszenzdetektor). Die DNA- und RNA-Sequenzierung einzelner Zellen wurde von der Zeitschrift Nature Methods zur Methode des Jahres 2013 gekürt. ⓘ