Genexpression
| Teil einer Serie über ⓘ |
| Biochemie |
|---|
 |
|
|
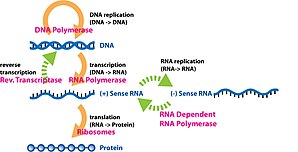
Genexpression ist der Prozess, bei dem die Information eines Gens zur Synthese eines funktionellen Genprodukts verwendet wird, das es ermöglicht, Endprodukte, Proteine oder nicht-kodierende RNA, zu produzieren und schließlich einen Phänotyp als Endwirkung zu beeinflussen. Bei diesen Produkten handelt es sich häufig um Proteine, aber bei nicht-proteinkodierenden Genen wie der Transfer-RNA (tRNA) und der kleinen Kern-RNA (snRNA) ist das Produkt eine funktionelle nicht-kodierende RNA. Die Genexpression wird in dem zentralen Dogma der Molekularbiologie zusammengefasst, das Francis Crick 1958 erstmals formulierte, in seinem Artikel von 1970 weiterentwickelte und durch die späteren Entdeckungen der reversen Transkription und der RNA-Replikation erweitert wurde. ⓘ
Der Prozess der Genexpression wird von allen bekannten Lebewesen - Eukaryoten (einschließlich mehrzelliger Organismen), Prokaryoten (Bakterien und Archaeen) - genutzt und von Viren verwendet, um die makromolekulare Maschinerie des Lebens zu erzeugen. ⓘ
In der Genetik ist die Genexpression die grundlegendste Ebene, auf der der Genotyp zum Phänotyp, d. h. zu einem beobachtbaren Merkmal, führt. Die in der DNA gespeicherte genetische Information stellt den Genotyp dar, während der Phänotyp aus der "Interpretation" dieser Information resultiert. Solche Phänotypen werden häufig durch die Synthese von Proteinen ausgedrückt, die die Struktur und Entwicklung des Organismus steuern oder als Enzyme fungieren, die bestimmte Stoffwechselwege katalysieren. ⓘ
Alle Schritte des Genexpressionsprozesses können moduliert (reguliert) werden, einschließlich der Transkription, des RNA-Spleißens, der Translation und der posttranslationalen Modifikation eines Proteins. Die Regulierung der Genexpression ermöglicht die Kontrolle über den Zeitpunkt, den Ort und die Menge eines bestimmten Genprodukts (Protein oder ncRNA) in einer Zelle und kann tiefgreifende Auswirkungen auf die Zellstruktur und -funktion haben. Die Regulierung der Genexpression ist die Grundlage für die zelluläre Differenzierung, Entwicklung, Morphogenese und die Vielseitigkeit und Anpassungsfähigkeit eines jeden Organismus. Die Genregulation kann daher als Substrat für evolutionäre Veränderungen dienen. ⓘ
Die unterschiedliche Genexpression ist bei (genetisch gleichen) eineiigen Zwillingen eine Ursache ihrer geringfügig verschiedenen Phänotypen. Bei genetisch verschiedenen Individuen basieren die Unterschiede im Phänotyp neben der Modifikation vor allem auf Abweichungen im Genom. Die zeitlichen und räumlichen Unterschiede der Genexpression werden als spatiotemporale Genexpression bezeichnet. ⓘ
Mechanismus
Transkription

Die Herstellung einer RNA-Kopie aus einem DNA-Strang wird als Transkription bezeichnet und erfolgt durch RNA-Polymerasen, die entsprechend dem Komplementaritätsgesetz der Nukleotidbasen ein Ribonukleotid nach dem anderen an einen wachsenden RNA-Strang anhängen. Diese RNA ist komplementär zum 3′ → 5′-DNA-Strang der Vorlage, mit der Ausnahme, dass in der RNA Thymin (T) durch Uracil (U) ersetzt wird. ⓘ
In Prokaryonten wird die Transkription von einem einzigen Typ von RNA-Polymerase durchgeführt, die mit Hilfe des Sigma-Faktor-Proteins (σ-Faktor) eine DNA-Sequenz, die so genannte Pribnow-Box, binden muss, um die Transkription zu starten. In Eukaryonten wird die Transkription im Zellkern von drei Arten von RNA-Polymerasen durchgeführt, von denen jede eine spezielle DNA-Sequenz, den so genannten Promotor, und eine Reihe von DNA-bindenden Proteinen - die Transkriptionsfaktoren - benötigt, um den Prozess in Gang zu setzen (siehe Regulierung der Transkription unten). Die RNA-Polymerase I ist für die Transkription der ribosomalen RNA (rRNA)-Gene verantwortlich. RNA-Polymerase II (Pol II) transkribiert alle proteinkodierenden Gene, aber auch einige nicht-kodierende RNAs (z. B. snRNAs, snoRNAs oder lange nicht-kodierende RNAs). RNA-Polymerase III transkribiert 5S rRNA, Transfer-RNA (tRNA)-Gene und einige kleine nicht-kodierende RNAs (z. B. 7SK). Die Transkription endet, wenn die Polymerase auf eine Sequenz stößt, die als Terminator bezeichnet wird. ⓘ
mRNA-Verarbeitung
Während bei der Transkription prokaryontischer proteinkodierender Gene eine Boten-RNA (mRNA) entsteht, die für die Übersetzung in Protein bereit ist, hinterlässt die Transkription eukaryontischer Gene ein primäres RNA-Transkript (pre-RNA), das zunächst eine Reihe von Modifikationen durchlaufen muss, um zu einer reifen RNA zu werden. Die Arten und Schritte, die an den Reifungsprozessen beteiligt sind, unterscheiden sich zwischen kodierenden und nicht-kodierenden prä-RNAs; d. h. obwohl prä-RNA-Moleküle sowohl für mRNA als auch für tRNA gespleißt werden, sind die beteiligten Schritte und Maschinen unterschiedlich. Die Verarbeitung von nicht-kodierender RNA wird weiter unten beschrieben (Reifung nicht-kodierender RNA). ⓘ
Die Verarbeitung von prä-mRNA umfasst die 5′-Kappung, eine Reihe von enzymatischen Reaktionen, die 7-Methylguanosin (m7G) an das 5′-Ende der prä-mRNA anfügen und so die RNA vor dem Abbau durch Exonukleasen schützen. Die m7G-Kappe wird dann von einem Kappenbindungskomplex-Heterodimer (CBC20/CBC80) gebunden, das den mRNA-Export ins Zytoplasma unterstützt und die RNA auch vor dem Dekappen schützt. ⓘ
Eine weitere Modifikation ist die 3′-Spaltung und Polyadenylierung. Sie treten auf, wenn die Polyadenylierungssignalsequenz (5′- AAUAAA-3′) in der prä-mRNA vorhanden ist, die normalerweise zwischen der proteinkodierenden Sequenz und dem Terminator liegt. Die prä-mRNA wird zunächst gespalten und dann wird eine Reihe von ~200 Adeninen (A) hinzugefügt, um einen Poly(A)-Schwanz zu bilden, der die RNA vor dem Abbau schützt. Der Poly(A)-Schwanz wird von mehreren Poly(A)-bindenden Proteinen (PABPs) gebunden, die für den mRNA-Export und die erneute Initiierung der Translation erforderlich sind. Beim umgekehrten Prozess der Deadenylierung werden die Poly(A)-Schwänze durch die CCR4-Not 3′-5′-Exonuklease verkürzt, was häufig zum vollständigen Zerfall des Transkripts führt. ⓘ

Eine sehr wichtige Modifikation der eukaryotischen prä-mRNA ist das RNA-Spleißen. Die meisten eukaryontischen prä-mRNAs bestehen aus abwechselnden Abschnitten, die Exons und Introns genannt werden. Während des Spleißvorgangs katalysiert ein als Spleißosom bezeichneter RNA-Protein-Katalysatorkomplex zwei Umesterungsreaktionen, durch die ein Intron entfernt und in Form einer Lariatstruktur freigesetzt wird, und spleißt anschließend benachbarte Exons zusammen. In bestimmten Fällen können einige Introns oder Exons in der reifen mRNA entweder entfernt oder beibehalten werden. Durch dieses so genannte alternative Spleißen entsteht eine Reihe unterschiedlicher Transkripte, die von einem einzigen Gen stammen. Da diese Transkripte potenziell in verschiedene Proteine übersetzt werden können, erweitert das Spleißen die Komplexität der eukaryotischen Genexpression und die Größe des Proteoms einer Art. ⓘ
Die umfangreiche RNA-Verarbeitung könnte ein evolutionärer Vorteil sein, der durch den Zellkern von Eukaryonten ermöglicht wird. Bei Prokaryonten finden Transkription und Translation gleichzeitig statt, während bei Eukaryonten die Kernmembran die beiden Prozesse trennt, so dass Zeit für die RNA-Verarbeitung zur Verfügung steht. ⓘ
Reifung der nichtcodierenden RNA
In den meisten Organismen werden nichtcodierende Gene (ncRNA) als Vorläufer transkribiert, die einer weiteren Verarbeitung unterzogen werden. Im Fall der ribosomalen RNAs (rRNA) werden sie oft als prä-rRNA transkribiert, die eine oder mehrere rRNAs enthält. Die prä-rRNA wird von etwa 150 verschiedenen kleinen, auf den Nukleolus beschränkten RNA-Arten, den so genannten snoRNAs, an bestimmten Stellen gespalten und modifiziert (2′-O-Methylierung und Pseudouridinbildung). SnoRNAs assoziieren mit Proteinen und bilden snoRNPs. Während der snoRNA-Teil ein Basenpaar mit der Ziel-RNA bildet und so die Modifikation an einer bestimmten Stelle positioniert, führt der Proteinteil die katalytische Reaktion durch. In Eukaryoten spaltet insbesondere ein snoRNP namens RNase, MRP die 45S pre-rRNA in die 28S, 5.8S und 18S rRNAs. Die rRNA und die RNA-Verarbeitungsfaktoren bilden große Aggregate, die Nukleolus genannt werden. ⓘ
Bei der Transfer-RNA (tRNA) beispielsweise wird die 5′-Sequenz durch RNase P entfernt, während das 3′-Ende durch das Enzym tRNase Z entfernt und der nicht-templierte 3′-CCA-Schwanz durch eine Nukleotidyltransferase angefügt wird. Im Falle der Mikro-RNA (miRNA) werden miRNAs zunächst als primäre Transkripte oder pri-miRNA mit einer Kappe und einem Poly-A-Schwanz transkribiert und im Zellkern durch die Enzyme Drosha und Pasha zu kurzen, 70-Nukleotid-Stammschleifenstrukturen, den so genannten pre-miRNAs, verarbeitet. Nach dem Export wird sie dann im Zytoplasma durch Interaktion mit der Endonuklease Dicer zu reifen miRNAs verarbeitet, die auch die Bildung des RNA-induzierten Silencing-Komplexes (RISC) einleitet, der aus dem Argonaute-Protein besteht. ⓘ
Auch snRNAs und snoRNAs selbst durchlaufen eine Reihe von Modifikationen, bevor sie Teil eines funktionellen RNP-Komplexes werden. Dies geschieht entweder im Nukleoplasma oder in den spezialisierten Kompartimenten, den so genannten Cajal-Körpern. Ihre Basen werden von einer Gruppe kleiner Cajal-Körper-spezifischer RNAs (scaRNAs), die strukturell den snoRNAs ähneln, methyliert oder pseudouridiniliert. ⓘ
RNA-Export
In Eukaryoten müssen die meisten reifen RNAs aus dem Zellkern in das Zytoplasma exportiert werden. Während einige RNAs im Zellkern funktionieren, werden viele RNAs durch die Kernporen in das Zytosol transportiert. Der Export von RNAs erfordert die Assoziation mit spezifischen Proteinen, den so genannten Exportinen. Spezifische Exportin-Moleküle sind für den Export eines bestimmten RNA-Typs verantwortlich. Der mRNA-Transport erfordert auch die korrekte Assoziation mit dem Exon Junction Complex (EJC), der gewährleistet, dass die mRNA vor dem Export korrekt verarbeitet wird. In einigen Fällen werden RNAs zusätzlich zu einem bestimmten Teil des Zytoplasmas transportiert, z. B. zu einer Synapse; sie werden dann von Motorproteinen geschleppt, die über Linkerproteine an bestimmte Sequenzen (so genannte "zipcodes") auf der RNA binden. ⓘ
Übersetzung
Bei einigen RNAs (nicht codierende RNA) ist die reife RNA das endgültige Genprodukt. Im Falle der Boten-RNA (mRNA) ist die RNA ein Informationsträger, der für die Synthese eines oder mehrerer Proteine kodiert. mRNA, die eine einzige Proteinsequenz trägt (häufig bei Eukaryoten), ist monocistronisch, während mRNA, die mehrere Proteinsequenzen trägt (häufig bei Prokaryoten), als polycistronisch bezeichnet wird. ⓘ

Jede mRNA besteht aus drei Teilen: einer 5′ untranslatierten Region (5′UTR), einer proteinkodierenden Region oder einem offenen Leseraster (ORF) und einer 3′ untranslatierten Region (3′UTR). Die kodierende Region enthält Informationen für die Proteinsynthese, die durch den genetischen Code in Form von Tripletts kodiert werden. Jedes Nukleotid-Triplett der kodierenden Region wird als Codon bezeichnet und entspricht einer Bindungsstelle, die komplementär zu einem Anticodon-Triplett in der Transfer-RNA ist. Transfer-RNAs mit der gleichen Anticodon-Sequenz tragen immer eine identische Art von Aminosäure. Die Aminosäuren werden dann vom Ribosom entsprechend der Reihenfolge der Tripletts in der kodierenden Region aneinandergereiht. Das Ribosom hilft der Transfer-RNA, sich an die Boten-RNA zu binden, und entnimmt jeder Transfer-RNA eine Aminosäure, aus der es ein strukturloses Protein herstellt. Jedes mRNA-Molekül wird in viele Proteinmoleküle übersetzt, bei Säugetieren im Durchschnitt ~2800. ⓘ
Bei Prokaryonten erfolgt die Translation in der Regel zum Zeitpunkt der Transkription (kotranskriptiv), oft unter Verwendung einer Boten-RNA, die noch im Entstehen begriffen ist. Bei Eukaryonten kann die Translation in verschiedenen Regionen der Zelle stattfinden, je nachdem, wo sich das zu schreibende Protein befinden soll. Die wichtigsten Orte sind das Zytoplasma für lösliche zytoplasmatische Proteine und die Membran des endoplasmatischen Retikulums für Proteine, die aus der Zelle exportiert oder in eine Zellmembran eingebaut werden sollen. Proteine, die im endoplasmatischen Retikulum exprimiert werden sollen, werden während des Übersetzungsprozesses erkannt. Dies wird durch das Signalerkennungspartikel geregelt - ein Protein, das an das Ribosom bindet und es zum endoplasmatischen Retikulum leitet, wenn es ein Signalpeptid auf der wachsenden (naszierenden) Aminosäurekette findet. ⓘ
Faltung

Jedes Protein liegt als ungefaltetes Polypeptid oder zufällige Spule vor, wenn es aus einer mRNA-Sequenz in eine lineare Kette von Aminosäuren übersetzt wird. Diesem Polypeptid fehlt jede entwickelte dreidimensionale Struktur (die linke Seite der nebenstehenden Abbildung). Das Polypeptid faltet sich dann von einer zufälligen Spule zu seiner charakteristischen und funktionellen dreidimensionalen Struktur. Die Aminosäuren interagieren miteinander, um eine wohldefinierte dreidimensionale Struktur zu erzeugen, das gefaltete Protein (rechte Seite der Abbildung), das als nativer Zustand bezeichnet wird. Die resultierende dreidimensionale Struktur wird durch die Aminosäuresequenz bestimmt (Anfinsen-Dogma). ⓘ
Die korrekte dreidimensionale Struktur ist für die Funktion unerlässlich, auch wenn einige Teile funktioneller Proteine ungefaltet bleiben können. Gelingt es nicht, sich in die vorgesehene Form zu falten, entstehen in der Regel inaktive Proteine mit unterschiedlichen Eigenschaften, darunter auch toxische Prionen. Es wird angenommen, dass mehrere neurodegenerative und andere Krankheiten auf die Ansammlung fehlgefalteter Proteine zurückzuführen sind. Viele Allergien werden durch die Faltung der Proteine verursacht, da das Immunsystem für bestimmte Proteinstrukturen keine Antikörper bildet. ⓘ
Enzyme, so genannte Chaperone, helfen dem neu gebildeten Protein, die dreidimensionale Struktur zu erreichen (sich zu falten), die es für seine Funktion benötigt. In ähnlicher Weise helfen RNA-Chaperone den RNAs, ihre funktionelle Form zu erreichen. Die Unterstützung der Proteinfaltung ist eine der Hauptaufgaben des endoplasmatischen Retikulums in Eukaryonten. ⓘ
Translokation
Sekretorische Proteine von Eukaryonten oder Prokaryonten müssen transloziert werden, um in den Sekretionsweg zu gelangen. Neu synthetisierte Proteine werden durch Signalpeptide zum eukaryotischen Sec61- oder prokaryotischen SecYEG-Translokationskanal geleitet. Die Effizienz der Proteinsekretion in Eukaryonten hängt stark von dem verwendeten Signalpeptid ab. ⓘ
Proteintransport
Viele Proteine sind für andere Teile der Zelle als das Zytosol bestimmt, und eine breite Palette von Signalsequenzen (Signalpeptide) wird verwendet, um Proteine dorthin zu leiten, wo sie hingehören. Bei Prokaryonten ist dies aufgrund der begrenzten Kompartimentierung der Zelle normalerweise ein einfacher Prozess. Bei Eukaryonten gibt es jedoch eine große Vielfalt an verschiedenen Zielprozessen, um sicherzustellen, dass das Protein an der richtigen Organelle ankommt. ⓘ
Nicht alle Proteine verbleiben in der Zelle, viele werden exportiert, z. B. Verdauungsenzyme, Hormone und extrazelluläre Matrixproteine. In Eukaryonten ist der Exportweg gut entwickelt, und der Hauptmechanismus für den Export dieser Proteine ist die Translokation in das endoplasmatische Retikulum, gefolgt vom Transport über den Golgi-Apparat. ⓘ
Regulierung der Genexpression

Die Regulation der Genexpression ist die Kontrolle der Menge und des Zeitpunkts des Auftretens des funktionellen Produkts eines Gens. Die Kontrolle der Expression ist von entscheidender Bedeutung, damit eine Zelle die Genprodukte, die sie benötigt, zum richtigen Zeitpunkt produzieren kann; dies wiederum gibt den Zellen die Flexibilität, sich an eine veränderliche Umgebung, externe Signale, Zellschäden und andere Stimuli anzupassen. Ganz allgemein verleiht die Genregulation der Zelle die Kontrolle über alle Strukturen und Funktionen und ist die Grundlage für die zelluläre Differenzierung, die Morphogenese und die Vielseitigkeit und Anpassungsfähigkeit eines jeden Organismus. ⓘ
Es werden zahlreiche Begriffe verwendet, um Gentypen zu beschreiben, je nachdem, wie sie reguliert werden; dazu gehören:
- Ein konstitutives Gen ist ein Gen, das ständig transkribiert wird, im Gegensatz zu einem fakultativen Gen, das nur bei Bedarf transkribiert wird.
- Ein Housekeeping-Gen ist ein Gen, das für die Aufrechterhaltung grundlegender zellulärer Funktionen erforderlich ist und daher normalerweise in allen Zelltypen eines Organismus exprimiert wird. Beispiele hierfür sind Aktin, GAPDH und Ubiquitin. Einige Housekeeping-Gene werden mit einer relativ konstanten Rate transkribiert, und diese Gene können in Experimenten als Referenzpunkt verwendet werden, um die Expressionsraten anderer Gene zu messen.
- Ein fakultatives Gen ist ein Gen, das nur bei Bedarf transkribiert wird, im Gegensatz zu einem konstitutiven Gen.
- Ein induzierbares Gen ist ein Gen, dessen Expression entweder auf Umweltveränderungen reagiert oder von der Position im Zellzyklus abhängig ist. ⓘ
Jeder Schritt der Genexpression kann moduliert werden, vom DNA-RNA-Transkriptionsschritt bis zur posttranslationalen Modifikation eines Proteins. Die Stabilität des endgültigen Genprodukts, egal ob es sich um RNA oder Protein handelt, trägt ebenfalls zum Expressionsniveau des Gens bei - ein instabiles Produkt führt zu einem niedrigen Expressionsniveau. Im Allgemeinen wird die Genexpression durch Veränderungen in der Anzahl und Art der Wechselwirkungen zwischen Molekülen reguliert, die zusammen die Transkription der DNA und die Translation der RNA beeinflussen. ⓘ
Einige einfache Beispiele dafür, wo die Genexpression wichtig ist, sind:
- Kontrolle der Insulinexpression als Signal für die Regulierung des Blutzuckerspiegels.
- Inaktivierung des X-Chromosoms bei weiblichen Säugetieren, um eine "Überdosis" der darin enthaltenen Gene zu verhindern.
- Die Cyclin-Expression steuert das Fortschreiten des eukaryontischen Zellzyklus. ⓘ
Generell kann eine Regulation der Genexpression auf verschiedenen Stufen stattfinden. Dabei können – insbesondere bei Eukaryoten – die genannten Prinzipien miteinander in Wechselwirkung treten und so im Zusammenspiel von Genetik und Epigenetik noch komplexere Regulationsmechanismen bilden. ⓘ
Transkriptionelle Regulierung

Die Regulierung der Transkription lässt sich in drei Haupteinflusswege unterteilen: genetisch (direkte Interaktion eines Kontrollfaktors mit dem Gen), modulierend (Interaktion eines Kontrollfaktors mit der Transkriptionsmaschinerie) und epigenetisch (sequenzunabhängige Veränderungen der DNA-Struktur, die die Transkription beeinflussen). ⓘ
Die direkte Interaktion mit der DNA ist die einfachste und direkteste Methode, mit der ein Protein die Transkriptionsmenge verändert. Gene haben oft mehrere Proteinbindungsstellen in der kodierenden Region mit der spezifischen Funktion, die Transkription zu regulieren. Es gibt viele Klassen von regulatorischen DNA-Bindungsstellen, die als Enhancer, Insulatoren und Silencer bekannt sind. Die Mechanismen zur Regulierung der Transkription sind vielfältig und reichen von der Blockierung von Schlüsselbindungsstellen auf der DNA für die RNA-Polymerase bis hin zur Tätigkeit als Aktivator und Förderung der Transkription durch Unterstützung der RNA-Polymerase-Bindung. ⓘ
Die Aktivität von Transkriptionsfaktoren wird außerdem durch intrazelluläre Signale moduliert, die eine posttranslationale Modifikation des Proteins bewirken, z. B. eine Phosphorylierung, Acetylierung oder Glykosylierung. Diese Veränderungen beeinflussen die Fähigkeit eines Transkriptionsfaktors, direkt oder indirekt an die Promotor-DNA zu binden, die RNA-Polymerase zu rekrutieren oder die Elongation eines neu synthetisierten RNA-Moleküls zu fördern. ⓘ
Die Kernmembran in Eukaryonten ermöglicht eine weitere Regulierung der Transkriptionsfaktoren durch die Dauer ihrer Anwesenheit im Zellkern, die durch reversible Veränderungen ihrer Struktur und durch die Bindung anderer Proteine reguliert wird. Umweltreize oder endokrine Signale können eine Veränderung der regulatorischen Proteine bewirken und Kaskaden intrazellulärer Signale auslösen, die zu einer Regulierung der Genexpression führen. ⓘ
In jüngerer Zeit hat sich gezeigt, dass auch nicht-DNA-Sequenz-spezifische Effekte einen erheblichen Einfluss auf die Transkription haben. Diese Effekte werden als epigenetisch bezeichnet und betreffen die DNA-Struktur höherer Ordnung, nicht sequenzspezifische DNA-Bindungsproteine und chemische Modifikationen der DNA. Im Allgemeinen verändern epigenetische Effekte die Zugänglichkeit der DNA für Proteine und modulieren so die Transkription. ⓘ
In Eukaryonten regelt die Struktur des Chromatins, die durch den Histoncode gesteuert wird, den Zugang zur DNA, was erhebliche Auswirkungen auf die Expression von Genen in Euchromatin- und Heterochromatinbereichen hat. ⓘ
Enhancer, Transkriptionsfaktoren, Mediatorenkomplexe und DNA-Schleifen in der Transkription bei Säugetieren

Die Genexpression bei Säugetieren wird durch zahlreiche cis-regulatorische Elemente reguliert, darunter Kernpromotoren und promotorproximale Elemente, die sich in der Nähe der Transkriptionsstartstellen von Genen stromaufwärts auf der DNA befinden (in Richtung der 5'-Region des Sinnstrangs). Andere wichtige cis-regulatorische Module sind in DNA-Regionen lokalisiert, die von den Transkriptionsstartstellen entfernt sind. Dazu gehören Enhancer, Silencer, Insulatoren und Tethering-Elemente. Enhancer und die mit ihnen verbundenen Transkriptionsfaktoren spielen eine wichtige Rolle bei der Regulierung der Genexpression. ⓘ
Enhancer sind Genomregionen, die Gene regulieren. Enhancer steuern zelltypspezifische Genexpressionsprogramme, indem sie sich meist über weite Strecken in die Nähe der Promotoren ihrer Zielgene schieben. Mehrere Enhancer, die oft Zehn- oder Hunderttausende von Nukleotiden von ihren Zielgenen entfernt sind, schlingen sich zu ihren Zielgen-Promotoren und koordinieren sich gegenseitig, um die Genexpression zu steuern. ⓘ
Die Abbildung zeigt einen Enhancer, der eine Schleife bildet, um in die Nähe des Promotors eines Zielgens zu gelangen. Die Schleife wird durch ein Dimer eines Verbindungsproteins (z. B. ein Dimer von CTCF oder YY1) stabilisiert. Ein Mitglied des Dimers ist an seinem Bindungsmotiv auf dem Enhancer und das andere Mitglied an seinem Bindungsmotiv auf dem Promotor verankert (in der Abbildung durch die roten Zickzacklinien dargestellt). Mehrere zellfunktionsspezifische Transkriptionsfaktoren (unter den etwa 1.600 Transkriptionsfaktoren in einer menschlichen Zelle) binden im Allgemeinen an bestimmte Motive auf einem Enhancer. Eine kleine Kombination dieser an den Enhancer gebundenen Transkriptionsfaktoren steuert, wenn sie durch eine DNA-Schleife in die Nähe eines Promotors gebracht werden, die Transkriptionsmenge des Zielgens. Der Mediator (ein Komplex, der in der Regel aus etwa 26 Proteinen in einer interagierenden Struktur besteht) übermittelt die regulatorischen Signale der an die Enhancer-DNA gebundenen Transkriptionsfaktoren direkt an das an den Promotor gebundene Enzym RNA-Polymerase II (pol II). ⓘ
Wenn Enhancer aktiv sind, werden sie im Allgemeinen von beiden DNA-Strängen transkribiert, wobei die RNA-Polymerasen in zwei verschiedene Richtungen wirken und zwei eRNAs produzieren, wie in der Abbildung dargestellt. Ein inaktiver Enhancer kann durch einen inaktiven Transkriptionsfaktor gebunden sein. Durch Phosphorylierung des Transkriptionsfaktors kann dieser aktiviert werden, und der aktivierte Transkriptionsfaktor kann dann den Enhancer, an den er gebunden ist, aktivieren (siehe kleiner roter Stern, der die Phosphorylierung des an den Enhancer gebundenen Transkriptionsfaktors in der Abbildung darstellt). Ein aktivierter Enhancer beginnt mit der Transkription seiner RNA, bevor er die Transkription der Boten-RNA seines Zielgens aktiviert. ⓘ
DNA-Methylierung und -Demethylierung bei der Transkriptionsregulation

Die DNA-Methylierung ist ein weit verbreiteter Mechanismus für die epigenetische Beeinflussung der Genexpression, der in Bakterien und Eukaryonten vorkommt und eine Rolle beim vererbbaren Transkriptions-Silencing und der Transkriptionsregulation spielt. Die Methylierung findet am häufigsten an einem Cytosin statt (siehe Abbildung). Die Methylierung von Cytosin erfolgt hauptsächlich in Dinukleotidsequenzen, in denen auf ein Cytosin ein Guanin folgt, eine CpG-Stelle. Die Zahl der CpG-Stellen im menschlichen Genom beträgt etwa 28 Millionen. Je nach Zelltyp weisen etwa 70 % der CpG-Stellen ein methyliertes Cytosin auf. ⓘ
Die Methylierung von Cytosin in der DNA spielt eine wichtige Rolle bei der Regulierung der Genexpression. Die Methylierung von CpGs in der Promotorregion eines Gens unterdrückt in der Regel die Gentranskription, während die Methylierung von CpGs im Körper eines Gens die Expression erhöht. TET-Enzyme spielen eine zentrale Rolle bei der Demethylierung von methylierten Cytosinen. Die Demethylierung von CpGs in einem Genpromotor durch TET-Enzymaktivität erhöht die Transkription des Gens. ⓘ
Transkriptionsregulation bei Lernen und Gedächtnis
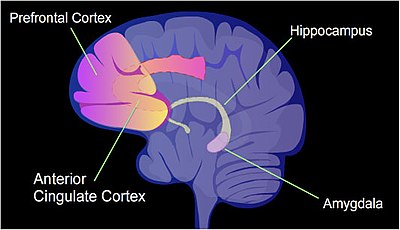
Bei der Ratte ist die kontextuelle Angstkonditionierung (CFC) eine schmerzhafte Lernerfahrung. Schon eine einzige CFC-Episode kann zu einer lebenslangen angstbesetzten Erinnerung führen. Nach einer CFC-Episode ist die Cytosin-Methylierung in den Promotorregionen von etwa 9,17 % aller Gene in der DNA der Neuronen des Hippocampus einer Ratte verändert. Der Hippocampus ist der Ort, an dem neue Erinnerungen zunächst gespeichert werden. Nach CFC haben etwa 500 Gene eine erhöhte Transkription (oft aufgrund der Demethylierung von CpG-Stellen in einer Promotorregion) und etwa 1.000 Gene eine verringerte Transkription (oft aufgrund von neu gebildetem 5-Methylcytosin an CpG-Stellen in einer Promotorregion). Das Muster der induzierten und unterdrückten Gene innerhalb der Neuronen scheint eine molekulare Grundlage für die Bildung des ersten Kurzzeitgedächtnisses dieses Trainingsereignisses im Hippocampus des Rattengehirns zu bilden. ⓘ
Einige spezifische Mechanismen, die neue DNA-Methylierungen und neue DNA-Demethylierungen im Hippocampus während der Gedächtnisbildung steuern, wurden kürzlich nachgewiesen (siehe Zusammenfassung). Ein Mechanismus besteht darin, dass die kurze Isoform des TET1-DNA-Demethylierungsenzyms, TET1s, zu etwa 600 Stellen im Genom gelenkt wird. Die Steuerung erfolgt durch die Assoziation von TET1s mit dem EGR1-Protein, einem für die Gedächtnisbildung wichtigen Transkriptionsfaktor. Wenn TET1s an diese Stellen gebracht wird, wird die DNA-Demethylierung an diesen Stellen eingeleitet und die damit verbundenen Gene werden hochreguliert. Ein zweiter Mechanismus betrifft DNMT3A2, eine Splice-Isoform der DNA-Methyltransferase DNMT3A, die Methylgruppen an Cytosine in der DNA anhängt. Diese Isoform wird durch synaptische Aktivität induziert, und ihr Wirkungsort scheint durch posttranslationale Histon-Modifikationen (einen Histon-Code) bestimmt zu werden. Die resultierenden neuen Boten-RNAs werden dann von Boten-RNP-Partikeln (neuronalen Granula) zu den Synapsen der Neuronen transportiert, wo sie in Proteine übersetzt werden können, die die Aktivitäten der Synapsen beeinflussen. ⓘ
Insbesondere das Gen des Brain-Derived Neurotrophic Factor (BDNF) ist als "Lerngen" bekannt. Nach CFC kam es zu einer Hochregulierung der BDNF-Genexpression, die mit einer verringerten CpG-Methylierung bestimmter interner Promotoren des Gens zusammenhing, und dies korrelierte mit dem Lernen. ⓘ
Transkriptionsregulierung bei Krebs
Die Mehrzahl der Genpromotoren enthält eine CpG-Insel mit zahlreichen CpG-Stellen. Wenn viele der CpG-Promotorstellen eines Gens methyliert sind, wird das Gen stillgelegt. Kolorektale Karzinome weisen in der Regel 3 bis 6 Treibermutationen und 33 bis 66 Anhalter- oder Passagiermutationen auf. Das transkriptionelle Silencing kann jedoch für das Fortschreiten der Krebserkrankung von größerer Bedeutung sein als Mutationen. So werden bei Darmkrebs etwa 600 bis 800 Gene durch CpG-Insel-Methylierung transkriptionell zum Schweigen gebracht (siehe Regulation der Transkription bei Krebs). Die Unterdrückung der Transkription bei Krebs kann auch durch andere epigenetische Mechanismen erfolgen, etwa durch eine veränderte Expression von microRNAs. Bei Brustkrebs kann die transkriptionelle Unterdrückung von BRCA1 häufiger durch eine überexprimierte microRNA-182 als durch eine Hypermethylierung des BRCA1-Promotors auftreten (siehe Geringe Expression von BRCA1 bei Brust- und Eierstockkrebs). ⓘ
Posttranskriptionelle Regulierung
In Eukaryonten, wo der Export von RNA erforderlich ist, bevor eine Translation möglich ist, wird angenommen, dass der Kernexport eine zusätzliche Kontrolle über die Genexpression bietet. Der gesamte Transport in und aus dem Zellkern erfolgt über die Kernpore, und der Transport wird durch eine Vielzahl von Importin- und Exportin-Proteinen kontrolliert. ⓘ
Die Expression eines Gens, das für ein Protein kodiert, ist nur möglich, wenn die Boten-RNA, die den Code trägt, lange genug überlebt, um übersetzt zu werden. In einer typischen Zelle ist ein RNA-Molekül nur dann stabil, wenn es spezifisch vor Abbau geschützt ist. Der RNA-Abbau ist besonders wichtig für die Regulierung der Expression in eukaryontischen Zellen, in denen die mRNA beträchtliche Strecken zurücklegen muss, bevor sie übersetzt wird. In Eukaryoten wird die RNA durch bestimmte posttranskriptionelle Modifikationen stabilisiert, insbesondere durch die 5′-Kappe und den polyadenylierten Schwanz. ⓘ
Der absichtliche Abbau von mRNA dient nicht nur als Abwehrmechanismus gegen fremde RNA (in der Regel von Viren), sondern auch als Weg zur Destabilisierung der mRNA. Wenn ein mRNA-Molekül eine komplementäre Sequenz zu einer kleinen interferierenden RNA aufweist, wird es gezielt über den RNA-Interferenzweg zerstört. ⓘ
Drei primär untranslatierte Regionen und microRNAs
Die nichttranslatierten Regionen (3′UTRs) von Boten-RNAs (mRNAs) enthalten häufig regulatorische Sequenzen, die die Genexpression posttranskriptiv beeinflussen. Solche 3′-UTRs enthalten oft sowohl Bindungsstellen für microRNAs (miRNAs) als auch für regulatorische Proteine. Durch Bindung an spezifische Stellen innerhalb der 3′-UTR können miRNAs die Genexpression verschiedener mRNAs verringern, indem sie entweder die Translation hemmen oder direkt den Abbau des Transkripts bewirken. Die 3′-UTR kann auch Silencer-Regionen aufweisen, die Repressorproteine binden, welche die Expression einer mRNA hemmen. ⓘ
Die 3′-UTR enthält häufig microRNA-Response-Elemente (MREs). MREs sind Sequenzen, an die miRNAs binden. Es handelt sich dabei um weit verbreitete Motive in 3′-UTRs. Unter allen regulatorischen Motiven innerhalb der 3′-UTRs (z. B. einschließlich Silencer-Regionen) machen MREs etwa die Hälfte der Motive aus. ⓘ
Im Jahr 2014 waren auf der miRBase-Website, einem Archiv für miRNA-Sequenzen und -Anmerkungen, 28.645 Einträge in 233 biologischen Spezies verzeichnet. Davon befanden sich 1 881 miRNAs in annotierten menschlichen miRNA-Loci. Es wurde vorhergesagt, dass miRNAs durchschnittlich etwa vierhundert Ziel-mRNAs haben (und die Expression von mehreren hundert Genen beeinflussen). Friedman et al. schätzen, dass >45.000 miRNA-Zielorte innerhalb menschlicher mRNA 3′UTRs über dem Hintergrundniveau konserviert sind und >60% der menschlichen proteinkodierenden Gene unter Selektionsdruck stehen, um die Kopplung an miRNAs aufrechtzuerhalten. ⓘ
Direkte Experimente zeigen, dass eine einzige miRNA die Stabilität von Hunderten einzigartiger mRNAs verringern kann. Andere Experimente zeigen, dass eine einzelne miRNA die Produktion von Hunderten von Proteinen unterdrücken kann, dass diese Unterdrückung aber oft relativ gering ist (weniger als 2-fach). ⓘ
Die Auswirkungen der miRNA-Dysregulation der Genexpression scheinen bei Krebs wichtig zu sein. So wurden beispielsweise bei Magen-Darm-Krebs neun miRNAs als epigenetisch verändert identifiziert, die die DNA-Reparaturenzyme herunterregulieren. ⓘ
Die Auswirkungen der miRNA-Dysregulation der Genexpression scheinen auch bei neuropsychiatrischen Erkrankungen wie Schizophrenie, bipolaren Störungen, schweren Depressionen, Parkinson-Krankheit, Alzheimer-Krankheit und Autismus-Spektrum-Störungen wichtig zu sein. ⓘ
Translationale Regulierung

Die direkte Regulierung der Translation ist weniger verbreitet als die Kontrolle der Transkription oder der mRNA-Stabilität, wird aber gelegentlich eingesetzt. Die Hemmung der Proteintranslation ist ein Hauptziel für Toxine und Antibiotika, so dass sie eine Zelle abtöten können, indem sie deren normale Kontrolle der Genexpression außer Kraft setzen. Zu den Proteinsyntheseinhibitoren gehören das Antibiotikum Neomycin und das Toxin Ricin. ⓘ
Posttranslationale Modifikationen
Posttranslationale Modifikationen (PTM) sind kovalente Modifikationen von Proteinen. Wie das RNA-Spleißen tragen sie zu einer erheblichen Diversifizierung des Proteoms bei. Diese Modifikationen werden in der Regel durch Enzyme katalysiert. Darüber hinaus können Prozesse wie kovalente Anfügungen an Aminosäure-Seitenkettenreste oft durch andere Enzyme rückgängig gemacht werden. Einige, wie die proteolytische Spaltung des Proteinrückgrats, sind jedoch irreversibel. ⓘ
PTMs spielen viele wichtige Rollen in der Zelle. So ist die Phosphorylierung vor allem an der Aktivierung und Deaktivierung von Proteinen und an Signalwegen beteiligt. PTMs sind an der Transkriptionsregulierung beteiligt: Eine wichtige Funktion der Acetylierung und Methylierung ist die Histonschwanzmodifikation, die den Zugang zur DNA für die Transkription verändert. Sie sind auch im Immunsystem zu beobachten, wo die Glykosylierung eine Schlüsselrolle spielt. Eine Art von PTM kann eine andere Art von PTM auslösen, wie man an der Ubiquitinierung sehen kann, die Proteine für den Abbau durch Proteolyse markiert. Die Proteolyse ist nicht nur am Abbau von Proteinen beteiligt, sondern auch an deren Aktivierung und Deaktivierung sowie an der Regulierung biologischer Prozesse wie der DNA-Transkription und des Zelltods. ⓘ
Messung
Die Messung der Genexpression ist ein wichtiger Bestandteil vieler Biowissenschaften, da die Fähigkeit, den Grad der Expression eines bestimmten Gens in einer Zelle, einem Gewebe oder einem Organismus zu quantifizieren, viele wertvolle Informationen liefern kann. Die Messung der Genexpression kann zum Beispiel:
- eine virale Infektion einer Zelle identifizieren (Expression viraler Proteine)
- die Anfälligkeit eines Individuums für Krebs feststellen (Expression von Onkogenen).
- feststellen, ob ein Bakterium gegen Penicillin resistent ist (Beta-Lactamase-Expression).
In ähnlicher Weise ist die Analyse des Ortes der Proteinexpression ein leistungsfähiges Instrument, und dies kann auf organismischer oder zellulärer Ebene erfolgen. Die Untersuchung der Lokalisierung ist besonders wichtig für die Untersuchung der Entwicklung in mehrzelligen Organismen und als Indikator für die Funktion von Proteinen in einzelnen Zellen. Idealerweise erfolgt die Messung der Expression durch den Nachweis des endgültigen Genprodukts (bei vielen Genen ist dies das Protein); oft ist es jedoch einfacher, eine der Vorstufen, in der Regel mRNA, nachzuweisen und aus diesen Messungen auf die Höhe der Genexpression zu schließen. ⓘ
mRNA-Quantifizierung
Der mRNA-Gehalt kann durch Northern Blotting quantitativ gemessen werden, das Informationen über Größe und Sequenz der mRNA-Moleküle liefert. Eine RNA-Probe wird auf einem Agarosegel aufgetrennt und mit einer radioaktiv markierten RNA-Sonde hybridisiert, die komplementär zur Zielsequenz ist. Die radioaktiv markierte RNA wird dann mit einem Autoradiographen nachgewiesen. Da die Verwendung radioaktiver Reagenzien das Verfahren zeitaufwändig und potenziell gefährlich macht, wurden alternative Markierungs- und Nachweismethoden wie Digoxigenin- und Biotin-Chemikalien entwickelt. Nachteilig am Northern Blotting ist, dass große RNA-Mengen benötigt werden und dass die Quantifizierung möglicherweise nicht ganz genau ist, da die Bandenstärke auf dem Bild eines Gels gemessen wird. Andererseits ermöglicht die zusätzliche mRNA-Größeninformation aus dem Northern Blot die Unterscheidung von alternativ gespleißten Transkripten. ⓘ
Ein weiterer Ansatz zur Messung der mRNA-Häufigkeit ist die RT-qPCR. Bei dieser Technik folgt auf die reverse Transkription eine quantitative PCR. Bei der reversen Transkription wird zunächst eine DNA-Vorlage aus der mRNA erzeugt; diese einzelsträngige Vorlage wird als cDNA bezeichnet. Die cDNA-Vorlage wird dann im quantitativen Schritt vervielfältigt, wobei sich die von markierten Hybridisierungssonden oder interkalierenden Farbstoffen emittierte Fluoreszenz mit dem Fortschreiten des DNA-Verstärkungsprozesses verändert. Mit einer sorgfältig erstellten Standardkurve kann die qPCR ein absolutes Maß für die Anzahl der Kopien der ursprünglichen mRNA liefern, typischerweise in Einheiten von Kopien pro Nanoliter homogenisierten Gewebes oder Kopien pro Zelle. Die qPCR ist sehr empfindlich (der Nachweis eines einzigen mRNA-Moleküls ist theoretisch möglich), kann aber je nach Art des verwendeten Reporters teuer sein; fluoreszenzmarkierte Oligonukleotid-Sonden sind teurer als unspezifische interkalierende Fluoreszenzfarbstoffe. ⓘ
Für die Erstellung von Expressionsprofilen oder die Hochdurchsatzanalyse vieler Gene in einer Probe kann bei Arrays mit geringer Dichte die quantitative PCR für Hunderte von Genen gleichzeitig durchgeführt werden. Ein zweiter Ansatz ist der Hybridisierungs-Mikroarray. Ein einziger Array oder "Chip" kann Sonden zur Bestimmung der Transkriptionsmengen für jedes bekannte Gen im Genom eines oder mehrerer Organismen enthalten. Alternativ können "Tag-basierte" Technologien wie die serielle Analyse der Genexpression (SAGE) und RNA-Seq verwendet werden, die ein relatives Maß für die zelluläre Konzentration verschiedener mRNAs liefern können. Ein Vorteil der Tag-basierten Methoden ist die "offene Architektur", die die genaue Messung jedes Transkripts mit bekannter oder unbekannter Sequenz ermöglicht. Die Sequenzierung der nächsten Generation (Next Generation Sequencing, NGS), wie z. B. RNA-Seq, ist ein weiterer Ansatz, der große Mengen an Sequenzdaten liefert, die mit einem Referenzgenom abgeglichen werden können. Obwohl NGS vergleichsweise zeitaufwändig, teuer und ressourcenintensiv ist, können damit Einzelnukleotid-Polymorphismen, Spleißvarianten und neuartige Gene identifiziert werden, und es können auch Profile der Expression in Organismen erstellt werden, für die nur wenige oder gar keine Sequenzinformationen verfügbar sind. ⓘ
RNA-Profile in Wikipedia
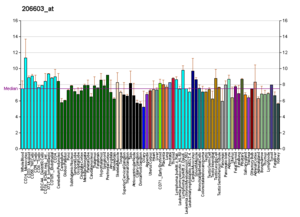
Profile wie diese gibt es für fast alle in Wikipedia aufgeführten Proteine. Sie werden von Organisationen wie dem Genomics Institute der Novartis Research Foundation und dem European Bioinformatics Institute erstellt. Zusätzliche Informationen können durch eine Suche in deren Datenbanken gefunden werden (für ein Beispiel des hier abgebildeten GLUT4-Transporters siehe Zitat). Diese Profile geben den Grad der DNA-Expression (und damit der RNA-Produktion) eines bestimmten Proteins in einem bestimmten Gewebe an und sind in den Bildern in der Protein-Box auf der rechten Seite jeder Wikipedia-Seite entsprechend farblich gekennzeichnet. ⓘ
Quantifizierung von Proteinen
Für Gene, die für Proteine kodieren, kann das Expressionsniveau direkt mit einer Reihe von Methoden bestimmt werden, die einige deutliche Analogien zu den Techniken für die Quantifizierung von mRNA aufweisen. ⓘ
Eine der am häufigsten verwendeten Methoden ist die Durchführung eines Western Blot gegen das betreffende Protein. Dies gibt nicht nur Aufschluss über die Identität, sondern auch über die Größe des Proteins. Eine Probe (oft ein Zelllysat) wird auf einem Polyacrylamidgel aufgetrennt, auf eine Membran übertragen und dann mit einem Antikörper gegen das gewünschte Protein gesampelt. Der Antikörper kann entweder mit einem Fluorophor oder mit Meerrettichperoxidase zur Bildgebung und/oder Quantifizierung konjugiert werden. Da dieser Test auf einem Gel basiert, ist die Quantifizierung weniger genau, hat aber den Vorteil, dass spätere Veränderungen des Proteins, z. B. Proteolyse oder Ubiquitinierung, anhand von Größenveränderungen erkannt werden können. ⓘ
mRNA-Protein-Korrelation
Während die Transkription die Genexpression direkt widerspiegelt, korreliert die Kopienzahl der mRNA-Moleküle nicht direkt mit der Zahl der aus der mRNA übersetzten Proteinmoleküle. Die Quantifizierung sowohl von Protein als auch von mRNA ermöglicht eine Korrelation der beiden Ebenen. Die Regulierung der einzelnen Schritte der Genexpression kann sich auf die Korrelation auswirken, wie bei der Regulierung der Translation oder der Proteinstabilität gezeigt wurde. Auch posttranslationale Faktoren, wie der Proteintransport in hochpolaren Zellen, können die gemessene mRNA-Protein-Korrelation beeinflussen. ⓘ
Lokalisierung
Die Analyse der Expression beschränkt sich nicht auf die Quantifizierung, sondern es kann auch die Lokalisierung bestimmt werden. Die mRNA kann mit einem entsprechend markierten komplementären mRNA-Strang nachgewiesen werden, und das Protein kann über markierte Antikörper nachgewiesen werden. Die sondierte Probe wird dann mikroskopisch beobachtet, um festzustellen, wo sich die mRNA oder das Protein befindet. ⓘ
Durch Ersetzen des Gens durch eine neue Version, die mit einem grün fluoreszierenden Protein (oder einem ähnlichen Marker) fusioniert ist, kann die Expression direkt in lebenden Zellen quantifiziert werden. Dies geschieht durch Bildgebung mit einem Fluoreszenzmikroskop. Es ist sehr schwierig, ein mit GFP fusioniertes Protein an seine ursprüngliche Stelle im Genom zu klonieren, ohne das Expressionsniveau zu beeinflussen, so dass diese Methode oft nicht zur Messung der endogenen Genexpression verwendet werden kann. Sie wird jedoch häufig verwendet, um die Expression eines Gens zu messen, das künstlich in die Zelle eingeführt wurde, z. B. über einen Expressionsvektor. Es ist wichtig zu wissen, dass durch die Kopplung eines Zielproteins an einen fluoreszierenden Reporter das Verhalten des Proteins, einschließlich seiner zellulären Lokalisierung und seines Expressionsniveaus, erheblich verändert werden kann. ⓘ
Der Enzymimmunoassay funktioniert mit Hilfe von Antikörpern, die auf einer Mikrotiterplatte immobilisiert sind, um die interessierenden Proteine aus den in die Vertiefung gegebenen Proben zu erfassen. Mit Hilfe eines Nachweisantikörpers, der mit einem Enzym oder Fluorophor konjugiert ist, kann die Menge des gebundenen Proteins durch fluorometrischen oder kolorimetrischen Nachweis genau gemessen werden. Der Nachweisprozess ist dem eines Western Blot sehr ähnlich, aber durch den Verzicht auf die Gelschritte kann eine genauere Quantifizierung erreicht werden. ⓘ
Expressionssystem

Ein Expressionssystem ist ein System, das speziell für die Produktion eines bestimmten Genprodukts entwickelt wurde. In der Regel handelt es sich dabei um ein Protein, es kann sich aber auch um RNA handeln, z. B. tRNA oder ein Ribozym. Ein Expressionssystem besteht aus einem Gen, das in der Regel durch DNA kodiert wird, und der molekularen Maschinerie, die erforderlich ist, um die DNA in mRNA zu transkribieren und die mRNA mit Hilfe der bereitgestellten Reagenzien in Protein zu übersetzen. Im weitesten Sinne schließt dies jede lebende Zelle ein, doch wird der Begriff normalerweise eher für die Expression als Laborwerkzeug verwendet. Ein Expressionssystem ist daher oft in irgendeiner Weise künstlich. Expressionssysteme sind jedoch ein grundsätzlich natürlicher Prozess. Viren sind ein hervorragendes Beispiel dafür, dass sie sich vermehren, indem sie die Wirtszelle als Expressionssystem für die viralen Proteine und das Genom nutzen. ⓘ
Induzierbare Expression
Doxycyclin wird auch bei der "Tet-on"- und "Tet-off"-Tetracyclin-gesteuerten Transkriptionsaktivierung verwendet, um die Expression von Transgenen in Organismen und Zellkulturen zu steuern. ⓘ
In der Natur
Zusätzlich zu diesen biologischen Werkzeugen werden bestimmte, in der Natur beobachtete Konfigurationen der DNA (Gene, Promotoren, Enhancer, Repressoren) und die dazugehörige Maschinerie selbst als Expressionssystem bezeichnet. Dieser Begriff wird in der Regel verwendet, wenn ein Gen oder eine Gruppe von Genen unter genau definierten Bedingungen eingeschaltet wird, z. B. das einfache Repressor-Switch-Expressionssystem in Lambda-Phagen und das lac-Operatorsystem in Bakterien. Mehrere natürliche Expressionssysteme werden direkt verwendet oder modifiziert und für künstliche Expressionssysteme wie das Tet-on und Tet-off Expressionssystem verwendet. ⓘ
Gennetzwerke
Gene werden manchmal als Knoten in einem Netzwerk betrachtet, wobei die Eingänge Proteine wie Transkriptionsfaktoren und die Ausgänge das Niveau der Genexpression sind. Der Knoten selbst erfüllt eine Funktion, und das Funktionieren dieser Funktionen wurde als eine Art Informationsverarbeitung innerhalb der Zellen interpretiert und bestimmt das zelluläre Verhalten. ⓘ
Gennetzwerke können auch ohne die Formulierung eines expliziten Kausalmodells konstruiert werden. Dies ist häufig der Fall, wenn Netzwerke aus großen Expressionsdatensätzen zusammengesetzt werden. Die Kovariation und Korrelation der Expression wird über eine große Stichprobe von Fällen und Messungen (oft Transkriptom- oder Proteomdaten) berechnet. Die Quelle der Variation kann entweder experimentell oder natürlich (Beobachtungsdaten) sein. Es gibt verschiedene Möglichkeiten, Genexpressionsnetzwerke zu konstruieren, aber ein gängiger Ansatz besteht darin, eine Matrix aller paarweisen Korrelationen der Expression über Bedingungen, Zeitpunkte oder Individuen hinweg zu berechnen und die Matrix (nach einem Schwellenwert) in eine grafische Darstellung zu konvertieren, in der Knoten Gene, Transkripte oder Proteine darstellen und Kanten, die diese Knoten verbinden, die Stärke der Assoziation repräsentieren (siehe GeneNetwork GeneNetwork 2). ⓘ
Techniken und Werkzeuge
Die folgenden experimentellen Techniken werden zur Messung der Genexpression verwendet und sind in grober chronologischer Reihenfolge aufgeführt, beginnend mit den älteren, etablierteren Technologien. Sie werden nach dem Grad der Multiplexität in zwei Gruppen eingeteilt.
- Low- bis Mid-Plex-Techniken:
- Reportergen
- Northern Blot
- Western Blot
- Fluoreszierende in situ Hybridisierung
- Reverse Transkription PCR
- Higher-plex Techniken:
- SAGE
- DNA-Mikroarray
- Tiling-Array
- RNA-Seq ⓘ
Datenbanken zur Genexpression
- Genexpression Omnibus (GEO) beim NCBI
- Expressionsatlas beim EBI
- Maus-Genexpressionsdatenbank im Jackson Laboratory
- CollecTF: eine Datenbank von experimentell validierten Transkriptionsfaktor-Bindungsstellen in Bakterien.
- COLOMBOS: Sammlung von Kompendien zur bakteriellen Expression.
- Many Microbe Microarrays Database: mikrobielle Affymetrix-Daten ⓘ



