Proteinbiosynthese

Die Proteinbiosynthese (oder Proteinsynthese) ist ein zentraler biologischer Prozess, der innerhalb der Zellen stattfindet und den Verlust von Zellproteinen (durch Abbau oder Export) durch die Produktion neuer Proteine ausgleicht. Proteine erfüllen eine Reihe wichtiger Funktionen als Enzyme, Strukturproteine oder Hormone. Die Proteinsynthese ist sowohl bei Prokaryonten als auch bei Eukaryonten ein sehr ähnlicher Prozess, es gibt jedoch einige deutliche Unterschiede. ⓘ
Die Proteinsynthese lässt sich grob in zwei Phasen unterteilen - Transkription und Translation. Bei der Transkription wird ein DNA-Abschnitt, der für ein Protein kodiert, ein so genanntes Gen, in ein Vorlagenmolekül, die Boten-RNA (mRNA), umgewandelt. Diese Umwandlung wird von Enzymen, den so genannten RNA-Polymerasen, im Zellkern durchgeführt. In Eukaryoten wird diese mRNA zunächst in einer vorzeitigen Form (pre-mRNA) produziert, die nach der Transkription verändert wird, um die reife mRNA zu erzeugen. Die reife mRNA wird aus dem Zellkern über Kernporen in das Zytoplasma der Zelle exportiert, wo die Translation stattfindet. Während der Translation wird die mRNA von Ribosomen gelesen, die anhand der Nukleotidsequenz der mRNA die Sequenz der Aminosäuren bestimmen. Die Ribosomen katalysieren die Bildung von kovalenten Peptidbindungen zwischen den kodierten Aminosäuren, um eine Polypeptidkette zu bilden. ⓘ
Nach der Übersetzung muss sich die Polypeptidkette falten, um ein funktionsfähiges Protein zu bilden; um z. B. als Enzym zu funktionieren, muss sich die Polypeptidkette richtig falten, um eine funktionelle aktive Stelle zu bilden. Um eine funktionelle dreidimensionale (3D) Form anzunehmen, muss die Polypeptidkette zunächst eine Reihe kleinerer darunter liegender Strukturen bilden, die als Sekundärstrukturen bezeichnet werden. Die Polypeptidkette in diesen Sekundärstrukturen faltet sich dann, um die gesamte 3D-Tertiärstruktur zu erzeugen. Sobald das Protein korrekt gefaltet ist, kann es durch verschiedene posttranslationale Modifikationen weiter gereift werden. Posttranslationale Modifikationen können die Funktionsfähigkeit des Proteins, seinen Standort in der Zelle (z. B. Zytoplasma oder Zellkern) und seine Fähigkeit zur Interaktion mit anderen Proteinen verändern. ⓘ
Der Proteinbiosynthese kommt bei Krankheiten eine Schlüsselrolle zu, da Veränderungen und Fehler in diesem Prozess durch zugrundeliegende DNA-Mutationen oder Proteinfehlfaltungen oft die Ursache für eine Krankheit sind. DNA-Mutationen verändern die nachfolgende mRNA-Sequenz, die dann die mRNA-kodierte Aminosäuresequenz verändert. Mutationen können dazu führen, dass die Polypeptidkette kürzer ist, indem sie eine Stoppsequenz erzeugen, die einen frühen Abbruch der Translation bewirkt. Oder eine Mutation in der mRNA-Sequenz verändert die spezifische Aminosäure, die an dieser Position in der Polypeptidkette kodiert wird. Diese Aminosäureveränderung kann sich auf die Fähigkeit des Proteins auswirken, zu funktionieren oder sich korrekt zu falten. Falsch gefaltete Proteine sind häufig an Krankheiten beteiligt, da falsch gefaltete Proteine dazu neigen, zusammenzukleben und dichte Proteinklumpen zu bilden. Diese Verklumpungen werden mit einer Reihe von Krankheiten in Verbindung gebracht, die häufig neurologischer Natur sind, wie z. B. die Alzheimer-Krankheit und die Parkinson-Krankheit. ⓘ

Proteinbiosynthese (PBS) ist die Neubildung von Proteinen in Zellen. Bei diesem für alle Lebewesen zentralen Prozess wird nach Vorgabe genetischer Information ein Protein aus Aminosäuren aufgebaut. ⓘ
Einige Arten von Bakterien, Archaeen und Pilzen können über ribosomale Proteinsynthese besondere Proteine aufbauen, die als Multienzymkomplexe eine nichtribosomale Peptidsynthese ermöglichen (NRPS). ⓘ
Transkription
Die Transkription findet im Zellkern statt, wobei die DNA als Vorlage zur Herstellung von mRNA dient. Bei Eukaryoten wird dieses mRNA-Molekül als prä-mRNA bezeichnet, da es im Zellkern posttranskriptionelle Veränderungen erfährt, um ein reifes mRNA-Molekül zu erzeugen. Bei Prokaryonten sind posttranskriptionelle Modifikationen jedoch nicht erforderlich, so dass das reife mRNA-Molekül sofort durch Transkription erzeugt wird. ⓘ
Zunächst wirkt ein als Helikase bezeichnetes Enzym auf das DNA-Molekül ein. Die DNA hat eine antiparallele Doppelhelixstruktur, die aus zwei komplementären Polynukleotidsträngen besteht, die durch Wasserstoffbrücken zwischen den Basenpaaren zusammengehalten werden. Die Helicase unterbricht die Wasserstoffbrücken, so dass sich ein Bereich der DNA - der einem Gen entspricht - abwickelt, die beiden DNA-Stränge trennt und eine Reihe von Basen freilegt. Obwohl die DNA ein doppelsträngiges Molekül ist, dient nur einer der Stränge als Vorlage für die Synthese der prä-mRNA - dieser Strang wird als Vorlagenstrang bezeichnet. Der andere DNA-Strang (der zum Template-Strang komplementär ist) wird als kodierender Strang bezeichnet. ⓘ
Sowohl die DNA als auch die RNA haben eine intrinsische Direktionalität, d. h. es gibt zwei unterschiedliche Enden des Moleküls. Diese Eigenschaft der Richtungsabhängigkeit ist auf die asymmetrischen Nukleotiduntereinheiten zurückzuführen, bei denen sich auf einer Seite des Pentosezuckers eine Phosphatgruppe und auf der anderen Seite eine Base befindet. Die fünf Kohlenstoffe im Pentosezucker sind von 1' (wobei ' für Primzahl steht) bis 5' nummeriert. Die Phosphodiesterbindungen, die die Nukleotide verbinden, werden also durch die Verbindung der Hydroxylgruppe am 3'-Kohlenstoff eines Nukleotids mit der Phosphatgruppe am 5'-Kohlenstoff eines anderen Nukleotids gebildet. Daher verläuft der kodierende DNA-Strang in einer Richtung von 5' nach 3' und der komplementäre Template-DNA-Strang in der entgegengesetzten Richtung von 3' nach 5'. ⓘ
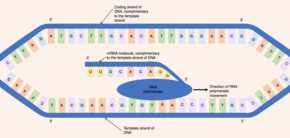
Das Enzym RNA-Polymerase bindet an den freiliegenden Vorlagenstrang und liest das Gen in der Richtung von 3' nach 5' ab. Gleichzeitig synthetisiert die RNA-Polymerase einen Einzelstrang der prä-mRNA in 5'-zu-3'-Richtung, indem sie die Bildung von Phosphodiesterbindungen zwischen aktivierten (im Kern freien) Nukleotiden katalysiert, die zu einer komplementären Basenpaarung mit dem Vorlagenstrang fähig sind. Hinter der sich bewegenden RNA-Polymerase fügen sich die beiden DNA-Stränge wieder zusammen, so dass jeweils nur 12 Basenpaare der DNA freigelegt sind. Die RNA-Polymerase baut das prä-mRNA-Molekül mit einer Geschwindigkeit von 20 Nukleotiden pro Sekunde auf, so dass in einer Stunde Tausende von prä-mRNA-Molekülen desselben Gens entstehen können. Trotz der hohen Syntheserate verfügt das Enzym RNA-Polymerase über einen eigenen Mechanismus zur Korrekturlesung. Der Korrekturlesemechanismus ermöglicht es der RNA-Polymerase, falsche Nukleotide (die nicht komplementär zum DNA-Vorlagestrang sind) durch eine Exzisionsreaktion aus dem wachsenden prä-mRNA-Molekül zu entfernen. Wenn die RNA-Polymerase eine bestimmte DNA-Sequenz erreicht, die die Transkription beendet, löst sich die RNA-Polymerase ab und die prä-mRNA-Synthese ist abgeschlossen. ⓘ
Das synthetisierte pre-mRNA-Molekül ist komplementär zum Template-DNA-Strang und hat die gleiche Nukleotidsequenz wie der codierende DNA-Strang. Es gibt jedoch einen entscheidenden Unterschied in der Nukleotidzusammensetzung von DNA- und mRNA-Molekülen. Die DNA besteht aus den Basen Guanin, Cytosin, Adenin und Thymin (G, C, A und T) - die RNA besteht ebenfalls aus vier Basen: Guanin, Cytosin, Adenin und Uracil. In RNA-Molekülen wird die DNA-Base Thymin durch Uracil ersetzt, das eine Basenpaarung mit Adenin eingehen kann. Daher werden im prä-mRNA-Molekül alle komplementären Basen, die im kodierenden DNA-Strang Thymin wären, durch Uracil ersetzt. ⓘ

Posttranskriptionelle Modifikationen

Nach Abschluss der Transkription wird das prä-mRNA-Molekül posttranskriptional modifiziert, um ein reifes mRNA-Molekül zu erzeugen. ⓘ
Innerhalb der posttranskriptionellen Modifikationen gibt es 3 Schlüsselschritte:
- Anfügen einer 5'-Kappe an das 5'-Ende des prä-mRNA-Moleküls
- Anhängen eines 3'-Poly(A)-Schwanzes an das 3'-Ende des prä-mRNA-Moleküls
- Entfernung von Introns durch RNA-Spleißen
Die 5'-Kappe wird an das 5'-Ende des prä-mRNA-Moleküls angefügt und besteht aus einem durch Methylierung modifizierten Guanin-Nukleotid. Der Zweck der 5'-Kappe ist es, den Abbau reifer mRNA-Moleküle vor der Translation zu verhindern. Die Kappe unterstützt auch die Bindung des Ribosoms an die mRNA, um die Translation zu starten, und ermöglicht die Unterscheidung der mRNA von anderen RNAs in der Zelle. Im Gegensatz dazu wird der 3'-Poly(A)-Schwanz an das 3'-Ende des mRNA-Moleküls angefügt und besteht aus 100-200 Adeninbasen. Diese unterschiedlichen mRNA-Modifikationen ermöglichen es der Zelle zu erkennen, dass die vollständige mRNA-Botschaft intakt ist, wenn sowohl die 5'-Kappe als auch der 3'-Schwanz vorhanden sind. ⓘ
Dieses modifizierte prä-mRNA-Molekül durchläuft dann den Prozess des RNA-Spleißens. Gene bestehen aus einer Reihe von Introns und Exons. Introns sind Nukleotidsequenzen, die nicht für ein Protein kodieren, während Exons Nukleotidsequenzen sind, die direkt für ein Protein kodieren. Introns und Exons sind sowohl in der zugrundeliegenden DNA-Sequenz als auch im Prä-mRNA-Molekül vorhanden. Um ein reifes mRNA-Molekül zu erzeugen, das für ein Protein kodiert, muss daher ein Spleißvorgang stattfinden. Während des Spleißens werden die dazwischenliegenden Introns durch einen Multiproteinkomplex, das so genannte Spleißosom (bestehend aus über 150 Proteinen und RNA), aus dem prä-mRNA-Molekül entfernt. Das reife mRNA-Molekül wird dann durch Kernporen in der Hülle des Zellkerns ins Zytoplasma exportiert. ⓘ
- Capping
- Währenddessen findet außerdem das sogenannte Capping statt, bei dem die Stabilität der RNA erhöht wird. Dabei wird eine sogenannte 5'-Cap-Struktur angehängt, wobei das 5' Ende der sich in Synthese befindlichen prä-mRNA zu einer Struktur umgewandelt wird, die als „Cap“ bezeichnet wird und die mRNA vor der Verdauung durch 5'-Exonucleasen und Phosphatasen schützt. ⓘ
- RNA-Edition
- Bei der RNA-Edition werden einzelne oder mehrere Nukleinbasen des RNA-Moleküls nach der Transkription verändert (modifiziert), eingefügt (insertiert) oder ausgeschnitten (deletiert). Beispielsweise kann das Editing so auf der mRNA ein neues Stopcodon ergeben, das stromaufwärts des vormaligen liegt; die Translation bricht dann hier ab und es wird die kürzere Isoform eines Proteins gebildet. RNA-Editieren kommt nur bei einigen Organismen, Zellen oder Zellorganellen vor und ist oft auf besondere Nukleotidsequenzen beschränkt. ⓘ
Übersetzung


Während der Translation synthetisieren die Ribosomen Polypeptidketten aus mRNA-Vorlagenmolekülen. Bei Eukaryonten findet die Translation im Zytoplasma der Zelle statt, wo sich die Ribosomen entweder frei schwebend oder an das endoplasmatische Retikulum gebunden befinden. Bei Prokaryonten, die keinen Zellkern haben, finden sowohl die Transkription als auch die Translation im Zytoplasma statt. ⓘ
Ribosomen sind komplexe molekulare Maschinen, die aus einer Mischung von Proteinen und ribosomaler RNA bestehen und in zwei Untereinheiten (eine große und eine kleine Untereinheit) unterteilt sind, die das mRNA-Molekül umgeben. Das Ribosom liest das mRNA-Molekül in 5'-3'-Richtung und verwendet es als Vorlage, um die Reihenfolge der Aminosäuren in der Polypeptidkette zu bestimmen. Um das mRNA-Molekül zu übersetzen, verwendet das Ribosom kleine Moleküle, die so genannten Transfer-RNAs (tRNAs), um die richtigen Aminosäuren an das Ribosom zu liefern. Jede tRNA besteht aus 70-80 Nukleotiden und nimmt eine charakteristische Kleeblattstruktur an, die durch die Bildung von Wasserstoffbrücken zwischen den Nukleotiden innerhalb des Moleküls entsteht. Es gibt etwa 60 verschiedene Arten von tRNAs. Jede tRNA bindet an eine bestimmte Sequenz von drei Nukleotiden (ein so genanntes Codon) innerhalb des mRNA-Moleküls und liefert eine bestimmte Aminosäure. ⓘ
Das Ribosom heftet sich zunächst am Startcodon (AUG) an die mRNA und beginnt mit der Übersetzung des Moleküls. Die mRNA-Nukleotidsequenz wird in Tripletts gelesen - drei benachbarte Nukleotide im mRNA-Molekül entsprechen einem einzigen Codon. Jede tRNA hat eine exponierte Sequenz von drei Nukleotiden, das so genannte Anticodon, die in ihrer Sequenz komplementär zu einem bestimmten Codon sind, das in der mRNA vorhanden sein kann. Das erste Codon, auf das man trifft, ist zum Beispiel das Startcodon, das aus den Nukleotiden AUG besteht. Die richtige tRNA mit dem Anticodon (komplementäre 3-Nukleotid-Sequenz UAC) bindet sich mit Hilfe des Ribosoms an die mRNA. Diese tRNA liefert die richtige Aminosäure, die dem mRNA-Codon entspricht, im Fall des Startcodons ist dies die Aminosäure Methionin. Das nächste Codon (neben dem Startcodon) wird dann von der richtigen tRNA mit komplementärem Anticodon gebunden, wodurch die nächste Aminosäure an das Ribosom geliefert wird. Das Ribosom nutzt dann seine enzymatische Aktivität der Peptidyltransferase, um die Bildung der kovalenten Peptidbindung zwischen den beiden benachbarten Aminosäuren zu katalysieren. ⓘ
Das Ribosom bewegt sich dann entlang des mRNA-Moleküls zum dritten Codon. Das Ribosom setzt dann das erste tRNA-Molekül frei, da nur zwei tRNA-Moleküle gleichzeitig von einem einzigen Ribosom zusammengebracht werden können. Die nächste komplementäre tRNA mit dem richtigen Anticodon, das zum dritten Codon komplementär ist, wird ausgewählt und liefert die nächste Aminosäure an das Ribosom, die kovalent an die wachsende Polypeptidkette angehängt wird. Dieser Prozess setzt sich fort, wobei sich das Ribosom entlang des mRNA-Moleküls bewegt und bis zu 15 Aminosäuren pro Sekunde an die Polypeptidkette anfügt. Hinter dem ersten Ribosom können bis zu 50 weitere Ribosomen an das mRNA-Molekül binden und ein Polysom bilden, das die gleichzeitige Synthese mehrerer identischer Polypeptidketten ermöglicht. Die wachsende Polypeptidkette wird abgebrochen, wenn das Ribosom auf ein Stoppcodon (UAA, UAG oder UGA) im mRNA-Molekül trifft. In diesem Fall kann keine tRNA das Stoppcodon erkennen, und ein Freisetzungsfaktor veranlasst die Freisetzung der vollständigen Polypeptidkette aus dem Ribosom. Dr. Har Gobind Khorana, ein aus Indien stammender Wissenschaftler, entschlüsselte die RNA-Sequenzen für etwa 20 Aminosäuren. Für seine Arbeit erhielt er 1968 zusammen mit zwei anderen Wissenschaftlern den Nobelpreis. ⓘ

Unter Translation versteht man die Übersetzung der Basensequenz der mRNA in die Aminosäuresequenz des Proteins, die an den Ribosomen geschieht. In der mRNA bilden jeweils drei aufeinander folgende Basen, ein Basentriplett, innerhalb des offenen Leserahmens ein Codon, welches für eine Aminosäure codiert (siehe hierzu genetischer Code). Am Ribosom werden die Codons entsprechend ihrer Abfolge in Aminosäuren translatiert und diese sequentiell zu einem Polypeptid verknüpft. ⓘ
Zur Ausbildung einer Peptidbindung zwischen zwei Aminosäuren müssen sie in räumliche Nähe zueinander gebracht werden. Dazu wird die Oberfläche einer großen supramolekularen Struktur benötigt. Diese Aufgabe erfüllen die Ribosomen, zusammengesetzt aus einer kleinen und einer großen Untereinheit, welche zwei nebeneinanderliegende Bindungsstellen formt: die A-Stelle und die P-Stelle. ⓘ
- Termination
- Trifft ein sich an der mRNA entlang bewegendes Ribosom auf eines der drei Stoppcodons, kommt es zunächst zum Stillstand der Translation, da keine passenden tRNA-Moleküle vorhanden sind, welche für eine Aminosäure codiert sind (Suppression). An ihre Stelle treten so genannte Terminations- oder Release-Faktoren (RFs), die an die A-Stelle binden und die Substratspezifität der Peptidyl-Transferase dahingehend verändern, dass ein Wassermolekül anstelle einer AA-tRNA aktiviert wird. Durch dessen nucleophilen Angriff auf die Bindung zwischen Peptidkette und tRNA kommt es schließlich zur Freisetzung des synthetisierten Proteins und zur Trennung der mRNA vom Ribosom. ⓘ
Proteinfaltung

Sobald die Synthese der Polypeptidkette abgeschlossen ist, faltet sich die Polypeptidkette, um eine spezifische Struktur anzunehmen, die es dem Protein ermöglicht, seine Funktionen auszuführen. Die Grundform der Proteinstruktur wird als Primärstruktur bezeichnet, bei der es sich einfach um die Polypeptidkette handelt, d. h. um eine Abfolge kovalent gebundener Aminosäuren. Die Primärstruktur eines Proteins wird durch ein Gen kodiert. Daher kann jede Änderung der Gensequenz die Primärstruktur des Proteins und alle nachfolgenden Ebenen der Proteinstruktur verändern, wodurch sich letztlich die Gesamtstruktur und -funktion ändert. ⓘ
Die Primärstruktur eines Proteins (die Polypeptidkette) kann sich dann falten oder winden, um die Sekundärstruktur des Proteins zu bilden. Die gebräuchlichsten Arten von Sekundärstrukturen sind als Alpha-Helix oder Beta-Faltblatt bekannt; dabei handelt es sich um kleine Strukturen, die durch die Bildung von Wasserstoffbrücken innerhalb der Polypeptidkette entstehen. Diese Sekundärstruktur faltet sich dann und bildet die Tertiärstruktur des Proteins. Die Tertiärstruktur ist die gesamte 3D-Struktur des Proteins, die aus verschiedenen zusammengefalteten Sekundärstrukturen besteht. In der Tertiärstruktur werden wichtige Proteinmerkmale, z. B. das aktive Zentrum, gefaltet und gebildet, damit das Protein funktionieren kann. Schließlich können einige Proteine eine komplexe quaternäre Struktur aufweisen. Die meisten Proteine bestehen aus einer einzigen Polypeptidkette, einige Proteine setzen sich jedoch aus mehreren Polypeptidketten (den so genannten Untereinheiten) zusammen, die sich falten und zusammenwirken, um die quaternäre Struktur zu bilden. Das Gesamtprotein ist also ein Komplex mit mehreren Untereinheiten, der aus mehreren gefalteten Untereinheiten mit Polypeptidketten besteht, z. B. Hämoglobin. ⓘ
Posttranslationsereignisse
Im Anschluss an die Proteinbiosynthese gibt es Ereignisse wie die Proteolyse und die Proteinfaltung. Unter Proteolyse versteht man die Spaltung von Proteinen durch Proteasen und die Zerlegung von Proteinen in Aminosäuren durch die Wirkung von Enzymen. ⓘ
Posttranslationale Modifikationen
Die Polypeptidketten einiger Proteine werden schon während der Translation (cotranslational) durch spezielle Enzyme verändert, in den meisten Fällen aber werden Proteine erst nach Abschluss der Translation (posttranslational) modifiziert. Während Chaperone den formgebenden Prozess der Proteinfaltung beeinflussen, von dem auch die Assoziation zu Proteinkomplexen abhängt, verfügt eine Zelle daneben über eine Vielzahl an Möglichkeiten, die Struktur eines Proteins spezifisch abzuwandeln, derart auch funktionell andere Proteinspezies zu schaffen und so durch Modifikationen das Proteom zu erweitern. ⓘ
Zu diesen Modifikationen gehören die Abspaltung von einzelnen endständigen Aminosäuren oder auch die längerer Peptidsequenzen bei Präkursor-Proteinen, die Einführung zusätzlicher Bindungen, z. B. Disulfidbrücken zwischen Cysteinresten, oder funktioneller Gruppen, wie Hydroxylierungen von Aminosäuren (Prolin zu 4-Hydroxyprolin durch die Prolyl-4-Hydroxylase, Lysin zu Hydroxylysin durch die Lysylhydroxylase), sowie Oxidationen (z. B. kovalente Quervernetzungen mittels Lysinresten durch die Lysyloxidase), Carboxylierungen oder Decarboxylierungen und zahlreiche weitere. Beispielsweise entstehen durch Glykosylierungen Glykoproteine, durch Acylierungen und Prenylierungen Lipoproteine. ⓘ
Die einzelnen Schritte von Modifizierungen werden jeweils durch besondere Enzyme katalysiert, deren Vorkommen oft auf bestimmte Organellen, Zellen oder Gewebe beschränkt ist. Außerdem kann die Abfolge modifizierender Schritte bzw. deren zeitlicher Verlauf variiert werden, abhängig von Zellmilieu, Entwicklungsphase oder Umgebungsbedingungen. Das Kollagenmolekül etwa durchläuft eine Reihe posttranslationaler Modifikationen, von denen einige erst im Extrazellularraum stattfinden. ⓘ
Es gibt vier Hauptklassen von posttranslationalen Modifikationen:
- Spaltung
- Hinzufügung von chemischen Gruppen
- Hinzufügen komplexer Moleküle
- Bildung von intramolekularen Bindungen ⓘ
Spaltung
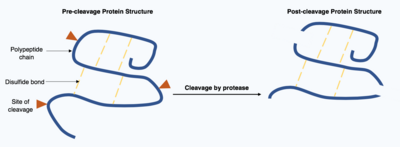
Die Spaltung von Proteinen ist eine irreversible posttranslationale Modifikation, die von Enzymen, den Proteasen, durchgeführt wird. Diese Proteasen sind oft sehr spezifisch und bewirken die Hydrolyse einer begrenzten Anzahl von Peptidbindungen innerhalb des Zielproteins. Das daraus resultierende verkürzte Protein hat eine veränderte Polypeptidkette mit unterschiedlichen Aminosäuren am Anfang und Ende der Kette. Diese posttranslationale Modifikation verändert häufig die Funktion des Proteins. Das Protein kann durch die Spaltung inaktiviert oder aktiviert werden und neue biologische Aktivitäten entfalten. ⓘ
Hinzufügung von chemischen Gruppen

Nach der Translation können kleine chemische Gruppen an Aminosäuren innerhalb der reifen Proteinstruktur angefügt werden. Beispiele für Prozesse, bei denen chemische Gruppen an das Zielprotein angefügt werden, sind Methylierung, Acetylierung und Phosphorylierung. ⓘ
Methylierung ist die reversible Anlagerung einer Methylgruppe an eine Aminosäure, die durch Methyltransferase-Enzyme katalysiert wird. Methylierung kommt bei mindestens 9 der 20 häufigsten Aminosäuren vor, hauptsächlich jedoch bei den Aminosäuren Lysin und Arginin. Ein Beispiel für ein Protein, das häufig methyliert wird, ist ein Histon. Histone sind Proteine, die im Kern der Zelle zu finden sind. Die DNA ist eng um die Histone gewickelt und wird durch andere Proteine und durch Wechselwirkungen zwischen negativen Ladungen in der DNA und positiven Ladungen auf dem Histon an ihrem Platz gehalten. Ein hochspezifisches Muster der Aminosäure-Methylierung auf den Histonproteinen wird verwendet, um zu bestimmen, welche Bereiche der DNA fest umwickelt sind und nicht transkribiert werden können und welche Bereiche locker umwickelt sind und transkribiert werden können. ⓘ
Die Histon-basierte Regulierung der DNA-Transkription wird auch durch Acetylierung verändert. Acetylierung ist die reversible kovalente Anlagerung einer Acetylgruppe an eine Lysinaminosäure durch das Enzym Acetyltransferase. Die Acetylgruppe wird von einem Spendermolekül, dem Acetylcoenzym A, entfernt und auf das Zielprotein übertragen. Histone werden an ihren Lysinresten durch Enzyme, die als Histon-Acetyltransferase bezeichnet werden, acetyliert. Durch die Acetylierung werden die Ladungswechselwirkungen zwischen dem Histon und der DNA geschwächt, wodurch mehr Gene in der DNA für die Transkription zugänglich werden. ⓘ
Die letzte, weit verbreitete posttranslationale chemische Gruppenmodifikation ist die Phosphorylierung. Die Phosphorylierung ist die reversible, kovalente Anlagerung einer Phosphatgruppe an bestimmte Aminosäuren (Serin, Threonin und Tyrosin) innerhalb des Proteins. Die Phosphatgruppe wird von einer Proteinkinase vom Spendermolekül ATP entfernt und auf die Hydroxylgruppe der Zielaminosäure übertragen, wobei Adenosindiphosphat als Nebenprodukt entsteht. Dieser Prozess kann umgekehrt und die Phosphatgruppe durch das Enzym Proteinphosphatase entfernt werden. Durch die Phosphorylierung kann auf dem phosphorylierten Protein eine Bindungsstelle entstehen, die es ihm ermöglicht, mit anderen Proteinen zu interagieren und große Multiproteinkomplexe zu bilden. Alternativ kann die Phosphorylierung das Niveau der Proteinaktivität verändern, indem sie die Fähigkeit des Proteins, sein Substrat zu binden, verändert. ⓘ
Hinzufügen komplexer Moleküle
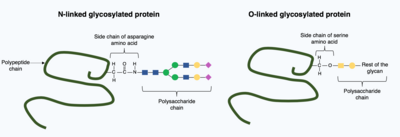
Posttranslationale Modifikationen können komplexere, große Moleküle in die gefaltete Proteinstruktur einbauen. Ein gängiges Beispiel hierfür ist die Glykosylierung, das Hinzufügen eines Polysaccharidmoleküls, das weithin als die häufigste posttranslationale Modifikation gilt. ⓘ
Bei der Glykosylierung wird ein Polysaccharidmolekül (ein so genanntes Glykan) durch Enzyme der Glykosyltransferasen kovalent an das Zielprotein angefügt und durch Glykosidasen im endoplasmatischen Retikulum und im Golgi-Apparat modifiziert. Die Glykosylierung kann eine entscheidende Rolle bei der Bestimmung der endgültigen, gefalteten 3D-Struktur des Zielproteins spielen. In einigen Fällen ist die Glykosylierung für die korrekte Faltung erforderlich. Die N-gebundene Glykosylierung fördert die Proteinfaltung, indem sie die Löslichkeit erhöht und die Bindung des Proteins an Proteinchaperone vermittelt. Chaperone sind Proteine, die für die Faltung und Aufrechterhaltung der Struktur anderer Proteine verantwortlich sind. ⓘ
Es gibt im Wesentlichen zwei Arten der Glykosylierung, die N-gebundene Glykosylierung und die O-gebundene Glykosylierung. Die N-gebundene Glykosylierung beginnt im endoplasmatischen Retikulum mit der Anlagerung eines Vorläuferglykans. Das Vorläuferglykan wird im Golgi-Apparat modifiziert, so dass ein komplexes Glykan entsteht, das kovalent an den Stickstoff einer Asparagin-Aminosäure gebunden ist. Im Gegensatz dazu ist die O-gebundene Glykosylierung die sequentielle kovalente Anlagerung einzelner Zucker an den Sauerstoff der Aminosäuren Serin und Threonin innerhalb der reifen Proteinstruktur. ⓘ
Bildung von kovalenten Bindungen
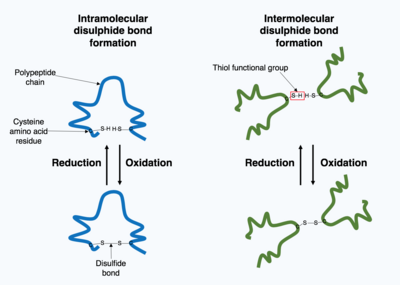
Viele in der Zelle produzierte Proteine werden außerhalb der Zelle sezerniert, um als extrazelluläre Proteine zu wirken. Extrazelluläre Proteine sind einer Vielzahl von Bedingungen ausgesetzt. Um die 3D-Proteinstruktur zu stabilisieren, werden kovalente Bindungen entweder innerhalb des Proteins oder zwischen den verschiedenen Polypeptidketten in der quaternären Struktur gebildet. Die am weitesten verbreitete Art ist die Disulfidbindung (auch als Disulfidbrücke bekannt). Eine Disulfidbindung wird zwischen zwei Cystein-Aminosäuren mit Hilfe ihrer chemischen Gruppen in der Seitenkette gebildet, die ein Schwefelatom enthalten; diese chemischen Gruppen werden als funktionelle Thiolgruppen bezeichnet. Disulfidbindungen dienen der Stabilisierung der bereits vorhandenen Struktur des Proteins. Disulfidbindungen werden durch eine Oxidationsreaktion zwischen zwei Thiolgruppen gebildet und benötigen daher eine oxidierende Umgebung, um zu reagieren. Daher werden Disulfidbindungen in der Regel in der oxidierenden Umgebung des endoplasmatischen Retikulums gebildet, katalysiert durch Enzyme, die Proteindisulfidisomerasen genannt werden. Im Zytoplasma werden Disulfidbindungen nur selten gebildet, da es sich um eine reduzierende Umgebung handelt. ⓘ
Die Rolle der Proteinsynthese bei Krankheiten
Viele Krankheiten werden durch Mutationen in den Genen verursacht, da ein direkter Zusammenhang zwischen der DNA-Nukleotidsequenz und der Aminosäuresequenz des kodierten Proteins besteht. Veränderungen in der Primärstruktur des Proteins können zu einer Fehlfaltung oder Fehlfunktion des Proteins führen. Mutationen innerhalb eines einzigen Gens wurden als Ursache für mehrere Krankheiten, einschließlich der Sichelzellkrankheit, identifiziert, die als Einzelgenstörungen bekannt sind. ⓘ
Sichelzellenkrankheit

Die Sichelzellenanämie ist eine Gruppe von Krankheiten, die durch eine Mutation in einer Untereinheit des Hämoglobins verursacht werden, einem Protein in den roten Blutkörperchen, das für den Sauerstofftransport verantwortlich ist. Die gefährlichste der Sichelzellkrankheiten ist die Sichelzellanämie. Die Sichelzellenanämie ist die häufigste homozygot-rezessiv vererbte Einzelgenstörung, d. h. die betroffene Person muss eine Mutation in beiden Kopien des betroffenen Gens (eine von jedem Elternteil) tragen, um die Krankheit zu bekommen. Hämoglobin hat eine komplexe quaternäre Struktur und besteht aus vier Polypeptiduntereinheiten - zwei A-Untereinheiten und zwei B-Untereinheiten. Patienten mit Sichelzellenanämie haben eine Fehlsens- oder Substitutionsmutation in dem Gen, das für die Polypeptidkette der Hämoglobin-B-Untereinheit kodiert. Eine Missense-Mutation bedeutet, dass die Nukleotidmutation das gesamte Codon-Triplett so verändert, dass eine andere Aminosäure mit dem neuen Codon gepaart wird. Im Falle der Sichelzellenanämie ist die häufigste Missense-Mutation eine einzelne Nukleotidmutation von Thymin zu Adenin im Gen der Hämoglobin-B-Untereinheit. Dadurch wird das Codon 6, das für die Aminosäure Glutaminsäure kodiert, durch Valin ersetzt. ⓘ
Diese Änderung in der Primärstruktur der Polypeptidkette der Hämoglobin-B-Untereinheit verändert die Funktionalität des Hämoglobin-Mehrfachuntereinheitenkomplexes unter sauerstoffarmen Bedingungen. Wenn die roten Blutkörperchen Sauerstoff in das Körpergewebe entladen, beginnt das mutierte Hämoglobinprotein zusammenzukleben und eine halbfeste Struktur innerhalb der roten Blutkörperchen zu bilden. Dadurch wird die Form der roten Blutkörperchen verzerrt, was zu der charakteristischen Sichelform führt, und die Flexibilität der Zellen wird verringert. Diese starren, verzerrten roten Blutkörperchen können sich in den Blutgefäßen ansammeln und eine Verstopfung verursachen. Die Verstopfung verhindert die Durchblutung des Gewebes und kann zum Absterben des Gewebes führen, was für den Betroffenen mit großen Schmerzen verbunden ist. ⓘ
Krebs
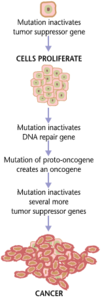
Krebs entsteht durch Genmutationen und eine fehlerhafte Proteinübersetzung. Krebszellen vermehren sich nicht nur abnormal, sie unterdrücken auch die Expression von anti-apoptotischen oder pro-apoptotischen Genen oder Proteinen. Die meisten Krebszellen weisen eine Mutation des Signalproteins Ras auf, das in den Zellen als An- und Ausschalter fungiert. In Krebszellen wird das RAS-Protein dauerhaft aktiv und fördert so die Proliferation der Zelle, da es nicht reguliert wird. Außerdem tragen die meisten Krebszellen zwei mutierte Kopien des Regulatorgens p53, das als Gatekeeper für geschädigte Gene fungiert und in bösartigen Zellen die Apoptose einleitet. Fehlt es, kann die Zelle keine Apoptose einleiten oder anderen Zellen signalisieren, sie zu zerstören. ⓘ
Wenn sich die Tumorzellen vermehren, bleiben sie entweder auf einen Bereich beschränkt und werden als gutartig bezeichnet, oder sie werden zu bösartigen Zellen, die in andere Bereiche des Körpers wandern. Häufig scheiden diese bösartigen Zellen Proteasen aus, die die extrazelluläre Matrix des Gewebes aufbrechen. Dadurch kann der Krebs in sein Endstadium eintreten, das als Metastasierung bezeichnet wird, bei der die Zellen in den Blutkreislauf oder das Lymphsystem eindringen, um in einen neuen Teil des Körpers zu wandern. ⓘ
Proteintargeting und Proteintransport
Da viele Proteine als Zielort (englisch target) nicht das Zytosol, sondern den Extrazellularraum, die Zellmembran, die Organellen wie Chloroplasten, Mitochondrien, Peroxisomen, Zellkern oder Endoplasmatisches Retikulum haben, hat die Zelle verschiedene Mechanismen, die Proteine dorthin zu verbringen. Diese Proteine enthalten meist eine N- oder auch C-terminale Signalsequenz, die je nach Targetmechanismus sehr unterschiedlich aufgebaut sein kann. In einigen Fällen gibt es keine terminale Signalsequenz, sondern interne Signale der Peptidkette, die über den Zielort des Proteins bestimmt. ⓘ
- Proteine, deren Ziel das Endoplasmatische Retikulum (ER) ist, tragen eine spezifische N-terminale Sequenz, die von einem Protein-RNA-Komplex, dem Signal Recognition Particle (SRP), erkannt wird. Der SRP-Peptid-Ribosom-Komplex wird dann zum Endoplasmatischen Retikulum rekrutiert, wo er erkannt und gebunden wird. Die Translation wird durch die Membran fortgesetzt. Durch die anheftenden Ribosomen entsteht der Eindruck eines „rauen ERs“. Siehe Cotranslationaler Proteintransport. Im Endoplasmatischen Retikulum findet die Qualitätskontrolle des neu synthetisierten Proteins statt.
- Proteine, die in die Chloroplasten verbracht werden müssen, besitzen eine N-terminale Signalsequenz, die gewöhnlich früh phosphoryliert wird. Die Proteine Hsp70, 14-3-3 und Toc64 können weiterhin durch Interaktion mit dem Protein-Vorläufer eine Rolle bei der Erkennung und Weiterleitung spielen. Der Protein-Precursor-Komplex wird nach der Ankunft auf der Oberfläche des Chloroplasten von Rezeptorstrukturen des Translokonapparates der äußeren Chloroplastenmebran (Translocon Of Outer Chloroplast Membrane, TOC) erkannt. Unter GTP-Hydrolyse wird das Protein dann in den Intermembranraum importiert oder direkt durch den Translokonapparat (TIC) der inneren Chloroplastenmembran in das Stroma importiert. Für den Import in die Membran oder das Lumen der Thylakoide werden mindestens 4 Wege genutzt, die als Sec-abhängig, SRP-abhängig, delta-pH/Tat-abhängig oder spontan bezeichnet werden.
- Für das Mitochondrium wurden für Hefe- und Tierzellen bislang drei verschiedene Import-Wege beschrieben:
- Der Präsequenz-Importweg, dessen Proteine eine N-terminale amphiphile alpha-Helix tragen. Diese Proteine sind meist für die Matrix, die innere Membran oder den Intermembranraum bestimmt.
- Der Carrier-Protein-Importweg für Proteine der inneren Membran, welche verschiedene interne Signale tragen.
- Der Importweg der Proteine der äußeren Hüllmembran, der zur Integration von Proteinen mit beta-Fass-Motiv genutzt wird. Auch hier liegen sequenzinterne Signale vor. ⓘ
- Alle drei Importwege beginnen am mitochondrialen Translokonapparat in der äußeren Membran (TOM), welcher verschiedene Rezeptoren besitzt. So erkennen die Rezeptoren Tom20 und Tom22 das N-terminale Signal und leiten das Vorläufer-Protein an die Pore Tom40 weiter. Der Rezeptor Tom70 erkennt die internen Signale der Proteine, die für die äußere Membran bestimmt sind.
- Nach dem Import in den Intermembranraum trennen sich die Wege: Die Proteine mit beta-Fass-Motiv, welche für die äußere Membran bestimmt sind, werden durch den SAM-Komplex (Sorting and assembly machinery) in die Membran integriert. Die Proteine der anderen beiden Importwege werden zu verschiedenen TIM-Komplexen dirigiert: Proteine mit Präsequenz werden von dem TIM23-Komplex erkannt, Proteine für die innere Membran dagegen vom TIM22-Komplex.
- Die Präsequenz wird durch das Enzym MPP (englisch mitochondrial processing peptidase) entfernt. ⓘ
Neben den oben beschriebenen Signalsequenzen ermöglicht eine Glykosylierung ein Targeting für den Einbau in die Zellmembran bzw. für die Exozytose. Beide Wege führen meist über Golgi-Vesikel. ⓘ

