Mikrobiom
| Teil einer Serie über ⓘ |
| Mikrobiome |
|---|
 |
|
Ein Mikrobiom (von altgriechisch μικρός (mikrós) "klein" und βίος (bíos) "Leben") ist die Gemeinschaft von Mikroorganismen, die normalerweise in einem bestimmten Lebensraum zusammenleben. Sie wurde 1988 von Whipps et al. genauer definiert als "eine charakteristische mikrobielle Gemeinschaft, die einen einigermaßen klar definierten Lebensraum mit ausgeprägten physikalisch-chemischen Eigenschaften bewohnt. Der Begriff bezieht sich also nicht nur auf die beteiligten Mikroorganismen, sondern umfasst auch deren Aktionsraum". Im Jahr 2020 veröffentlichte ein internationales Expertengremium das Ergebnis seiner Diskussionen über die Definition des Mikrobioms. Sie schlugen eine Definition des Mikrobioms vor, die auf einer Wiederbelebung der "kompakten, klaren und umfassenden Beschreibung des Begriffs" beruht, wie sie ursprünglich von Whipps et al. vorgelegt wurde, aber durch zwei erläuternde Absätze ergänzt wurde. Der erste erläuternde Absatz spricht den dynamischen Charakter des Mikrobioms aus, und der zweite erläuternde Absatz trennt den Begriff Mikrobiota klar vom Begriff Mikrobiom. ⓘ
Die Mikrobiota besteht aus allen lebenden Mitgliedern, die das Mikrobiom bilden. Die meisten Mikrobiomforscher sind sich einig, dass Bakterien, Archaeen, Pilze, Algen und kleine Protisten als Mitglieder des Mikrobioms betrachtet werden sollten. Die Integration von Phagen, Viren, Plasmiden und mobilen genetischen Elementen ist eher umstritten. Whipps' "Theater der Aktivität" umfasst die wesentliche Rolle, die Sekundärmetaboliten bei der Vermittlung komplexer Interaktionen zwischen den Arten und bei der Sicherung des Überlebens in einer konkurrenzbetonten Umgebung spielen. Das durch kleine Moleküle ausgelöste Quorum Sensing ermöglicht es den Bakterien, kooperative Aktivitäten zu steuern und ihren Phänotyp an die biotische Umwelt anzupassen, was beispielsweise zur Zell-Zell-Adhäsion oder Biofilmbildung führt. ⓘ
Alle Tiere und Pflanzen gehen Verbindungen mit Mikroorganismen ein, darunter Protisten, Bakterien, Archaeen, Pilze und Viren. Im Ozean wurden die Beziehungen zwischen Tieren und Mikroorganismen in der Vergangenheit in einzelnen Wirts-Symbionten-Systemen erforscht. Neue Untersuchungen der Vielfalt von Mikroorganismen, die mit verschiedenen marinen Tierwirten assoziiert sind, verlagern das Feld jedoch auf Studien, die sich mit den Wechselwirkungen zwischen dem tierischen Wirt und dem vielgliedrigen Mikrobiom befassen. Das Potenzial des Mikrobioms, die Gesundheit, die Physiologie, das Verhalten und die Ökologie von Meerestieren zu beeinflussen, könnte das derzeitige Verständnis darüber verändern, wie sich Meerestiere an Veränderungen anpassen. Dies gilt insbesondere für die zunehmenden klimabedingten und anthropogenen Veränderungen, die sich bereits auf den Ozean auswirken. Das pflanzliche Mikrobiom spielt eine Schlüsselrolle für die Pflanzengesundheit und die Nahrungsmittelproduktion und hat in den letzten Jahren große Aufmerksamkeit erhalten. Pflanzen leben in Verbindung mit verschiedenen mikrobiellen Konsortien, die als Pflanzenmikrobiota bezeichnet werden und sowohl innerhalb (Endosphäre) als auch außerhalb (Episphäre) des Pflanzengewebes leben. Sie spielen eine wichtige Rolle in der Ökologie und Physiologie der Pflanzen. Man geht davon aus, dass das pflanzliche Kernmikrobiom wichtige mikrobielle Taxa enthält, die für die Pflanzengesundheit und die Fitness des Pflanzenholobionten von entscheidender Bedeutung sind. Ebenso hat sich das Darmmikrobiom von Säugetieren als wichtiger Regulator der Wirtsphysiologie erwiesen, und die Koevolution zwischen Wirts- und Mikrobenlinien hat eine Schlüsselrolle bei der Anpassung der Säugetiere an ihre vielfältigen Lebensweisen gespielt. ⓘ
Die Mikrobiomforschung hat ihren Ursprung in der Mikrobiologie im siebzehnten Jahrhundert. Die Entwicklung neuer Techniken und Geräte gab der mikrobiologischen Forschung Auftrieb und bewirkte einen Paradigmenwechsel im Verständnis von Gesundheit und Krankheit. Die Entwicklung der ersten Mikroskope ermöglichte die Entdeckung einer neuen, unbekannten Welt und führte zur Identifizierung von Mikroorganismen. Infektionskrankheiten standen von Anfang an im Mittelpunkt des Interesses und der Forschung. Allerdings wird nur ein kleiner Teil der Mikroorganismen mit Krankheit oder Pathogenität in Verbindung gebracht. Die überwältigende Mehrheit der Mikroben ist für das gesunde Funktionieren von Ökosystemen unerlässlich und für ihre positiven Wechselwirkungen mit anderen Mikroben und Organismen bekannt. Die Vorstellung, dass Mikroorganismen als einzelne Zellen existieren, begann sich zu ändern, als immer deutlicher wurde, dass Mikroben in komplexen Verbänden vorkommen, in denen die Interaktionen zwischen den Arten und die Kommunikation entscheidend sind. Die Entdeckung der DNA, die Entwicklung von Sequenzierungstechnologien, PCR und Klonierungstechniken ermöglichten die Untersuchung mikrobieller Gemeinschaften mit kultivierungsunabhängigen Ansätzen. Zu Beginn dieses Jahrhunderts kam es zu weiteren Paradigmenwechseln, die bis heute andauern, da neue Sequenzierungstechnologien und gesammelte Sequenzdaten sowohl die Allgegenwart mikrobieller Gemeinschaften in Verbindung mit höheren Organismen als auch die entscheidende Rolle von Mikroben für die Gesundheit von Menschen, Tieren und Pflanzen deutlich gemacht haben. Dies hat die mikrobielle Ökologie revolutioniert. Die Analyse von Genomen und Metagenomen im Hochdurchsatzverfahren bietet nun hocheffektive Methoden zur Erforschung der Funktionsweise sowohl einzelner Mikroorganismen als auch ganzer mikrobieller Gemeinschaften in natürlichen Lebensräumen. ⓘ

Die Ausdrücke Mikrobiom, Mikrobiota und Mikroflora (griechisch μικρός mikrós ‚klein‘, griechisch βίος bios ‚Leben‘) bezeichnen im weitesten Sinn die Gesamtheit aller Mikroorganismen der Erde (als Teil des Bioms), welche die Erdkruste, die Gewässer und die Erdatmosphäre besiedeln. ⓘ
Im engeren Sinn ist die Gesamtheit aller Mikroorganismen gemeint, die ein vielzelliges Lebewesen natürlicherweise (d. h. ohne Auslösung von Krankheitssymptomen) besiedeln. Es werden auch spezielle Teil-Mikrobiome/-Mikrobiota des Körpers eines solchen Lebewesens (also von bestimmten Körperteilen oder -bereichen) mithilfe der Bezeichnungen „Mikrobiom“ und „Mikrobiota“ beschrieben. Sie werden speziell erforscht, so auch die Wechselwirkungen zwischen diesen Teil-Mikrobiomen eines solchen Lebewesens und auch zwischen den (Teil-)Mikrobiomen verschiedener solcher Lebewesen (also z. B. zwischen den Mikrobiomen der Münder zweier Kusspartner), etwa im Rahmen der Immunologie. ⓘ
Das Mykobiom ist die Gesamtheit aller im Mikrobiom enthaltenen Pilze. ⓘ
Hintergrund
Geschichte
Die Mikrobiomforschung hat ihren Ursprung in der Mikrobiologie und begann bereits im siebzehnten Jahrhundert. Die Entwicklung neuer Techniken und Geräte hat die mikrobiologische Forschung vorangetrieben und einen Paradigmenwechsel im Verständnis von Gesundheit und Krankheit bewirkt. Da Infektionskrankheiten die menschliche Bevölkerung fast die gesamte Geschichte hindurch betroffen haben, stand die medizinische Mikrobiologie schon früh im Mittelpunkt der Forschung und des öffentlichen Interesses. Darüber hinaus ist die Lebensmittelmikrobiologie ein altes Feld empirischer Anwendungen. Die Entwicklung der ersten Mikroskope ermöglichte die Entdeckung einer neuen, unbekannten Welt und führte zur Identifizierung von Mikroorganismen. ⓘ
- Paradigmenwechsel
Paradigmenwechsel von Mikroben als unsoziale Organismen, die Krankheiten verursachen, hin zu einer ganzheitlichen Betrachtung der Mikroorganismen als Mittelpunkt des One Health-Konzepts, das alle Bereiche des menschlichen Lebens miteinander verbindet. ⓘ
Der Zugang zu der bis dahin unsichtbaren Welt öffnete den Forschern des siebzehnten Jahrhunderts die Augen und den Verstand. Antonie van Leeuwenhoek untersuchte verschiedene Bakterien unterschiedlicher Formen, Pilze und Protozoen, die er als "animalcules" bezeichnete, vor allem aus Wasser-, Schlamm- und Zahnbelagsproben und entdeckte Biofilme als ersten Hinweis auf Mikroorganismen, die in komplexen Gemeinschaften zusammenwirken. Robert Kochs Erklärung der Entstehung von Krankheiten bei Mensch und Tier als Folge einer mikrobiellen Infektion und die Entwicklung des Konzepts der Pathogenität waren ein wichtiger Meilenstein in der Mikrobiologie. Diese Erkenntnisse rückten die Rolle von Mikroorganismen als Krankheitserreger, die es zu beseitigen gilt, in den Mittelpunkt der Forschung und der Öffentlichkeit. ⓘ
Die umfassende Forschung des letzten Jahrhunderts hat jedoch gezeigt, dass nur ein kleiner Teil der Mikroorganismen mit Krankheiten oder Pathogenität in Verbindung gebracht wird. Die überwältigende Mehrheit der Mikroben ist für das Funktionieren des Ökosystems unverzichtbar und für nützliche Interaktionen mit anderen Mikroben und Makroorganismen bekannt. Tatsächlich ist die Aufrechterhaltung eines gesunden Mikrobioms für die menschliche Gesundheit von entscheidender Bedeutung und könnte ein Ziel für neue Therapeutika sein. Jahrhunderts begann die mikrobielle Ökologie mit der bahnbrechenden Arbeit von Martinus W. Beijerinck und Sergei Winogradsky. Die neu etablierte Wissenschaft der Umweltmikrobiologie führte zu einem weiteren Paradigmenwechsel: Mikroorganismen sind in der natürlichen Umwelt allgegenwärtig, oft in Verbindung mit ihren Wirten, und zum ersten Mal wurde über positive Auswirkungen auf ihre Wirte berichtet. ⓘ
In der Folgezeit begann sich das Konzept, dass Mikroorganismen als einzelne Zellen existieren, zu ändern, da es immer offensichtlicher wurde, dass Mikroben in komplexen Assemblagen vorkommen, in denen die Interaktionen und die Kommunikation zwischen den Arten für die Populationsdynamik und die funktionellen Aktivitäten entscheidend sind. Die Entdeckung der DNA, die Entwicklung von Sequenzierungstechnologien, PCR und Klonierungstechniken ermöglichten die Untersuchung mikrobieller Gemeinschaften mit kultivierungsunabhängigen, DNA- und RNA-basierten Ansätzen. ⓘ
Ein weiterer wichtiger Schritt war die Einführung von phylogenetischen Markern wie dem 16S rRNA-Gen für die Analyse mikrobieller Gemeinschaften durch Carl Woese und George E. Fox im Jahr 1977. Heutzutage können Biologen Bakterien, Archaeen, Pilze, Algen und Protisten in ihren natürlichen Lebensräumen mit einem Barcode versehen, indem sie z. B. ihre 16S- und 18S-rRNA-Gene, den internen transkribierten Spacer (ITS) oder alternativ bestimmte funktionelle Regionen von Genen, die für bestimmte Enzyme kodieren, anvisieren. ⓘ
Ein weiterer wichtiger Paradigmenwechsel wurde zu Beginn dieses Jahrhunderts eingeleitet und setzt sich bis heute fort, da neue Sequenzierungstechnologien und akkumulierte Sequenzdaten sowohl die Allgegenwart mikrobieller Gemeinschaften in Verbindung mit höheren Organismen als auch die entscheidende Rolle von Mikroben für die Gesundheit von Menschen, Tieren und Pflanzen deutlich gemacht haben. Diese neuen Möglichkeiten haben die mikrobielle Ökologie revolutioniert, denn die Analyse von Genomen und Metagenomen im Hochdurchsatzverfahren bietet effiziente Methoden, um das funktionelle Potenzial einzelner Mikroorganismen sowie ganzer Gemeinschaften in ihren natürlichen Lebensräumen zu untersuchen. Multiomics-Technologien einschließlich Metatranskriptom-, Metaproteom- und Metabolom-Ansätzen liefern jetzt detaillierte Informationen über mikrobielle Aktivitäten in der Umwelt. Auf der Grundlage dieser reichhaltigen Datenbasis hat die Kultivierung von Mikroben, die in den letzten dreißig Jahren oft ignoriert oder unterschätzt wurde, neue Bedeutung erlangt, und die Hochdurchsatz-Kulturomik ist jetzt ein wichtiger Bestandteil des Instrumentariums zur Untersuchung von Mikrobiomen. Das große Potenzial und die Leistungsfähigkeit der Kombination mehrerer "Omics"-Techniken zur Analyse von Wirt-Mikroben-Interaktionen werden in mehreren Übersichtsarbeiten hervorgehoben. ⓘ
| Zeitleiste der Mikrobiomforschung vom siebzehnten Jahrhundert bis heute ⓘ | ||||
|---|---|---|---|---|
Technologische Fortschritte
|
Jahr | Wissenschaftliche Entdeckungen
|
Wissenschaftler | Quellen |
| Mikroskopie | 1670 | Entdeckung der Mikroorganismen | Antonie van Leeuwenhoek Vater der Mikrobiologie |
|
| 1729 | Klassifizierung von Pflanzen und Pilzen | Pier Antonio Micheli | ||
| 1796 | erste Impfung | Edward Jenner | ||
| 1837 | Hefe bei der alkoholischen Gärung | Charles de la Tour Friedrich Kützing Theodor Schwann |
||
| kultivierungsbasierte Ansätze | 1855 -1857 |
Pasteurisierung, Gärung, Impfstoff gegen Tollwut |
Ludwig Pasteur | |
| 1875 | Grundlage für die bakteriologische Taxonomie | Ferdinand Cohn | ||
| 1884 | Die Kochschen Postulate | Robert Koch | ||
| 1888 | Beginn der mikrobiellen Ökologie Nitrifikation, Stickstoff-Fixierung, Bodenmikrobiologie, Lebenszyklus |
Sergej Winogradski | ||
| 1892 | Extraktion des Tabakmosaikvirus aus Blättern | Dmitri Iwanowsky Martinus Beijerinck |
||
| 1904 | Konzept der Rhizosphäre | Lorenz Hiltner | ||
| Fluoreszenzmikroskopie | 1911 | |||
| Massenspektrometrie | 1919 | Franz Aston | ||
| 1922 | Chemolithotrophie | Sergej Winogradski | ||
| 1928 | Übertragung der genetischen Information auf Nachkommen |
Frederick Griffith | ||
| 1928 | Entdeckung von Antibiotika | Alexander Fleming | ||
| Rasterelektronenmikroskopie | 1931 -1938 |
|||
| 1944 | DNA als Träger der genetischen Information | Oswald Avery Colin Macleod Maclyn McCarty |
||
| 1946 | "Sexuelle Fortpflanzung" von Bakterien | Joshua Lederberg Edward Tatum |
||
| 1953 | 3D-Doppelhelix-Struktur | James Watson Francis Crick |
||
| In-situ-Hybridisierung iSIS | 1969 | |||
| HPLC | 1970s | zentrales Dogma der Molekularbiologie | Francis Crick | |
| DNA-Array/Kolonie-Hybridisierung | 1975 | |||
| Sanger-Sequenzierung | 1977 | Frederick Sanger | ||
| 1977 | Entdeckung der Archaea | Carl Woese George E. Fox |
||
| 1977 | Erste vollständige Genomsequenz eines Virus | |||
| 1982 | Entdeckung der Prionen | Stanley B. Prusiner | ||
| PCR-Technik | 1983 | |||
| Fluoreszenz-in-situ-Hybridisierung | 1988 | |||
| 1991 | Theorie des Holobionten | Eugene Rosenberg Ilana Zilber-Rosenberg |
||
| Quantitative Echtzeit-PCR | 1993 | |||
| 1993 | die komplexe Struktur von Biofilmen | Hans-Curt Flemming | ||
| rRNA-Vollzyklus-Ansatz | 1995 | |||
| 1995 | erste vollständige Genomsequenzierung von Haemophilus influenzae |
Craig Venter und Kollegen |
||
| Sequenzierung der nächsten Generation | 2005 | |||
| 2005 | HMP: Menschliches Mikrobiom-Projekt | |||
| Sequenzierung der dritten Generation | 2008 | |||
| 2008 | TerraGenom: Referenz-Boden-Metagenom-Projekt |
|||
| 2010 | Erd-Mikrobiom-Projekt | |||
Etymologie
Das Wort Mikrobiom (aus dem Griechischen micro für "klein" und bíos für "Leben") wurde erstmals 1952 von J.L. Mohr in The Scientific Monthly verwendet, um die in einer bestimmten Umgebung vorkommenden Mikroorganismen zu bezeichnen. ⓘ
Definitionen
Mikrobengemeinschaften werden gemeinhin als eine Ansammlung von zusammenlebenden Mikroorganismen definiert. Genauer gesagt werden mikrobielle Gemeinschaften als artenreiche Ansammlungen definiert, in denen (Mikro-)Organismen in einer zusammenhängenden Umgebung miteinander interagieren. Im Jahr 1988 lieferten Whipps und Kollegen, die sich mit der Ökologie von Mikroorganismen in der Rhizosphäre beschäftigten, die erste Definition des Begriffs Mikrobiom. Sie beschrieben das Mikrobiom als eine Kombination aus den Wörtern Mikro und Biom und bezeichneten eine "charakteristische mikrobielle Gemeinschaft" in einem "einigermaßen gut definierten Lebensraum mit ausgeprägten physikalisch-chemischen Eigenschaften" als ihren "Wirkungsort". Diese Definition stellt eine wesentliche Weiterentwicklung der Definition einer mikrobiellen Gemeinschaft dar, da sie eine mikrobielle Gemeinschaft mit bestimmten Eigenschaften und Funktionen und ihren Interaktionen mit ihrer Umwelt definiert, die zur Bildung spezifischer ökologischer Nischen führen. ⓘ
In den letzten Jahrzehnten wurden jedoch viele andere Mikrobiom-Definitionen veröffentlicht. Die bis 2020 am häufigsten zitierte Definition stammte von Lederberg und beschrieb Mikrobiome in einem ökologischen Kontext als eine Gemeinschaft von kommensalen, symbiotischen und pathogenen Mikroorganismen in einem Körperraum oder einer anderen Umgebung. Marchesi und Ravel konzentrierten sich in ihrer Definition auf die Genome und mikrobiellen (und viralen) Genexpressionsmuster und Proteome in einer bestimmten Umgebung und deren vorherrschenden biotischen und abiotischen Bedingungen. All diese Definitionen implizieren, dass allgemeine Konzepte der Makroökologie leicht auf Interaktionen zwischen Mikroben und Mikroben sowie zwischen Mikroben und Wirt angewendet werden können. Es ist jedoch nicht ganz klar, inwieweit diese für Makro-Eukaryoten entwickelten Konzepte auf Prokaryoten mit ihren unterschiedlichen Lebensweisen in Bezug auf Dormanz, Variation des Phänotyps und horizontalen Gentransfer sowie auf Mikro-Eukaryoten angewendet werden können. Daraus ergibt sich die Herausforderung, einen völlig neuen Korpus an konzeptionellen Ökologiemodellen und -theorien für die Mikrobiom-Ökologie in Betracht zu ziehen, insbesondere in Bezug auf die vielfältigen Hierarchien der Interaktionen von Mikroben untereinander und mit der biotischen und abiotischen Umwelt des Wirts. Viele aktuelle Definitionen erfassen diese Komplexität nicht und beschreiben den Begriff Mikrobiom so, dass er nur die Genome der Mikroorganismen umfasst. ⓘ
| Mikrobiom-Definitionen ⓘ | |
|---|---|
| Art der Definition | Beispiele |
| Ökologisch | Auf der Ökologie basierende Definitionen beschreiben das Mikrobiom in Anlehnung an die aus der Ökologie mehrzelliger Organismen abgeleiteten Konzepte. Das Hauptproblem dabei ist, dass die Theorien aus der Makroökologie nicht immer zu den Regeln in der mikrobiellen Welt passen. |
| |
| Organismen/Wirt-abhängig | Die wirtsabhängigen Definitionen beruhen auf den mikrobiellen Interaktionen mit dem Wirt. Die Hauptlücken betreffen hier die Frage, ob die von einem Wirt gewonnenen Daten über Mikroben-Wirt-Interaktionen auf einen anderen Wirt übertragen werden können. Auch das Verständnis von Koevolution und Selektion ist in den wirtsabhängigen Definitionen unterrepräsentiert. |
| |
| Genomisch/ methodengesteuert | Es gibt eine Vielzahl von Mikrobiom-Definitionen, die sich nach den angewandten Methoden richten. Meistens stützen sich diese Definitionen auf DNA-Sequenzanalysen und beschreiben das Mikrobiom als kollektives Genom von Mikroorganismen in einer bestimmten Umgebung. Der größte Engpass besteht darin, dass jede neue verfügbare Technologie eine neue Definition erforderlich macht. |
| |
| Kombiniert | Es gibt einige Mikrobiom-Definitionen, die mehrere Kategorien mit ihren Vor- und Nachteilen abdecken. |
| |
Im Jahr 2020 veröffentlichte ein internationales Expertengremium, das von dem von der EU finanzierten Projekt MicrobiomeSupport organisiert wurde, die Ergebnisse seiner Überlegungen zur Definition des Mikrobioms. Das Gremium setzte sich aus etwa 40 führenden Persönlichkeiten aus verschiedenen Bereichen des Mikrobioms zusammen, und etwa einhundert weitere Experten aus der ganzen Welt trugen über eine Online-Umfrage bei. Sie schlugen eine Definition des Mikrobioms vor, die auf einer Wiederbelebung dessen beruht, was sie als "kompakte, klare und umfassende Beschreibung des Begriffs" bezeichneten, wie sie ursprünglich von Whipps et al. im Jahr 1988 vorgelegt wurde, ergänzt durch eine Reihe von Empfehlungen unter Berücksichtigung späterer technologischer Entwicklungen und Forschungsergebnisse. Sie grenzen die Begriffe Mikrobiom und Mikrobiota klar voneinander ab und bieten eine umfassende Diskussion über die Zusammensetzung der Mikrobiota, die Heterogenität und Dynamik von Mikrobiomen in Zeit und Raum, die Stabilität und Widerstandsfähigkeit mikrobieller Netzwerke, die Definition von Kernmikrobiomen und funktionell relevanten Schlüsselspezies sowie koevolutionäre Prinzipien von Mikroben-Wirt- und Inter-Spezies-Interaktionen innerhalb des Mikrobioms. ⓘ
Das Gremium erweiterte die Definition von Whipps et al., die alle wichtigen Punkte enthält, die auch 30 Jahre nach ihrer Veröffentlichung im Jahr 1988 noch gültig sind, um zwei erläuternde Absätze, die die Begriffe Mikrobiom und Mikrobiota voneinander abgrenzen und den dynamischen Charakter des Mikrobioms betonen, wie folgt:
- Das Mikrobiom ist definiert als eine charakteristische mikrobielle Gemeinschaft, die einen angemessenen, genau definierten Lebensraum mit ausgeprägten physikalisch-chemischen Eigenschaften bewohnt. Das Mikrobiom bezieht sich nicht nur auf die beteiligten Mikroorganismen, sondern umfasst auch deren Aktionsraum, was zur Bildung spezifischer ökologischer Nischen führt. Das Mikrobiom, das ein dynamisches und interaktives Mikroökosystem bildet, das sich in Zeit und Ausmaß verändern kann, ist in Makroökosysteme, einschließlich eukaryontischer Wirte, integriert und für deren Funktion und Gesundheit von entscheidender Bedeutung. ⓘ
- Die Mikrobiota besteht aus der Gesamtheit von Mikroorganismen, die verschiedenen Reichen angehören (Prokaryonten (Bakterien, Archaeen), Eukaryonten (Algen, Protozoen, Pilze usw.), während das "Theater ihrer Aktivität" mikrobielle Strukturen, Metaboliten, mobile genetische Elemente (wie Transposons, Phagen und Viren) und in die Umweltbedingungen des Lebensraums eingebettete DNA-Relikte umfasst. ⓘ
Zugehörigkeit
Mikrobiota
Die Mikrobiota umfasst alle lebenden Mitglieder, die das Mikrobiom bilden. Die meisten Mikrobiomforscher sind sich einig, dass Bakterien, Archaeen, Pilze, Algen und kleine Protisten als Mitglieder des Mikrobioms betrachtet werden sollten. Die Integration von Phagen, Viren, Plasmiden und mobilen genetischen Elementen ist ein umstritteneres Thema bei der Definition des Mikrobioms. Es besteht auch kein eindeutiger Konsens darüber, ob extrazelluläre DNA, die von toten Zellen stammt, so genannte "Relikt-DNA", zum Mikrobiom gehört. Relikt-DNA kann bis zu 40 % der sequenzierten DNA im Boden ausmachen und machte in einer breiteren Analyse von Lebensräumen im Durchschnitt bis zu 33 % der gesamten bakteriellen DNA aus, wobei der höchste Anteil in einigen Proben 80 % betrug. Trotz ihrer Omnipräsenz und Häufigkeit hatte die Relikt-DNA nur minimale Auswirkungen auf die Schätzungen der taxonomischen und phylogenetischen Vielfalt. ⓘ
Wenn es um die Verwendung spezifischer Begriffe geht, hilft eine klare Unterscheidung zwischen Mikrobiom und Mikrobiota, die Kontroverse über die Mitglieder eines Mikrobioms zu vermeiden. Die Mikrobiota wird in der Regel als die Gesamtheit der lebenden Mikroorganismen in einer bestimmten Umgebung definiert. Da Phagen, Viren, Plasmide, Prionen, Viroide und freie DNA in der Regel nicht als lebende Mikroorganismen betrachtet werden, gehören sie nicht zur Mikrobiota. ⓘ
Der Begriff Mikrobiom, wie er ursprünglich von Whipps und Mitarbeitern postuliert wurde, umfasst nicht nur die Gemeinschaft der Mikroorganismen, sondern auch deren "Aktionsraum". Letzteres umfasst das gesamte Spektrum der von den Mikroorganismen produzierten Moleküle, einschließlich ihrer Strukturelemente (Nukleinsäuren, Proteine, Lipide, Polysaccharide), Metaboliten (Signalmoleküle, Toxine, organische und anorganische Moleküle) sowie Moleküle, die von koexistierenden Wirten produziert und durch die Umgebungsbedingungen strukturiert werden. Daher sollten alle mobilen genetischen Elemente wie Phagen, Viren, "Relikte" und extrazelluläre DNA in den Begriff Mikrobiom einbezogen werden, sind aber nicht Teil der Mikrobiota. Der Begriff Mikrobiom wird manchmal auch mit dem Metagenom verwechselt. Das Metagenom ist jedoch eindeutig definiert als eine Sammlung von Genomen und Genen der Mitglieder einer Mikrobiota. ⓘ
Mikrobiomstudien konzentrieren sich manchmal auf das Verhalten einer bestimmten Gruppe von Mikrobiota, im Allgemeinen in Verbindung mit einer klaren Hypothese oder durch diese begründet. In der wissenschaftlichen Literatur tauchen immer mehr Begriffe wie Bakteriom, Archäom, Mykobiom oder Viom auf, aber diese Begriffe beziehen sich nicht auf Biome (ein regionales Ökosystem mit einer bestimmten Ansammlung von (Mikro-)Organismen und einer physikalischen Umgebung, die oft ein bestimmtes Klima und einen bestimmten Boden widerspiegelt) als das Mikrobiom selbst. Daher wäre es besser, die ursprünglichen Begriffe (Bakterien-, Archaeen- oder Pilzgemeinschaft) zu verwenden. Im Gegensatz zur Mikrobiota, die separat untersucht werden kann, setzt sich das Mikrobiom immer aus allen Mitgliedern zusammen, die miteinander interagieren, im selben Lebensraum leben und ihre ökologische Nische gemeinsam bilden. Der etablierte Begriff Virom leitet sich von Virus und Genom ab und wird verwendet, um virale Schrotflinten-Metagenome zu beschreiben, die aus einer Sammlung von Nukleinsäuren bestehen, die mit einem bestimmten Ökosystem oder Holobionten verbunden sind. Virale Metagenome kann als semantisch und wissenschaftlich besserer Begriff vorgeschlagen werden. ⓘ
Netzwerke
Co-occurrence-Netzwerke helfen bei der Visualisierung mikrobieller Interaktionen
Knoten repräsentieren in der Regel Taxa von Mikroorganismen, und Kanten stellen statistisch signifikante Assoziationen zwischen Knoten dar.
–––––––––––––––––––––––––––
Für eine umfassende Untersuchung der mikrobiellen Interaktionen müssen die aus den Netzwerkanalysen resultierenden Hypothesen überprüft werden. ⓘ
Mikroben interagieren miteinander, und diese symbiotischen Interaktionen haben vielfältige Auswirkungen auf die mikrobielle Fitness, die Populationsdynamik und die funktionellen Kapazitäten innerhalb des Mikrobioms. Die mikrobiellen Interaktionen können entweder zwischen Mikroorganismen der gleichen Art oder zwischen verschiedenen Arten, Gattungen, Familien und Lebensbereichen stattfinden. Die Interaktionen können in positive, negative und neutrale Typen unterteilt werden. Zu den positiven Interaktionen gehören Mutualismus, Synergismus und Kommensalismus. Zu den negativen Interaktionen gehören Amensalismus wie Raub, Parasitismus, Antagonismus und Konkurrenz. Neutrale Interaktionen sind Interaktionen, bei denen keine Auswirkungen auf die funktionellen Kapazitäten oder die Fitness der interagierenden Spezies beobachtet werden können Mikrobielle Lebensstrategiekonzepte. ⓘ
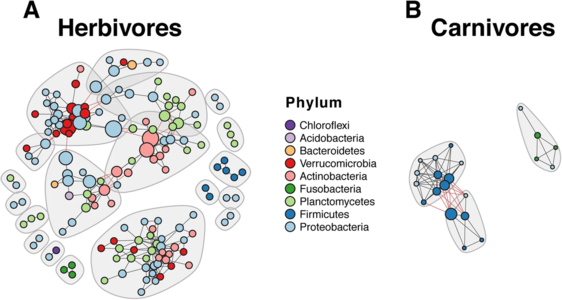
Koinzidenznetzwerke zeigen Unterschiede in der Darmmikrobiota zwischen pflanzenfressenden und fleischfressenden Buntbarschen
Die Knoten sind je nach Stamm gefärbt. Das Netzwerk der Pflanzenfresser ist komplexer (156 Knoten und 339 Kanten) als das der Fleischfresser (21 Knoten und 70 Kanten). ⓘ
Mikrobiome weisen unterschiedliche Anpassungsstrategien auf. Oligotrophe Organismen sind Organismen, die in einer Umgebung leben können, die sehr geringe Mengen an Nährstoffen, insbesondere Kohlenstoff, enthält. Sie zeichnen sich durch langsames Wachstum, niedrige Stoffwechselraten und eine allgemein geringe Populationsdichte aus. Zu den oligotrophen Lebensräumen gehören tiefe ozeanische Sedimente, Höhlen, Gletscher- und Polareis, tiefe unterirdische Böden, Aquifere, Meerwasser und ausgelaugte Böden. Im Gegensatz dazu stehen die kopiotrophen Organismen, die in viel höheren Kohlenstoffkonzentrationen gedeihen und sich unter Bedingungen mit hohem organischem Substrat, wie z. B. in Klärteichen, wohlfühlen. ⓘ
Zusätzlich zu den oligotrophen und copiotrophen Strategen kann das System aus Konkurrenten, Stresstoleranz und Ruderalismus die Ergebnisse von Interaktionen beeinflussen. So können beispielsweise Mikroorganismen, die um dieselbe Quelle konkurrieren, auch voneinander profitieren, wenn sie auf unterschiedlichen trophischen Ebenen um dieselbe Verbindung konkurrieren. Die Stabilität eines komplexen mikrobiellen Ökosystems hängt von den trophischen Interaktionen um dasselbe Substrat auf verschiedenen Konzentrationsstufen ab. Seit 2020 sind die sozialen Anpassungen von Mikroorganismen in der Natur noch wenig erforscht. Hier können molekulare Marker Aufschluss über soziale Anpassungen geben, indem sie die Theorien, z. B. von Altruisten und Betrügern in einheimischen Mikrobiomen, unterstützen. ⓘ
Koevolution
- Verschiebung des Verständnisses der Koevolution zwischen Mikroben und Wirten
In einem ganzheitlichen Ansatz wird davon ausgegangen, dass sich die Wirte und die mit ihnen verbundenen Mikrobiota gemeinsam entwickelt haben. ⓘ
Nach dem "Trennungs"-Ansatz können die Mikroorganismen je nach ihrer Interaktion mit ihrem Wirt in Pathogene, Neutrale und Symbionten unterteilt werden. Die Koevolution zwischen Wirt und seiner assoziierten Mikrobiota kann dementsprechend als antagonistisch (basierend auf negativen Interaktionen) oder mutualistisch (basierend auf positiven Interaktionen) beschrieben werden. ⓘ
Ab 2020 hat das Aufkommen von Veröffentlichungen über opportunistische Pathogene und Pathobionten zu einer Verschiebung hin zu einem holistischen Ansatz in der Koevolutionstheorie geführt. Der holistische Ansatz betrachtet den Wirt und die mit ihm verbundene Mikrobiota als eine Einheit (den sogenannten Holobionten), die sich als eine Einheit koevolviert. Nach dem holistischen Ansatz ist der Krankheitszustand des Holobionten mit Dysbiose, geringer Diversität der assoziierten Mikrobiota und deren Variabilität verbunden: ein so genannter pathobiomischer Zustand. Der gesunde Zustand hingegen ist mit Eubiose, hoher Diversität und Einheitlichkeit der jeweiligen Mikrobiota verbunden. ⓘ
Arten
Meer
- Beziehung zwischen Wirt und Mikrobiom bei Meerestieren
Im Allgemeinen geht man davon aus, dass Beziehungen in einem symbiotischen Zustand existieren und normalerweise umwelt- und tierbezogenen Faktoren ausgesetzt sind, die natürliche Veränderungen verursachen können. Einige Ereignisse können die Beziehung in einen funktionierenden, aber veränderten symbiotischen Zustand versetzen, während extreme Stressereignisse eine Dysbiose oder einen Zusammenbruch der Beziehung und der Interaktionen verursachen können. ⓘ
Alle Tiere auf der Erde gehen Verbindungen mit Mikroorganismen ein, darunter Protisten, Bakterien, Archaeen, Pilze und Viren. Im Meer wurden die Beziehungen zwischen Tieren und Mikroorganismen in der Vergangenheit in einzelnen Wirts-Symbionten-Systemen erforscht. Neue Untersuchungen der Vielfalt von Mikroorganismen, die mit verschiedenen marinen Tierwirten assoziiert sind, verlagern das Feld jedoch auf Studien, die sich mit den Interaktionen zwischen dem tierischen Wirt und einem mehrgliedrigen Mikrobiom befassen. Das Potenzial des Mikrobioms, die Gesundheit, Physiologie, das Verhalten und die Ökologie von Meerestieren zu beeinflussen, könnte das derzeitige Verständnis darüber verändern, wie sich Meerestiere an Veränderungen anpassen, insbesondere an die zunehmenden klimabedingten und anthropogenen Veränderungen, die sich bereits auf die Meeresumwelt auswirken. ⓘ
Derzeit werden die Mikrobiome verschiedener Meerestiere untersucht, von einfachen Organismen wie Schwämmen und Ctenophoren bis hin zu komplexeren Organismen wie Seescheiden und Haien. ⓘ
Die Beziehung zwischen dem hawaiianischen Bobtail-Tintenfisch und dem biolumineszenten Bakterium Aliivibrio fischeri ist eine der am besten untersuchten symbiotischen Beziehungen im Meer und stellt ein bevorzugtes System für die allgemeine Symbioseforschung dar. Diese Beziehung hat Einblicke in grundlegende Prozesse bei tierisch-mikrobiellen Symbiosen und insbesondere in die biochemischen Interaktionen und die Signalübertragung zwischen Wirt und Bakterium gewährt. ⓘ
Der darmlose marine Oligochaetenwurm Olavius algarvensis ist ein weiterer relativ gut untersuchter mariner Wirt für Mikroben. Diese drei Zentimeter langen Würmer leben in flachen Meeressedimenten des Mittelmeers. Die Würmer haben weder ein Maul noch ein Verdauungs- oder Ausscheidungssystem, sondern ernähren sich mit Hilfe einer Reihe von extrazellulären bakteriellen Endosymbionten, die auf einer koordinierten Nutzung des in der Umgebung vorhandenen Schwefels beruhen. Dieses System hat von einigen der fortschrittlichsten "Omics"- und Visualisierungswerkzeuge profitiert. So hat beispielsweise die Mehrfachmarkierung die Visualisierung des Mikrobioms verbessert, und Transkriptomik und Proteomik wurden zur Untersuchung der Interaktionen zwischen Wirt und Mikrobiom eingesetzt, einschließlich der Energieübertragung zwischen Wirt und Mikroben und der Erkennung der Konsortien durch das angeborene Immunsystem des Wurms. Die große Stärke dieses Systems besteht darin, dass es die Möglichkeit bietet, Wirt-Mikrobiom-Interaktionen mit einem mikrobiellen Konsortium von geringer Diversität zu untersuchen, und es bietet auch eine Reihe von genomischen Ressourcen des Wirts und der Mikroben ⓘ

Korallen sind eines der häufigsten Beispiele für einen tierischen Wirt, dessen Symbiose mit Mikroalgen in eine Dysbiose umschlagen kann, was sich als Bleichen bemerkbar macht. Das Mikrobiom von Korallen wurde in einer Reihe von Studien untersucht, die zeigen, wie sich Schwankungen der Meeresumwelt, insbesondere Temperatur, Licht und anorganische Nährstoffe, auf die Häufigkeit und Leistungsfähigkeit der Mikroalgensymbionten sowie auf die Kalkbildung und Physiologie des Wirts auswirken. Studien haben auch gezeigt, dass die ansässigen Bakterien, Archaeen und Pilze zusätzlich zum Nährstoff- und organischen Stoffkreislauf in der Koralle beitragen, wobei möglicherweise auch Viren eine Rolle bei der Strukturierung der Zusammensetzung dieser Mitglieder spielen, was einen der ersten Einblicke in eine vielschichtige Meerestiersymbiose bietet. Das Gammaproteobakterium Endozoicomonas hat sich als zentrales Mitglied des Korallenmikrobioms herauskristallisiert und ist in seiner Lebensweise flexibel. Angesichts der jüngsten Massenbleiche in den Riffen werden Korallen wahrscheinlich weiterhin ein nützliches und beliebtes System für die Erforschung von Symbiosen und Dysbiosen sein. ⓘ
Schwämme sind weit verbreitete Mitglieder der vielfältigen benthischen Lebensräume des Ozeans. Ihre Häufigkeit und ihre Fähigkeit, große Mengen an Meerwasser zu filtern, haben zu der Erkenntnis geführt, dass diese Organismen eine entscheidende Rolle bei der Beeinflussung benthischer und pelagischer Prozesse im Ozean spielen. Sie gehören zu den ältesten Tierstämmen und haben einen relativ einfachen Körperplan, der häufig mit Bakterien, Archaeen, Algenprotisten, Pilzen und Viren vergesellschaftet ist. Das Mikrobiom von Schwämmen setzt sich aus Spezialisten und Generalisten zusammen, und die Komplexität ihres Mikrobioms scheint von der Phylogenie des Wirts geprägt zu sein. Studien haben gezeigt, dass das Schwamm-Mikrobiom zum Stickstoffkreislauf in den Ozeanen beiträgt, insbesondere durch die Oxidation von Ammoniak durch Archaeen und Bakterien. Kürzlich wurde gezeigt, dass mikrobielle Symbionten tropischer Schwämme Polyphosphatgranulat produzieren und speichern, wodurch der Wirt möglicherweise in der Lage ist, Zeiten der Phosphatverarmung in oligotrophen Meeresumgebungen zu überleben. Das Mikrobiom einiger Schwammarten scheint sich als Reaktion auf veränderte Umweltbedingungen wie Temperatur und Versauerung der Ozeane sowie auf synergistische Einflüsse in seiner Gemeinschaftsstruktur zu verändern. ⓘ
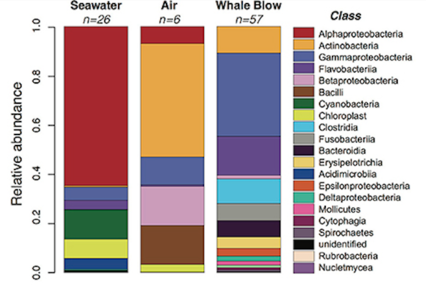
Entnahme einer Blasprobe eines Blauwals mit einer Hubschrauberdrohne
Relative Häufigkeit von Bakterienklassen aus Blas-, Luft- und Meerwasserproben von Walen. ⓘ
Das Mikrobiom von Walen kann schwer zu beurteilen sein, da der Zugang zu mikrobiellen Proben schwierig ist. Zum Beispiel sind viele Walarten selten und tauchen tief. Es gibt verschiedene Techniken zur Entnahme von Proben des Darmmikrobioms von Walen. Die gebräuchlichste ist die Entnahme von Kotproben aus der Umgebung und die Entnahme einer Sonde aus der Mitte, die nicht kontaminiert ist. Die Haut ist eine Barriere, die Meeressäuger vor der Außenwelt schützt. Das epidermale Mikrobiom auf der Haut ist ein Indikator dafür, wie gesund das Tier ist, und ist auch ein ökologischer Indikator für den Zustand der Umgebung. Wenn man weiß, wie das Mikrobiom der Haut von Meeressäugern unter typischen Bedingungen aussieht, kann man verstehen, wie sich diese Gemeinschaften von den freien mikrobiellen Gemeinschaften im Meer unterscheiden. Wale und Delfine sind gefährdet, weil sie zahlreichen Stressfaktoren ausgesetzt sind, die sie anfälliger für verschiedene Krankheiten machen. Sie sind sehr anfällig für Infektionen der Atemwege, aber es ist nur wenig über ihr Atemwegsmikrobiom bekannt. Die Entnahme von Proben des ausgeatmeten Atems oder "Blows" von Walen kann eine Einschätzung ihres Gesundheitszustands liefern. Die Ausatemluft besteht aus einer Mischung von Mikroorganismen und organischem Material, einschließlich Lipiden, Proteinen und Zelltrümmern, die von den Auskleidungen der Atemwege stammen und die, wenn sie in die relativ kühlere Außenluft abgegeben werden, kondensieren und eine sichtbare Dampfmasse bilden, die aufgefangen werden kann. Es gibt verschiedene Methoden zur Sammlung von Ausatemluftproben, eine der neuesten ist der Einsatz von Drohnen aus der Luft. Diese Methode bietet eine sicherere, leisere und weniger invasive Alternative und ist oft eine kostengünstige Option für die Überwachung von Fauna und Flora. Die Luftproben werden ins Labor gebracht, wo die Mikrobiota der Atemwege amplifiziert und sequenziert werden. Der Einsatz von Drohnen aus der Luft war bei großen Walen erfolgreicher, da diese langsame Schwimmgeschwindigkeiten und größere Atemzüge haben. ⓘ
Terrestrisch
Pflanze
Mikrobiome im pflanzlichen Ökosystem ⓘ
Das pflanzliche Mikrobiom spielt eine Rolle für die Pflanzengesundheit und die Lebensmittelproduktion und hat in den letzten Jahren große Aufmerksamkeit erregt. Pflanzen leben in Verbindung mit verschiedenen mikrobiellen Konsortien. Diese Mikroben, die als Mikrobiota der Pflanze bezeichnet werden, leben sowohl innerhalb (Endosphäre) als auch außerhalb (Episphäre) des Pflanzengewebes und spielen eine wichtige Rolle in der Ökologie und Physiologie der Pflanzen. "Man geht davon aus, dass das pflanzliche Kernmikrobiom mikrobielle Taxa umfasst, die für die Fitness der Pflanze wichtig sind und sich durch evolutionäre Mechanismen der Selektion und Anreicherung mikrobieller Taxa, die für die Fitness des pflanzlichen Holobionten wesentliche Funktionsgene enthalten, herausgebildet haben." ⓘ
Das Mikrobiom von Pflanzen wird sowohl durch Faktoren beeinflusst, die mit der Pflanze selbst zusammenhängen, wie Genotyp, Organ, Art und Gesundheitszustand, als auch durch Faktoren, die mit der Umwelt der Pflanze zusammenhängen, wie Bewirtschaftung, Landnutzung und Klima. In einigen Studien wurde berichtet, dass der Gesundheitszustand einer Pflanze durch ihr Mikrobiom widergespiegelt wird oder mit diesem in Verbindung steht. ⓘ
Pflanzen und pflanzenassoziierte Mikrobiota besiedeln verschiedene Nischen auf und im Pflanzengewebe. Alle oberirdischen Pflanzenteile zusammen, die so genannte Phyllosphäre, sind ein sich ständig verändernder Lebensraum, der durch ultraviolette Strahlung (UV) und wechselnde klimatische Bedingungen beeinflusst wird. Sie besteht hauptsächlich aus Blättern. Die unterirdischen Pflanzenteile, hauptsächlich die Wurzeln, werden im Allgemeinen von den Bodeneigenschaften beeinflusst. Schädliche Interaktionen beeinträchtigen das Pflanzenwachstum durch die pathogenen Aktivitäten einiger Mitglieder der Mikrobiota. Andererseits fördern nützliche mikrobielle Interaktionen das Pflanzenwachstum. ⓘ
Auch Pflanzen besitzen ein Mikrobiom, werden also von Kleinstlebewesen besiedelt. Diese Mikroorganismen – etwa zu finden in den Pflanzenwurzeln oder Pflanzensamen – haben Einfluss auf Entwicklung, Wachstum und Gesundheit der Pflanzen. Seit langem bekannt ist die Rolle von Knöllchenbakterien, die eine Symbiose mit Hülsenfrüchtlern eingehen, indem sie die Pflanze mit Stickstoff versorgen und von ihr dafür Zucker erhalten. Die Erforschung und die Optimierung des Mikrobioms von Nutzpflanzen sollen langfristig den Einsatz von Düngern und Pflanzenschutzmitteln reduzieren und Ertragssteigerungen bewirken. Das Mikrobiom von Lebensmitteln und Nutzpflanzen wirkt sich auch auf die Gesundheit und das Mikrobiom von Menschen und Tieren aus. Hygienemaßnahmen wie Desinfektion und Sterilisationsverfahren zerstören oder verändern das Mikrobiom, manche Mikroorganismen gelten dadurch als ausgerottet. Daraus können sich Änderungen im Mikrobiom übergeordneter Lebewesen ergeben, wie beispielsweise beim Menschen, wo das vermehrte Auftreten von Zivilisationserkrankungen wie Allergieneigung, Asthma oder Reizdarm unter anderem darauf zurückgeführt wird. ⓘ
Tier
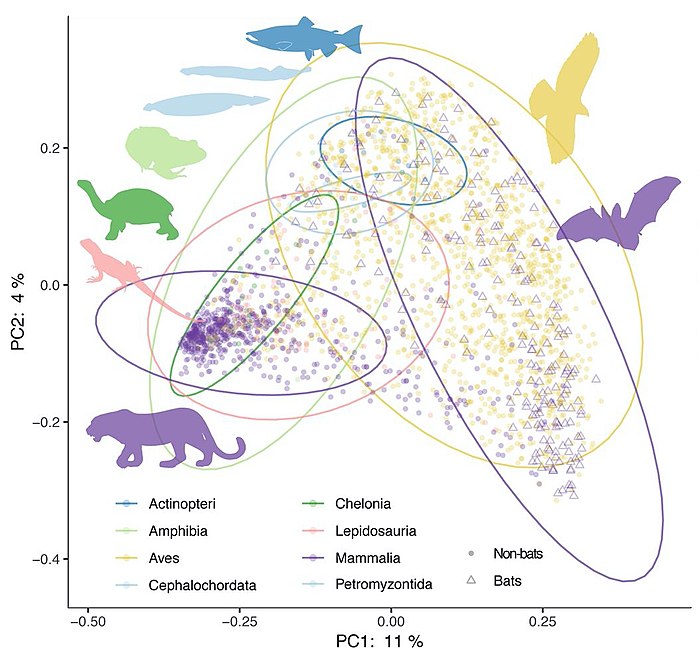
Hauptkoordinatenanalyse von Daten aus dem Darmmikrobiom von Tieren ⓘ
Das Darmmikrobiom von Säugetieren hat sich als wichtiger Regulator der Wirtsphysiologie erwiesen, und die Koevolution zwischen Wirts- und Mikrobenlinien hat eine Schlüsselrolle bei der Anpassung der Säugetiere an ihre vielfältigen Lebensweisen gespielt. Die Ernährung, insbesondere die Pflanzenfresserei, ist ein wichtiges Korrelat der mikrobiellen Vielfalt bei Säugetieren. Die meisten Säugetier-Mikrobiome sind auch stark mit der Phylogenie des Wirts korreliert, trotz tief greifender Veränderungen in der Ernährung. Dies deutet darauf hin, dass Wirtsfaktoren, die sich über die Wirtsphylogenie hinweg verändern, wie die Darmphysiologie, eine wichtige Rolle bei der Strukturierung der Darmmikrobiome von Säugetieren spielen. Es wird sogar spekuliert, dass sich das adaptive Immunsystem der Wirbeltiere als ein solcher Faktor zur selektiven Aufrechterhaltung der symbiotischen Homöostase entwickelt hat. ⓘ
Die Bedeutung von Faktoren, die mit der Phylogenie zusammenhängen, für die Vielfalt der Mikrobiome von Wirbeltieren im Allgemeinen ist noch wenig bekannt. Phylosymbiose, d. h. die Beobachtung, dass näher verwandte Wirtsarten ähnlichere Mikrobiome haben, wurde bei einer Reihe von Taxa beschrieben, die keine Säugetiere sind. Andere Analysen haben erhebliche Unterschiede bei phylosymbiotischen Signalen zwischen Säugetier-Taxa festgestellt, manchmal mit widersprüchlichen Ergebnissen. Das Vorhandensein einer robusten phylosymbiotischen Korrelation bedeutet, dass Wirtsfaktoren den mikrobiellen Aufbau steuern. Selbst wenn die spezifischen Mechanismen nicht bekannt sind, könnte sich die Variation der Stärke oder des Vorhandenseins eines messbaren phylosymbiotischen Signals über die Wirtsphylogenie hinweg als nützlich erweisen, um solche Mechanismen durch vergleichende Studien zu identifizieren. Bis 2020 haben sich die meisten Studien jedoch jeweils nur auf einige wenige Taxa konzentriert, und unterschiedliche Methoden sowohl für die Untersuchung des Mikrobioms als auch für die Messung der Phylosymbiose und der Wirtsspezifität (oder der Beschränkung der Mikroben auf bestimmte Wirtsstämme) haben Verallgemeinerungen erschwert. ⓘ
Ohne einen breiteren evolutionären Kontext ist es unklar, wie universell konserviert die Muster der Phylosymbiose zwischen Wirt und Mikrobe tatsächlich sind. Es gibt immer mehr Hinweise darauf, dass die bei Säugetieren festgestellten starken Muster bei Wirbeltieren eher die Ausnahme als die Regel sind. In Meta-Analysen von Fischen und Vögeln konnten keine so starken Korrelationen zu Ernährung und Phylogenie festgestellt werden wie bei Säugetieren. Eine kürzlich durchgeführte Analyse von Proben von mehr als 100 Wirbeltierarten ergab ebenfalls, dass die Stärke der phylogenetischen Korrelation bei Säugetieren viel höher ist als bei Vögeln, Reptilien, Amphibien oder Fischen. Bei Nicht-Wirbeltieren wird zunehmend erkannt, dass sich grundlegende Aspekte der Beziehung des Wirts zu seiner symbiotischen Gemeinschaft von Taxa zu Taxa drastisch unterscheiden können: Viele Insekten sind bei der Versorgung mit wichtigen Stoffwechselprodukten vollständig von Mikroben abhängig, während es bei anderen anscheinend keine ansässigen Darmmikroben gibt. ⓘ
Mensch
Das menschliche Mikrobiom ist die Gesamtheit aller Mikroorganismen, die sich auf oder in menschlichen Geweben und Bioflüssigkeiten befinden, zusammen mit den entsprechenden anatomischen Stellen, an denen sie sich aufhalten, einschließlich der Haut, der Brustdrüsen, der Samenflüssigkeit, der Gebärmutter, der Eierstockfollikel, der Lunge, des Speichels, der Mundschleimhaut, der Bindehaut, der Gallenwege und des Gastrointestinaltrakts. Zu den Arten der menschlichen Mikrobiota gehören Bakterien, Archaeen, Pilze, Protisten und Viren. Obwohl auch Mikrotiere auf dem menschlichen Körper leben können, werden sie in der Regel von dieser Definition ausgeschlossen. Im Zusammenhang mit der Genomik wird der Begriff menschliches Mikrobiom manchmal verwendet, um sich auf die kollektiven Genome der ansässigen Mikroorganismen zu beziehen; der Begriff menschliches Metagenom hat die gleiche Bedeutung. ⓘ
Der Mensch wird von vielen Mikroorganismen besiedelt, wobei die Größenordnung der nicht-menschlichen Zellen in etwa derjenigen der menschlichen Zellen entspricht. Einige Mikroorganismen, die den Menschen besiedeln, sind Kommensalen, d. h. sie koexistieren, ohne dem Menschen zu schaden oder zu nützen; andere haben eine mutualistische Beziehung zu ihren menschlichen Wirten. Umgekehrt können einige nicht-pathogene Mikroorganismen den menschlichen Wirt durch die von ihnen produzierten Metaboliten schädigen, wie z. B. Trimethylamin, das der menschliche Körper durch FMO3-vermittelte Oxidation in Trimethylamin-N-oxid umwandelt. Bestimmte Mikroorganismen erfüllen Aufgaben, von denen bekannt ist, dass sie für den menschlichen Wirt nützlich sind, aber die Rolle der meisten von ihnen ist nicht gut bekannt. Diejenigen, von denen man erwartet, dass sie vorhanden sind, und die unter normalen Umständen keine Krankheiten verursachen, werden manchmal als normale Flora oder normale Mikrobiota bezeichnet. ⓘ
Das Human Microbiome Project (HMP) hat es sich zur Aufgabe gemacht, das Genom der menschlichen Mikrobiota zu sequenzieren, wobei der Schwerpunkt auf der Mikrobiota liegt, die normalerweise Haut, Mund, Nase, Verdauungstrakt und Vagina bewohnt. Mit der Veröffentlichung der ersten Ergebnisse im Jahr 2012 wurde ein Meilenstein erreicht. ⓘ
Das Mikrobiom des Menschen ist Teil intensiver Forschung und noch nicht umfassend verstanden. Die Hochdurchsatz-Gensequenzierung ermöglicht es, komplexe ökologische Studien durchzuführen. Herausforderungen in der durch den technischen Fortschritt geprägten Forschung ist die Datenauswertung und deren Interpretation. ⓘ
Mittlerweile werden auch separate Mikrobiome betrachtet, die jeweils nur einen Teil des menschlichen Körpers umfassen (Mundraum, Hautoberfläche). Beispielsweise wurde Anfang 2008 das Mund-Mikrobiom vom National Institute of Dental and Craniofacial Research (NIDCR) in Zusammenarbeit mit Forschern aus anderen Ländern in Angriff genommen. Es umfasst bereits 600 Mikroorganismen. Die Forscher ordnen diese Mikroorganismen allmählich in einem Stammbaum an, was durch die Sequenzierung der 16S-rRNA ermöglicht wurde. Damit erhoffen sie sich ein besseres Verständnis ihrer Bedeutung bei der Entstehung der Karies oder verschiedenen Störungen der Verdauung. ⓘ
Bewertung
Die derzeit verfügbaren Methoden zur Untersuchung von Mikrobiomen, die so genannten Multi-omics, reichen von der Isolierung im Hochdurchsatzverfahren (Culturomics) und der Visualisierung (Mikroskopie) bis hin zur gezielten Erfassung der taxonomischen Zusammensetzung (Metabarcoding) oder der Untersuchung des metabolischen Potenzials (Metabarcoding funktioneller Gene, Metagenomics), um die mikrobielle Aktivität zu analysieren (Metatranscriptomics, Metaproteomics, Metabolomics). Auf der Grundlage von Metagenomdaten können mikrobielle Genome rekonstruiert werden. Während die ersten metagenom-assemblierten Genome aus Umweltproben rekonstruiert wurden, wurden in den letzten Jahren mehrere Tausend bakterielle Genome entnommen, ohne die dahinter stehenden Organismen zu kultivieren. Zum Beispiel wurden 2019 154.723 mikrobielle Genome des globalen menschlichen Mikrobioms aus 9.428 Metagenomen rekonstruiert. ⓘ
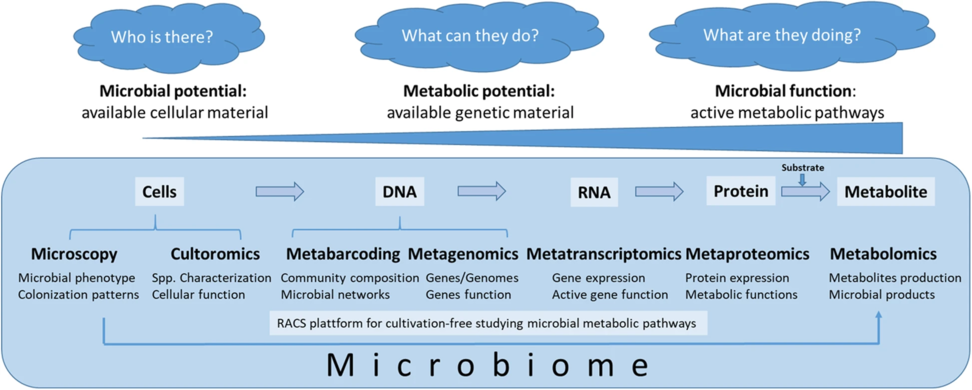
- Methoden zur Bewertung der mikrobiellen Funktionsweise
Komplexe Mikrobiomstudien decken verschiedene Bereiche ab, beginnend mit der Ebene der vollständigen mikrobiellen Zellen (Mikroskopie, Kulturomik), gefolgt von der DNA (Einzelzellgenomik, Metabarcoding, Metagenomik), RNA (Metatranskriptomik), Proteinen (Metaproteomik) und Metaboliten (Metabolomik). In dieser Reihenfolge verlagert sich der Schwerpunkt der Studien vom mikrobiellen Potenzial (Lernen über die in einem bestimmten Lebensraum vorhandene Mikrobiota) über das metabolische Potenzial (Entschlüsselung des vorhandenen genetischen Materials) hin zur mikrobiellen Funktionsweise (z. B. die Entdeckung der aktiven Stoffwechselwege). ⓘ
Die computergestützte Modellierung von Mikrobiomen wurde eingesetzt, um experimentelle Methoden zur Untersuchung der mikrobiellen Funktion zu ergänzen, indem Multi-Omic-Daten zur Vorhersage komplexer Dynamiken zwischen verschiedenen Arten und Wirtsarten verwendet werden. Eine beliebte In-silico-Methode ist die Kombination von Stoffwechselnetzwerkmodellen der in einer Gemeinschaft vorhandenen mikrobiellen Taxa und die Anwendung einer mathematischen Modellierungsstrategie wie der Flussbilanzanalyse zur Vorhersage der Stoffwechselfunktion der mikrobiellen Gemeinschaft auf Taxon- und Gemeinschaftsebene. ⓘ
Bis zum Jahr 2020 ist das Verständnis aufgrund der fehlenden Verbindungen zwischen der massiven Verfügbarkeit von Mikrobiom-DNA-Sequenzdaten einerseits und der begrenzten Verfügbarkeit von mikrobiellen Isolaten, die zur Bestätigung metagenomischer Vorhersagen der Genfunktion benötigt werden, andererseits begrenzt. Metagenomdaten bieten eine Spielwiese für neue Vorhersagen, aber es werden noch viel mehr Daten benötigt, um die Verbindungen zwischen Sequenz und rigorosen Funktionsvorhersagen zu stärken. Dies wird deutlich, wenn man bedenkt, dass der Ersatz eines einzigen Aminosäurerests durch einen anderen zu einer radikalen Funktionsänderung führen kann, was eine falsche funktionelle Zuordnung zu einer bestimmten Gensequenz zur Folge hat. Darüber hinaus ist die Kultivierung neuer Stämme erforderlich, um den großen Anteil unbekannter Sequenzen aus Metagenomics-Analysen zu identifizieren, der bei schlecht untersuchten Ökosystemen mehr als 70 % betragen kann. Je nach angewandter Methode haben selbst in gut untersuchten Mikrobiomen 40-70 % der annotierten Gene in vollständig sequenzierten mikrobiellen Genomen keine bekannte oder vorhergesagte Funktion. Bis 2019 war für 85 der damals 118 Phyla keine einzige Art beschrieben worden, was eine Herausforderung für das Verständnis der funktionellen Vielfalt der Prokaryoten darstellt. ⓘ
Die Zahl der prokaryotischen Phyla kann Hunderte erreichen, und die Archaeen gehören zu den am wenigsten erforschten. Die wachsende Kluft zwischen der Vielfalt der Bakterien und Archaeen in Reinkultur und der mit molekularen Methoden nachgewiesenen Vielfalt hat zu dem Vorschlag geführt, eine formale Nomenklatur für noch nicht kultivierte Taxa zu erstellen, die in erster Linie auf Sequenzinformationen beruht. Diesem Vorschlag zufolge würde das Konzept der Candidatus-Arten auf die Gruppen eng verwandter Genomsequenzen ausgedehnt, und ihre Namen würden nach den etablierten Regeln der bakteriellen Nomenklatur veröffentlicht. ⓘ
Jedes Mikrobiom-System eignet sich für unterschiedliche Fragestellungen, je nachdem, ob die Mikroben kultiviert werden können, ob Mikroben und Wirt (sofern relevant) genetisch vertretbar sind, ob das System in einer Laborumgebung aufrechterhalten werden kann und ob Wirt/Umwelt keimfrei gemacht werden können. ⓘ
- Zugrundeliegende Komplexität
Kompromisse zwischen experimentellen Fragen und der Komplexität von Mikrobiom-Systemen
(A) Paarweise Interaktionen zwischen den Bodenbakterien Bacillus subtilis und Streptomyces spp. eignen sich gut für die Charakterisierung der Funktionen von Sekundärmetaboliten in mikrobiellen Interaktionen.
(B) Die Symbiose zwischen Bobtail-Tintenfisch und dem Meeresbakterium Aliivibrio fischeri ist von grundlegender Bedeutung für das Verständnis von Wirts- und mikrobiellen Faktoren, die die Besiedlung beeinflussen.
(C) Die Verwendung von gnotobiotischen Mäusen ist entscheidend für die Herstellung von Verbindungen zwischen der Ernährung des Wirts und den Auswirkungen auf spezifische mikrobielle Taxa in einer Gemeinschaft. ⓘ
Siehe auch
- Holobiont
- Mikrobiomtransfer ⓘ
Literatur
- A. Shade, J. Handelsman: Beyond the Venn diagram: the hunt for a core microbiome. In: Environmental microbiology. [elektronische Veröffentlichung vor dem Druck] Oktober 2011. doi:10.1111/j.1462-2920.2011.02585.x, PMID 22004523 (Short review).
- Mikroben-Inventur: Das Gewimmel im Körper. Spiegel-Artikel vom 14. Juni 2012 Auf: spiegel.de; zuletzt abgerufen am 19. September 2015.
- Felix Goeser: Mikrobiomforschung: Wie körpereigene Keime als „Superorgan“ agieren. In: Dtsches Ärzteblatt. 2012, Band 109, Nr. 25, A-1317.
- I. B. Autenrieth: Die Ökologie der humanen Darmflora: physiologische und pathophysiologische Aspekte. In: Journal für Ernährungsmedizin. 2000, Band 2, Nr. 4, S. 6–9 (Volltext als PDF-Datei).
- Andreas Stallmach, Maria J G T Vehreschild (Hg): Mikrobiom: Wissensstand und Perspektiven. De Gruyter, Berlin 2016, ISBN 978-3-11-045249-5. ⓘ
Populärwissenschaftlich
- Bernhard Kegel: Die Herrscher der Welt : wie Mikroben unser Leben bestimmen. DuMont, Köln 2015, ISBN 978-3-8321-9773-5.
- Sebastian Jutzi: Der bewohnte Mensch: Darm, Haut, Psyche; besser leben mit Mikroben (= Heyne. Band 60307). Heyne, München 2014, ISBN 978-3-453-60307-3.
- Hanno Charisius & Richard Friebe: Bund fürs Leben. Warum Bakterien unsere Freunde sind. Hanser, München 2014, ISBN 978-3-446-43879-8. ⓘ
Dokumentarfilme
- Unser Bauch - Die wunderbare Welt des Mikrobioms Regie: Thierry de Lestrade und Sylvie Gilman, ARTE F, 2019. ⓘ