Salmonellen
| Salmonellen ⓘ | |
|---|---|
| |
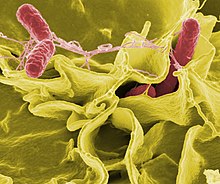
| Farbverstärkte rasterelektronenmikroskopische Aufnahme von Salmonella Typhimurium (rot), die in kultivierte menschliche Zellen eindringt | |
| Wissenschaftliche Klassifizierung | |
| Bereich: | Bakterien |
| Stamm: | Pseudomonadota |
| Klasse: | Gammaproteobakterien |
| Ordnung: | Enterobacterales |
| Familie: | Enterobacteriaceae |
| Gattung: | Salmonellen Lignières, 1900 |
| Arten und Unterarten | |
| |
Salmonella ist eine Gattung stäbchenförmiger (Bazillen) gramnegativer Bakterien aus der Familie der Enterobacteriaceae. Die beiden Arten von Salmonella sind Salmonella enterica und Salmonella bongori. S. enterica ist die Typusart und wird in sechs Unterarten unterteilt, die über 2 600 Serotypen umfassen. Salmonella wurde nach Daniel Elmer Salmon (1850-1914), einem amerikanischen Veterinärmediziner, benannt. ⓘ
Salmonella-Arten sind nicht sporenbildende, überwiegend bewegliche Enterobakterien mit Zelldurchmessern zwischen etwa 0,7 und 1,5 μm, Längen von 2 bis 5 μm und peritrichen Geißeln (rund um den Zellkörper, wodurch sie sich bewegen können). Sie sind chemotroph, d. h. sie beziehen ihre Energie aus Oxidations- und Reduktionsreaktionen, wobei sie organische Quellen nutzen. Sie sind auch fakultative Anaerobier, die in der Lage sind, ATP mit Sauerstoff zu erzeugen (aerob"), wenn dieser verfügbar ist, oder andere Elektronenakzeptoren oder Fermentation zu nutzen (anaerob"), wenn kein Sauerstoff verfügbar ist. ⓘ
Salmonella-Arten sind intrazelluläre Krankheitserreger, von denen bestimmte Serotypen Krankheiten verursachen. Die meisten Infektionen sind auf die Aufnahme von Lebensmitteln zurückzuführen, die durch tierische Fäkalien oder menschliche Fäkalien verunreinigt sind, z. B. durch einen Mitarbeiter in einem kommerziellen Restaurant. Die Salmonella-Serotypen können in zwei Hauptgruppen eingeteilt werden - in typhoide und nicht-typhoide. Nicht-typhoide Serotypen sind zoonotisch und können von Tier zu Mensch und von Mensch zu Mensch übertragen werden. Sie befallen in der Regel nur den Magen-Darm-Trakt und verursachen eine Salmonellose, deren Symptome ohne Antibiotika behandelt werden können. In Afrika südlich der Sahara können nicht-typhoide Salmonellen jedoch invasiv sein und Paratyphus verursachen, das eine sofortige Behandlung mit Antibiotika erfordert. Typhoide Serotypen können nur von Mensch zu Mensch übertragen werden und können lebensmittelbedingte Infektionen, Typhus und paratyphoides Fieber verursachen. Typhus wird durch Salmonellen verursacht, die in die Blutbahn eindringen (typhoide Form) oder sich im ganzen Körper ausbreiten, Organe befallen und Endotoxine absondern (septische Form). Dies kann zu einem lebensbedrohlichen hypovolämischen Schock und septischen Schock führen und erfordert eine intensivmedizinische Behandlung einschließlich Antibiotika. ⓘ
Taxonomie
Die Gattung Salmonella ist Teil der Familie der Enterobacteriaceae. Ihre Taxonomie wurde überarbeitet und kann zu Verwirrung führen. Die Gattung umfasst zwei Arten, S. bongori und S. enterica, von denen letztere in sechs Unterarten unterteilt wird: S. e. enterica, S. e. salamae, S. e. arizonae, S. e. diarizonae, S. e. houtenae, und S. e. indica. Die taxonomische Gruppe umfasst mehr als 2500 Serotypen (auch Serovare), die auf der Grundlage der somatischen O- (Lipopolysaccharid) und flagellaren H-Antigene (Kauffman-White-Klassifikation) definiert werden. Der vollständige Name eines Serotyps lautet z. B. Salmonella enterica subsp. enterica Serotyp Typhimurium, kann aber auch mit Salmonella Typhimurium abgekürzt werden. Eine weitere Differenzierung der Stämme zur Unterstützung klinischer und epidemiologischer Untersuchungen kann durch Antibiotika-Empfindlichkeitstests und andere molekularbiologische Verfahren wie die Pulsfeld-Gelelektrophorese, die Multilocus-Sequenztypisierung und zunehmend auch die Sequenzierung des gesamten Genoms erfolgen. In der Vergangenheit wurden Salmonellen klinisch in invasive (typhoide) und nicht-invasive (nicht-typhoide) Salmonellen eingeteilt, basierend auf der Wirtspräferenz und den Krankheitsmanifestationen beim Menschen. ⓘ
Geschichte
Salmonellen wurden erstmals 1880 von Karl Eberth in den Peyerschen Flecken und der Milz von Typhuspatienten nachgewiesen. Vier Jahre später gelang es Georg Theodor Gaffky, den Erreger in Reinkultur zu züchten. Ein Jahr später entdeckte der Mediziner Theobald Smith den Erreger, der später als Salmonella enterica (var. Choleraesuis) bekannt werden sollte. Zu dieser Zeit arbeitete Smith als Forschungslaborant in der Veterinärabteilung des Landwirtschaftsministeriums der Vereinigten Staaten. Die Abteilung stand unter der Leitung von Daniel Elmer Salmon, einem Veterinärpathologen. Ursprünglich wurde Salmonella Choleraesuis für den Erreger der Schweinecholera gehalten, weshalb Salmon und Smith ihn "Hog-cholerabacillus" nannten. Der Name Salmonella wurde erst 1900 verwendet, als Joseph Leon Lignières vorschlug, den von Salmons Gruppe entdeckten Erreger ihm zu Ehren Salmonella zu nennen. ⓘ
Serotypisierung
Bei der Serotypisierung werden Zellen mit Antikörpern gegen ein bestimmtes Antigen gemischt. Sie kann Aufschluss über das Risiko geben. Eine Studie aus dem Jahr 2014 zeigte, dass S. reading in Proben von jungen Puten sehr häufig vorkommt, aber nicht wesentlich zur Salmonellose beim Menschen beiträgt. Die Serotypisierung kann bei der Identifizierung der Kontaminationsquelle helfen, indem die Serotypen beim Menschen mit den Serotypen der vermuteten Infektionsquelle verglichen werden. Anhand der bekannten Antibiotikaresistenz des Serotyps kann eine geeignete prophylaktische Behandlung ermittelt werden. ⓘ
Nachweis, Kultur und Wachstumsbedingungen

Die meisten Unterarten von Salmonella produzieren Schwefelwasserstoff, der sich leicht nachweisen lässt, indem man sie auf eisen(II)-sulfathaltigen Nährböden kultiviert, wie sie im Dreifachzucker-Eisentest verwendet werden. Die meisten Isolate existieren in zwei Phasen, einer beweglichen und einer unbeweglichen Phase. Kulturen, die in der Primärkultur unbeweglich sind, können mit Hilfe eines Craigie-Röhrchens oder einer Grabenplatte in die bewegliche Phase überführt werden. RVS-Bouillon kann zur Anreicherung von Salmonella-Arten für den Nachweis in einer klinischen Probe verwendet werden. ⓘ
Salmonellen können auch mittels Multiplex- oder Echtzeit-Polymerase-Kettenreaktion (qPCR) aus extrahierter Salmonellen-DNA nachgewiesen und subtypisiert werden. ⓘ
Mathematische Modelle der Wachstumskinetik von Salmonellen wurden für Hühner, Schweine, Tomaten und Melonen entwickelt. Salmonellen vermehren sich asexuell mit einem Zellteilungsintervall von 40 Minuten. ⓘ
Salmonella-Arten leben überwiegend wirtsassoziiert, aber es wurde festgestellt, dass die Bakterien in der Lage sind, nach einer Kontamination wochenlang in einem Badezimmer zu überleben, und häufig aus Wasserquellen isoliert werden, die als Bakterienreservoir dienen und die Übertragung zwischen Wirten erleichtern können. Salmonellen sind berüchtigt für ihre Fähigkeit, die Austrocknung zu überleben, und können jahrelang in trockenen Umgebungen und Lebensmitteln überleben. ⓘ
Die Bakterien werden durch Einfrieren nicht zerstört, aber UV-Licht und Hitze beschleunigen ihre Zerstörung. Sie gehen zugrunde, wenn sie 90 Minuten lang auf 55 °C (131 °F) oder 12 Minuten lang auf 60 °C (140 °F) erhitzt werden. Wenn sie jedoch in fettreichen, flüssigen Substanzen wie Erdnussbutter geimpft werden, werden sie hitzeresistent und können 30 Minuten lang bis zu 90 °C (194 °F) überleben. Zum Schutz vor einer Salmonelleninfektion wird empfohlen, Lebensmittel auf eine Innentemperatur von 75 °C zu erhitzen. ⓘ
Salmonella-Arten können im Verdauungstrakt von Menschen und Tieren, insbesondere von Reptilien, vorkommen. Salmonellen auf der Haut von Reptilien oder Amphibien können auf Menschen übertragen werden, die mit diesen Tieren umgehen. Auch Lebensmittel und Wasser können mit den Bakterien kontaminiert werden, wenn sie mit den Ausscheidungen infizierter Menschen oder Tiere in Berührung kommen. ⓘ
Nomenklatur
Ursprünglich wurden die einzelnen Salmonella-"Arten" nach klinischen Gesichtspunkten benannt, z. B. Salmonella typhi-murium (Maus-Typhus), S. cholerae-suis. Nachdem erkannt wurde, dass es für viele Arten keine Wirtsspezifität gibt, wurden neue Stämme nach dem Ort benannt, an dem der neue Stamm isoliert wurde. Später führten molekulare Erkenntnisse zu der Hypothese, dass Salmonella aus nur einer Art, S. enterica, besteht, und die Serotypen wurden in sechs Gruppen eingeteilt, von denen zwei medizinisch relevant sind. Da diese nun formalisierte Nomenklatur nicht mit dem traditionellen Sprachgebrauch der Mikrobiologen und Infektiologen übereinstimmt, ist die traditionelle Nomenklatur immer noch üblich. Derzeit sind die beiden anerkannten Arten S. enterica und S. bongori. Im Jahr 2005 wurde eine dritte Art, Salmonella subterranea, vorgeschlagen, aber laut Weltgesundheitsorganisation gehört das gemeldete Bakterium nicht zur Gattung Salmonella. Die sechs wichtigsten anerkannten Unterarten sind: enterica (Serotyp I), salamae (Serotyp II), arizonae (IIIa), diarizonae (IIIb), houtenae (IV) und indica (VI). Der frühere Serotyp V war bongori, der jetzt als eigene Art betrachtet wird. ⓘ
Der Serotyp oder Serovar ist eine Klassifizierung von Salmonella in Unterarten auf der Grundlage von Antigenen, die der Organismus aufweist. Er basiert auf dem Kauffman-White-Klassifizierungsschema, das die serologischen Varietäten voneinander unterscheidet. Die Serotypen werden in der Regel nach der Gattung und der Art in Unterartengruppen eingeteilt, wobei die Serotypen/Serovare groß, aber nicht kursiv geschrieben werden: Ein Beispiel ist Salmonella enterica serovar Typhimurium. Zu den moderneren Ansätzen für die Typisierung und Subtypisierung von Salmonella gehören DNA-basierte Methoden wie die gepulste Feldelektrophorese, die VNTR-Mehrfachloci-Analyse, die Multilocus-Sequenztypisierung und Multiplex-PCR-basierte Methoden. ⓘ
Pathogenität
Salmonella-Arten sind fakultativ intrazelluläre Pathogene. Salmonellen können in verschiedene Zelltypen eindringen, darunter Epithelzellen, M-Zellen, Makrophagen und dendritische Zellen. Als fakultativ anaerober Organismus nutzt Salmonella in aerober Umgebung (d. h. wenn Sauerstoff verfügbar ist) Sauerstoff zur ATP-Herstellung. In anaerober Umgebung (d. h. wenn kein Sauerstoff verfügbar ist) produziert Salmonella ATP durch Fermentation, indem es einen oder mehrere der vier weniger effizienten Elektronenakzeptoren als Sauerstoff am Ende der Elektronentransportkette ersetzt: Sulfat, Nitrat, Schwefel oder Fumarat. ⓘ
Die meisten Infektionen sind auf die Aufnahme von Lebensmitteln zurückzuführen, die durch tierische Fäkalien oder menschliche Fäkalien verunreinigt sind, z. B. durch einen Mitarbeiter in einem kommerziellen Restaurant. Die Salmonella-Serotypen können in zwei Hauptgruppen eingeteilt werden - in typhoide und nicht-typhoide. Nicht-typhoide Serotypen sind häufiger anzutreffen und verursachen in der Regel selbstlimitierende Magen-Darm-Erkrankungen. Sie können eine Reihe von Tieren infizieren und sind zoonotisch, d. h. sie können zwischen Menschen und anderen Tieren übertragen werden. Zu den typhoiden Serotypen gehören Salmonella Typhi und Salmonella Paratyphi A, die an den Menschen angepasst sind und bei anderen Tieren nicht vorkommen. ⓘ
Nicht-typhoide Salmonellen
Nicht-invasiv
Eine Infektion mit nicht-typhoiden Salmonella-Serotypen führt in der Regel zu einer Lebensmittelvergiftung. Die Infektion tritt in der Regel auf, wenn eine Person Lebensmittel zu sich nimmt, die eine hohe Konzentration der Bakterien enthalten. Säuglinge und Kleinkinder sind viel anfälliger für eine Infektion, die leicht durch die Aufnahme einer geringen Anzahl von Bakterien erfolgt. Bei Säuglingen ist eine Infektion durch Einatmen von bakterienhaltigem Staub möglich. ⓘ
Die Organismen gelangen über den Verdauungstrakt und müssen in großer Zahl aufgenommen werden, um bei gesunden Erwachsenen eine Krankheit auszulösen. Eine Infektion kann erst beginnen, wenn lebende Salmonellen (und nicht nur die von Salmonellen produzierten Toxine) den Magen-Darm-Trakt erreichen. Ein Teil der Mikroorganismen wird im Magen abgetötet, während die überlebenden Mikroorganismen in den Dünndarm gelangen und sich im Gewebe vermehren. Die Magensäure ist für die Zerstörung der meisten aufgenommenen Bakterien verantwortlich, aber Salmonella hat eine gewisse Toleranz gegenüber sauren Umgebungen entwickelt, die es einer Teilmenge der aufgenommenen Bakterien ermöglicht, zu überleben. Bakterienkolonien können sich auch im Schleim der Speiseröhre festsetzen. Am Ende der Inkubationszeit sind die nahe gelegenen Wirtszellen durch die von den toten Salmonellen freigesetzten Endotoxine vergiftet. Die lokale Reaktion auf die Endotoxine ist Enteritis und gastrointestinale Störung. ⓘ
Es sind etwa 2.000 Serotypen von nicht-typhoiden Salmonellen bekannt, die in den Vereinigten Staaten jedes Jahr für bis zu 1,4 Millionen Erkrankungen verantwortlich sein können. Zu den Personen, bei denen ein Risiko für schwere Erkrankungen besteht, gehören Säuglinge, ältere Menschen, Empfänger von Organtransplantaten und immungeschwächte Personen. ⓘ
Invasiv
Während in den Industrieländern nicht-typhoide Serotypen meist als gastrointestinale Erkrankungen auftreten, können diese Serotypen in Afrika südlich der Sahara ein großes Problem bei Blutstrominfektionen darstellen und sind die am häufigsten isolierten Bakterien aus dem Blut von Menschen, die Fieber haben. Blutstrominfektionen, die durch nicht-typhoide Salmonellen in Afrika verursacht werden, hatten 2012 eine Sterblichkeitsrate von 20-25 %. Die meisten Fälle einer invasiven nicht-typhoiden Salmonelleninfektion (iNTS) werden durch Salmonella enterica Typhimurium oder Salmonella enterica Enteritidis verursacht. Eine neue Form von Salmonella Typhimurium (ST313) tauchte vor 75 Jahren im Südosten des afrikanischen Kontinents auf, gefolgt von einer zweiten Welle, die 18 Jahre später von Zentralafrika ausging. Diese zweite Welle von iNTS hatte ihren Ursprung möglicherweise im Kongobecken und nahm früh ein Gen auf, das sie gegen das Antibiotikum Chloramphenicol resistent machte. Dies führte dazu, dass in sehr armen Gebieten Afrikas teure antimikrobielle Medikamente eingesetzt werden mussten, was die Behandlung erschwerte. Die im Vergleich zu anderen Regionen erhöhte Prävalenz von iNTS in den afrikanischen Ländern südlich der Sahara ist vermutlich auf den großen Anteil der afrikanischen Bevölkerung zurückzuführen, der aufgrund der Belastung durch HIV, Malaria und Unterernährung, insbesondere bei Kindern, ein gewisses Maß an Immunsuppression oder Beeinträchtigung aufweist. Das Erbgut von iNTS entwickelt sich zu einem eher typhusähnlichen Bakterium, das sich effizient im menschlichen Körper ausbreiten kann. Es wird von verschiedenen Symptomen berichtet, darunter Fieber, Hepatosplenomegalie und Atemwegssymptome, wobei gastrointestinale Symptome häufig fehlen. ⓘ
Epidemiologie
Da Salmonelleninfektionen als sporadisch auftreten, werden zwischen 60 % und 80 % der Fälle nicht diagnostiziert. Im März 2010 wurde eine Datenanalyse abgeschlossen, die eine Inzidenzrate von 1140 pro 100.000 Personenjahre ergab. In der gleichen Analyse wurden 93,8 Millionen Fälle von Gastroenteritis auf Salmonelleninfektionen zurückgeführt. Beim 5. Perzentil lag die geschätzte Zahl bei 61,8 Millionen Fällen und beim 95. Perzentil bei 131,6 Millionen Fällen. Die geschätzte Zahl der durch Salmonellen verursachten Todesfälle lag bei etwa 155.000 Todesfällen. Im Jahr 2014 war in Ländern wie Bulgarien und Portugal die Wahrscheinlichkeit einer Salmonelleninfektion bei Kindern unter 4 Jahren 32 bzw. 82 Mal höher. Am anfälligsten für eine Infektion sind Kinder, Schwangere, ältere Menschen und Menschen mit einem schwachen Immunsystem. ⓘ
Zu den Risikofaktoren für Salmonelleninfektionen gehören eine Vielzahl von Lebensmitteln. Fleisch wie Huhn und Schweinefleisch kann kontaminiert sein. Auch verschiedene Gemüsesorten und Sprossen können Salmonellen enthalten. Schließlich können auch eine Reihe von verarbeiteten Lebensmitteln wie Chicken Nuggets und Pasteten diese Bakterien enthalten. ⓘ
Erfolgreiche Präventionsmaßnahmen werden von bestehenden Einrichtungen wie der FDA, dem Landwirtschaftsministerium der Vereinigten Staaten und dem Food Safety and Inspection Service durchgeführt. Alle diese Organisationen schaffen Normen und Inspektionen, um die öffentliche Sicherheit in den USA zu gewährleisten. Die FSIS-Behörde, die mit dem USDA zusammenarbeitet, hat beispielsweise einen Salmonellen-Aktionsplan aufgestellt. Im Februar 2016 wurde der Plan für zwei Jahre aktualisiert. In den Plänen werden ihre Leistungen und Strategien zur Verringerung von Salmonelleninfektionen vorgestellt. Die Centers for Disease Control and Prevention (Zentren für Krankheitskontrolle und -prävention) stellen ebenfalls wertvolle Informationen zur Vorbeugung zur Verfügung, z. B. über den sicheren Umgang mit rohen Lebensmitteln und die richtige Lagerung dieser Produkte. In der Europäischen Union hat die Europäische Behörde für Lebensmittelsicherheit durch Risikomanagement und Risikobewertung Präventionsmaßnahmen geschaffen. Von 2005 bis 2009 verfolgte die EFSA einen Ansatz zur Verringerung der Salmonellenbelastung. Ihr Ansatz umfasste eine Risikobewertung und ein Risikomanagement für Geflügel, was zu einem Rückgang der Infektionsfälle um die Hälfte führte. In Lateinamerika wurde ein von Dr. Sherry Layton entwickelter, oral verabreichter Impfstoff gegen Salmonellen bei Geflügel eingeführt, der eine Kontamination der Vögel mit den Bakterien verhindert. ⓘ
Typhusähnliche Salmonellen
Typhus wird durch Salmonella-Serotypen verursacht, die streng an den Menschen oder höhere Primaten angepasst sind - dazu gehören Salmonella Typhi, Paratyphi A, Paratyphi B und Paratyphi C. Bei der systemischen Form der Krankheit gelangen Salmonellen über das Lymphsystem des Darms in das Blut der Patienten (typhoide Form) und werden in verschiedene Organe (Leber, Milz, Nieren) verschleppt, wo sie sekundäre Herde bilden (septische Form). Die Endotoxine wirken zunächst auf den Gefäß- und Nervenapparat, was zu einer erhöhten Durchlässigkeit und einem verminderten Tonus der Gefäße, einer Störung der Wärmeregulation sowie zu Erbrechen und Durchfall führt. Bei schweren Formen der Erkrankung gehen so viel Flüssigkeit und Elektrolyte verloren, dass der Wasser-Salz-Stoffwechsel gestört wird, das zirkulierende Blutvolumen und der arterielle Druck sinken und ein hypovolämischer Schock entsteht. Auch ein septischer Schock kann sich entwickeln. Ein gemischter Schock (mit Anzeichen sowohl eines hypovolämischen als auch eines septischen Schocks) kommt bei schwerer Salmonellose häufiger vor. Oligurie und Azotämie können sich in schweren Fällen als Folge der Nierenbeteiligung aufgrund von Hypoxie und Toxämie entwickeln. ⓘ
Globale Überwachung
In Deutschland sind lebensmittelbedingte Infektionen meldepflichtig. Von 1990 bis 2016 ist die Zahl der offiziell registrierten Fälle von etwa 200.000 auf etwa 13.000 zurückgegangen. In den Vereinigten Staaten werden jährlich etwa 1.200.000 Fälle von Salmonelleninfektionen geschätzt. Eine Studie der Weltgesundheitsorganisation schätzt, dass im Jahr 2000 21.650.974 Fälle von Typhus auftraten, von denen 216.510 zum Tode führten, sowie 5.412.744 Fälle von Paratyphus. ⓘ
Molekulare Mechanismen der Infektion
Die Infektionsmechanismen unterscheiden sich zwischen typhösen und nichttyphösen Serotypen aufgrund ihrer unterschiedlichen Angriffspunkte im Körper und der unterschiedlichen Symptome, die sie verursachen. Beide Gruppen müssen die von der Darmzellwand gebildete Barriere überwinden, aber sobald sie diese Barriere überwunden haben, wenden sie unterschiedliche Strategien an, um eine Infektion zu verursachen. ⓘ
Wechsel zur Virulenz
Auf dem Weg zu ihrem Zielgewebe im Magen-Darm-Trakt sind Salmonellen der Magensäure, der detergenzienähnlichen Aktivität der Galle im Darm, der abnehmenden Sauerstoffzufuhr, der konkurrierenden normalen Darmflora und schließlich den antimikrobiellen Peptiden auf der Oberfläche der Zellen, die die Darmwand auskleiden, ausgesetzt. All dies sind Stressfaktoren, die Salmonellen wahrnehmen und auf die sie reagieren können. Sie bilden Virulenzfaktoren und regulieren so den Übergang von ihrem normalen Wachstum im Darm zur Virulenz. ⓘ
Der Übergang zur Virulenz ermöglicht den Zugang zu einer Replikationsnische im Wirt (z. B. dem Menschen) und lässt sich in mehrere Phasen unterteilen:
- Annäherung, bei der sie sich entweder über die Darmperistaltik oder durch aktives Schwimmen über die Geißeln zu einer Wirtszelle bewegen, die Schleimbarriere durchdringen und sich in der Nähe des den Darm auskleidenden Epithels ansiedeln,
- Adhäsion, bei der sie mit Hilfe von bakteriellen Adhäsinen und einem Typ-3-Sekretionssystem an einer Wirtszelle haften,
- Invasion, bei der die Salmonellen in die Wirtszelle eindringen (siehe die verschiedenen Mechanismen weiter unten),
- Replikation, bei der sich das Bakterium innerhalb der Wirtszelle vermehren kann,
- Ausbreitung, bei der sich das Bakterium über Zellen im Blut in andere Organe ausbreiten kann (wenn es ihm gelungen ist, die Immunabwehr zu umgehen). Alternativ können die Bakterien in den Darm zurückwandern und die Darmpopulation neu besiedeln.
- Re-Invasion (eine Sekundärinfektion, wenn sie nun an einer systemischen Stelle auftritt) und weitere Vermehrung. ⓘ
Mechanismen des Eindringens
Nicht-typhoide Serotypen dringen bevorzugt durch bakteriell vermittelte Endozytose in die M-Zellen der Darmwand ein, ein Prozess, der mit Darmentzündungen und Diarrhöe einhergeht. Sie sind auch in der Lage, die engen Verbindungen zwischen den Zellen der Darmwand zu unterbrechen, wodurch die Fähigkeit der Zellen beeinträchtigt wird, den Fluss von Ionen, Wasser und Immunzellen in den und aus dem Darm zu stoppen. Es wird angenommen, dass die Kombination aus der durch die bakterienvermittelte Endozytose verursachten Entzündung und der Unterbrechung der tight junctions wesentlich zur Auslösung von Durchfall beiträgt. ⓘ
Salmonellen sind auch in der Lage, die Darmbarriere durch Phagozytose und Trafficking durch CD18-positive Immunzellen zu durchbrechen, was möglicherweise ein Schlüsselmechanismus für die typhusartige Salmonelleninfektion ist. Dieser Mechanismus ist möglicherweise der Schlüssel zur Infektion mit typhoiden Salmonellen. Man geht davon aus, dass dies ein heimlicherer Weg ist, um die Darmbarriere zu überwinden, und könnte daher dazu beitragen, dass für eine Infektion eine geringere Anzahl von typhoiden Salmonellen erforderlich ist als von nicht-typhoiden Salmonellen. Salmonellen sind in der Lage, über Makropinozytose in Makrophagen einzudringen. Typhoide Serotypen können dies nutzen, um sich über das mononukleare Phagozytensystem, ein Netzwerk aus Bindegewebe, das Immunzellen enthält und mit dem Immunsystem assoziiertes Gewebe im ganzen Körper umgibt, im Körper zu verbreiten. ⓘ
Ein Großteil des Erfolgs von Salmonellen bei der Verursachung von Infektionen wird auf zwei Typ-III-Sekretionssysteme (T3SS) zurückgeführt, die zu unterschiedlichen Zeitpunkten während der Infektion exprimiert werden. Das T3SS-1 ermöglicht die Injektion von bakteriellen Effektoren in das Zytosol des Wirts. Diese T3SS-1-Effektoren stimulieren die Bildung von Membrankräuseln, die die Aufnahme von Salmonellen durch nichtphagozytische Zellen ermöglichen. Salmonellen befinden sich außerdem in einem membrangebundenen Kompartiment, das als Salmonella-Containing Vacuole (SCV) bezeichnet wird. Die Ansäuerung der SCV führt zur Expression von T3SS-2. Die Sekretion von T3SS-2-Effektoren durch Salmonellen ist für ihr effizientes Überleben im Zytosol des Wirts und die Entstehung einer systemischen Erkrankung erforderlich. Darüber hinaus sind beide T3SS an der Besiedlung des Darms, der Auslösung von Entzündungsreaktionen im Darm und der Diarrhöe beteiligt. Diese Systeme enthalten viele Gene, die zusammenarbeiten müssen, um eine Infektion zu erreichen. ⓘ
Das AvrA-Toxin, das durch das SPI1-Typ-III-Sekretionssystem von S. Typhimurium injiziert wird, hemmt das angeborene Immunsystem durch seine Serin/Threonin-Acetyltransferase-Aktivität und erfordert die Bindung an eukaryotische Zielzellen mit Phytinsäure (IP6). Dies macht den Wirt anfälliger für Infektionen. ⓘ
Klinische Symptome
Es ist bekannt, dass die Salmonellose Rückenschmerzen oder Spondylose verursachen kann. Sie kann sich in fünf klinischen Mustern manifestieren: Infektion des Gastrointestinaltrakts, enterisches Fieber, Bakteriämie, lokale Infektion und chronischer Reservoirzustand. Die ersten Symptome sind unter anderem unspezifisches Fieber, Schwäche und Myalgie. Im Stadium der Bakteriämie kann sich der Erreger auf alle Teile des Körpers ausbreiten und eine lokale Infektion verursachen oder Abszesse bilden. Zu den Formen der lokalisierten Salmonelleninfektionen gehören Arthritis, Harnwegsinfektionen, Infektionen des zentralen Nervensystems, Knocheninfektionen, Weichteilinfektionen usw. Die Infektion kann lange als latente Form bestehen bleiben, und wenn sich die Funktion der retikulären Endothelzellen verschlechtert, kann sie aktiviert werden und folglich mehrere Monate oder Jahre nach der akuten Salmonellose sekundär eine sich ausbreitende Infektion im Knochen hervorrufen. ⓘ
Eine Studie des Imperial College London aus dem Jahr 2018 zeigt auch, wie Salmonellen mithilfe einer Familie von Zink-Metalloproteinase-Effektoren bestimmte Bereiche des Immunsystems (z. B. 3 von 5 NF-kappaB-Proteinen) stören und andere unberührt lassen. ⓘ
Resistenz gegen oxidativen Burst
Ein Markenzeichen der Salmonella-Pathogenese ist die Fähigkeit des Bakteriums, in Phagozyten zu überleben und sich dort zu vermehren. Phagozyten produzieren zur Abwehr von Krankheitserregern DNA-schädigende Stoffe wie Stickstoffmonoxid und Sauerstoffradikale. Daher müssen Salmonella-Arten mit Molekülen angegriffen werden, die die Integrität des Genoms in Frage stellen. Buchmeier et al. zeigten, dass Mutanten von S. enterica, denen die Funktion des RecA- oder RecBC-Proteins fehlt, sehr empfindlich auf oxidative Verbindungen reagieren, die von Makrophagen synthetisiert werden, und diese Ergebnisse deuten darauf hin, dass eine erfolgreiche systemische Infektion durch S. enterica eine RecA- und RecBC-vermittelte rekombinative Reparatur von DNA-Schäden erfordert. ⓘ
Wirtsanpassung
S. enterica zeigt mit einigen seiner Serotypen wie Typhimurium und Enteritidis Anzeichen für die Fähigkeit, mehrere verschiedene Wirtssäugetierarten zu infizieren, während andere Serotypen wie Typhi anscheinend auf einige wenige Wirte beschränkt sind. Zu den Möglichkeiten, wie sich Salmonella-Serotypen an ihre Wirte angepasst haben, gehören der Verlust von genetischem Material und Mutation. Bei komplexeren Säugetierarten richten sich die Immunsysteme, zu denen auch erregerspezifische Immunreaktionen gehören, gegen Salmonella-Serotypen, indem sie Antikörper an Strukturen wie die Geißeln binden. Durch den Verlust des genetischen Materials, das für die Bildung einer Geißel kodiert, können Salmonellen das Immunsystem des Wirts umgehen. Die mgtC-Leader-RNA des bakteriellen Virulenzgens (mgtCBR-Operon) verringert die Flagellinproduktion während der Infektion durch direkte Basenpaarung mit mRNAs des fljB-Gens, das für Flagellin kodiert, und fördert den Abbau. In der Studie von Kisela et al. wurde festgestellt, dass mehr pathogene Serovare von S. enterica bestimmte Adhäsine gemeinsam haben, die sich aus einer konvergenten Evolution entwickelt haben. Das bedeutet, dass diese Salmonellenstämme, die ähnlichen Bedingungen wie Immunsystemen ausgesetzt waren, ähnliche Strukturen entwickelt haben, um diese ähnlichen, fortschrittlicheren Abwehrmechanismen der Wirte zu überwinden. Es bleiben immer noch viele Fragen über die Art und Weise, wie sich Salmonella in so viele verschiedene Typen entwickelt hat, aber Salmonella hat sich möglicherweise in mehreren Phasen entwickelt. Wie Baumler et al. vorschlagen, hat sich Salmonella höchstwahrscheinlich durch horizontalen Gentransfer, die Bildung neuer Serovare aufgrund zusätzlicher Pathogenitätsinseln und eine Annäherung an seine Abstammung entwickelt. Salmonella könnte sich also durch den Erwerb von genetischen Informationen aus verschiedenen pathogenen Bakterien zu seinen vielen verschiedenen Serotypen entwickelt haben. Das Vorhandensein mehrerer Pathogenitätsinseln im Genom der verschiedenen Serotypen hat diese Theorie untermauert. ⓘ
Salmonella sv. Newport weist Anzeichen für eine Anpassung an eine pflanzliche Besiedlungsweise auf, was eine Rolle bei der überproportionalen Assoziation mit lebensmittelbedingten Krankheiten im Zusammenhang mit Produkten spielen könnte. Eine Vielzahl von Funktionen, für die während der Persistenz von sv. Newport in Tomaten selektiert wurden, ähneln denen, für die bei sv. Typhimurium aus tierischen Wirten. Das papA-Gen, das nur bei sv. Newport ist, trägt zur Fitness des Stammes in Tomaten bei und hat Homologe in Genomen anderer Enterobacteriaceae, die pflanzliche und tierische Wirte besiedeln können. ⓘ
Forschung
Neben ihrer Bedeutung als Krankheitserreger werden nicht-typhoide Salmonella-Arten wie S. enterica serovar Typhimurium häufig als Homologe von Typhus-Arten verwendet. Viele Befunde sind übertragbar und mindern die Gefahr für den Forscher im Falle einer Kontamination, sind aber auch begrenzt. So ist es beispielsweise nicht möglich, mit diesem Modell spezifische Typhustoxine zu untersuchen. Starke Forschungsinstrumente wie das häufig verwendete Maus-Darm-Gastroenteritis-Modell bauen jedoch auf der Verwendung von Salmonella Typhimurium auf. ⓘ
Im Bereich der Genetik war S. Typhimurium maßgeblich an der Entwicklung von genetischen Werkzeugen beteiligt, die zu einem Verständnis der grundlegenden bakteriellen Physiologie führten. Diese Entwicklungen wurden durch die Entdeckung des ersten generalisierten transduzierenden Phagen P22 in S. Typhimurium ermöglicht, der eine schnelle und einfache genetische Bearbeitung ermöglichte. Dies wiederum ermöglichte die genetische Feinstrukturanalyse. Die große Zahl der Mutanten führte zu einer Überarbeitung der genetischen Nomenklatur für Bakterien. Viele der Anwendungen von Transposons als genetische Werkzeuge, einschließlich der Transposonübertragung, der Mutagenese und der Konstruktion von Chromosomenumlagerungen, wurden ebenfalls in S. Typhimurium entwickelt. Diese genetischen Werkzeuge führten auch zu einem einfachen Test auf Karzinogene, dem Ames-Test. ⓘ
Alte DNA
Die Genome von S. enterica wurden aus bis zu 6 500 Jahre alten menschlichen Überresten aus Westeurasien rekonstruiert, was Hinweise auf geografisch weit verbreitete Infektionen mit systemischen S. enterica in der Vorgeschichte und eine mögliche Rolle des Neolithisierungsprozesses bei der Evolution der Wirtsanpassung liefert. Weitere rekonstruierte Genome aus dem kolonialen Mexiko deuten darauf hin, dass S. enterica die Ursache von Cocoliztli, einer Epidemie im Neuspanien des 16. ⓘ
Beschreibung
Salmonellen sind stäbchenförmige gramnegative Bakterien mit einem Durchmesser zwischen etwa 0,7 bis 1,5 µm und einer Länge von etwa 2 bis 5 µm. Sie sind vorwiegend aktiv beweglich, peritrich begeißelt, fakultativ anaerob, chemoorganotroph mit oxidativem und fermentativem Energiestoffwechsel und nicht sporenbildend. Sie werden in der Gattung Salmonella zusammengefasst und der Familie der Enterobacteriaceae zugeordnet. Sie sind eng verwandt mit der Gattung Escherichia. Sie kommen weltweit in wechsel- und gleichwarmen Tieren, in Menschen und in Habitaten außerhalb von Lebewesen vor. ⓘ
Epidemiologische Gruppen
Einordnung der Serovare nach der Anpassung an bestimmte Wirte:
- an den Menschen angepasste Serovare, die bei diesem Typhus oder Paratyphus verursachen (z. B. Salmonella Typhi, Salmonella Paratyphi A, B und C),
- an bestimmte Tierarten angepasste Serovare, die tierspezifische Erkrankungen hervorrufen und für andere Tierarten und den Menschen nicht von Bedeutung sind Salmonella Dublin (Rind), Salmonella Choleraesuis (Schwein), Salmonella Abortusovis, Abortusequi (Schaf, Pferd) und
- Serovare ohne spezielle Wirtsanpassung, die bei allen Tierspezies als Erreger von Enteritiden auftreten und beim Menschen Lebensmittelvergiftungen hervorrufen.
- Serovare ohne spezielle Wirtsanpassung, die bei Mensch und Tier als Erreger von Salmonellosen auftreten und eine hohe Virulenz besitzen. ⓘ
Infektionsmöglichkeiten
Infektionen mit Salmonellen sind möglich:
- durch Unsauberkeit im Lebensmittelbereich, insbesondere in Küchen
- durch die Ausscheidungen von erkrankten, aber auch klinisch gesund erscheinenden infizierten Menschen (Dauerausscheider) und Tieren (gefährdet: Pflegepersonal und andere Tiere); vor allem auch von unerkannt infizierten Reptilien (Befallsrate bei 90 %), eine Gefahr vor allem für Kleinkinder
- durch verunreinigtes Oberflächenwasser und abgestandenes Wasser (beispielsweise in Duschschläuchen und lange nicht benutzten Wasserspendern)
- durch unhygienisch aufgetautes Geflügel (viele Bakterien befinden sich im Tauwasser) sowie
- durch rohe Eier, die von mit Salmonellen infiziertem Geflügel stammen (die Salmonellen befinden sich normalerweise nur auf der Eierschale, können bei verletzter Kutikula jedoch auch ins Innere gelangen). ⓘ
Medizinisch bedeutsame Vertreter
- Salmonella enterica ssp. arizonae, bei Kaltblütern, Geflügel, Säugetieren
- Salmonella Choleraesuis (Bacillus paratyphus B und C), Darmkommensale des Schweines, pathogen bei Resistenzschwäche; Menschen können sich durch den Verzehr vom Fleisch erkrankter Schweine infizieren, verursacht die Salmonellose des Schweins.
- Salmonella Enteritidis, Vorkommen im Darm von Rindern, Nagetieren, Enten (auch deren Eiern) und Menschen; Erreger des Kälberparatyphus und akuter Gastroenteritis des Menschen.
- Salmonella Paratyphi A, rein humanpathogen, Erreger des „Paratyphus A“ (Paratyphöse Gastroenteritis), Übertragung durch Kontakt und infektiöse Lebensmittel oder Wasser.
- Salmonella Paratyphi B: Von dieser Serovarietät existieren zwei Varianten, die sich hinsichtlich der Fermentation von d-Tartrat unterscheiden. Die d-Tartrat-negative Variante kommt praktisch nur bei Menschen vor, gilt als Erreger des „Paratyphus B“ und kommt in Mitteleuropa kaum vor. Die d-Tartrat-positive Variante, ist hingegen in Mitteleuropa vorherrschend. Sie hat ein tierisches Reservoir, gilt als Auslöser einer Salmonellenenteritis und wird auch als Salmonella Java bezeichnet.
- Salmonella Typhi, Vorkommen in gemäßigten und subtropischen Zonen, humanpathogener Erreger des Typhus abdominalis, Übertragung durch Kontakt und infektiöse Lebensmittel, Wasser oder Fliegenkot. 3–5 % aller Erkrankten bleiben Dauerausscheider.
- Salmonella Typhimurium, Erreger einer meist tödlich verlaufenden, fieberhaften Darminfektion bei Vögeln und Säugetieren, durch kontaminierte Futtermittel. Auslöser der Salmonellenenteritis („Lebensmittelvergiftung“) des Menschen.
- Salmonella Dublin, einer der Erreger der anzeigepflichtigen Rindersalmonellose
- Salmonella Typhisuis, einer der Verursacher der Salmonellose des Schweins ⓘ
Meldepflicht
In Deutschland ist jeder direkte Nachweis von Salmonella Paratyphi oder Salmonella Typhi namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes. Zudem der direkte oder indirekte Nachweis sonstiger Salmonellen, soweit der Nachweis auf eine akute Infektion hinweist. Die Meldepflicht betrifft in erster Linie die Leitungen von Laboren (§ 8 IfSG). ⓘ
In der Schweiz ist der positive und negative laboranalytische Befund zu Salmonellen (Salmonella spp.) für Laboratorien meldepflichtig und zwar nach dem Epidemiengesetz (EpG) in Verbindung mit der Epidemienverordnung und Anhang 3 der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen. ⓘ
