Pseudomonas
| Pseudomonas ⓘ | |
|---|---|
| |
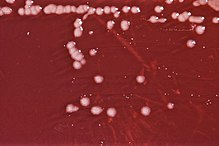
| P. aeruginosa-Kolonien auf einer Agarplatte | |
| Wissenschaftliche Klassifizierung | |
| Bereich: | Bakterien |
| Phylum: | Pseudomonadota |
| Klasse: | Gammaproteobakterien |
| Ordnung: | Pseudomonadales |
| Familie: | Pseudomonadaceae |
| Gattung: | Pseudomonas Migula 1894 |
| Typusart | |
| Pseudomonas aeruginosa | |
| Art | |
|
Siehe Text. | |
| Synonyme | |
| |
Pseudomonas ist eine Gattung gramnegativer Gammaproteobakterien, die zur Familie der Pseudomonadaceae gehört und 191 beschriebene Arten umfasst. Die Mitglieder der Gattung weisen eine große Stoffwechselvielfalt auf und sind daher in der Lage, ein breites Spektrum von Nischen zu besiedeln. Ihre leichte Kultivierbarkeit in vitro und die Verfügbarkeit einer zunehmenden Anzahl von Genomsequenzen von Pseudomonas-Stämmen haben die Gattung zu einem ausgezeichneten Fokus für die wissenschaftliche Forschung gemacht; zu den am besten untersuchten Arten gehören P. aeruginosa in seiner Rolle als opportunistischer Humanpathogen, der Pflanzenpathogen P. syringae, das Bodenbakterium P. putida und die das Pflanzenwachstum fördernden P. fluorescens, P. lini, P. migulae und P. graminis. ⓘ
Aufgrund ihres weit verbreiteten Vorkommens im Wasser und in Pflanzensamen, z. B. bei Dikotyledonen, wurden die Pseudomonaden schon früh in der Geschichte der Mikrobiologie beobachtet. Der für diese Organismen geschaffene Gattungsname Pseudomonas wurde von Walter Migula in den Jahren 1894 und 1900 recht vage als Gattung gramnegativer, stäbchenförmiger und polar geflaggter Bakterien mit einigen sporulierenden Arten definiert. Die letztgenannte Aussage erwies sich später als falsch und war auf refraktive Körnchen von Reservestoffen zurückzuführen. Trotz der vagen Beschreibung erwies sich die Typusart, Pseudomonas pyocyanea (Basonym von Pseudomonas aeruginosa), als die beste Beschreibung. ⓘ
Pseudomonas (fem.: die Pseudomonas, Plural für die der Gattung angehörigen Organismen: Pseudomonaden, von altgriechisch ψεῦδος pseúdos, deutsch ‚Täuschung‘, μονάς monás, deutsch ‚Einheit‘) ist eine Gattung stäbchenförmiger, mit polaren Geißeln sich aktiv bewegender, immer anaerob oder aerob veratmend, nie fermentierender, gramnegativer Bakterien. Sporen werden nicht gebildet. Das Genom der Art P. aeruginosa wurde vollständig sequenziert. ⓘ
Geschichte der Klassifizierung
Wie die meisten Bakteriengattungen lebte auch der letzte gemeinsame Vorfahre der Pseudomonaden vor Hunderten von Millionen von Jahren. Ihre erste Klassifizierung erfolgte Ende des 19. Jahrhunderts, als sie von Walter Migula erstmals identifiziert wurden. Die Etymologie des Namens wurde damals nicht spezifiziert und erschien erstmals in der siebten Ausgabe von Bergey's Manual of Systematic Bacteriology (der wichtigsten Autorität in der bakteriellen Nomenklatur) als griechisches pseudes (ψευδής) "falsch" und -monas (μονάς/μονάδος) "eine einzelne Einheit", was falsche Einheit bedeuten kann; Migula meinte damit jedoch möglicherweise den falschen Monas, einen nanoflagellaten Protisten (später wurde der Begriff "Monade" in der frühen Geschichte der Mikrobiologie zur Bezeichnung von Einzellern verwendet). Schon bald wurden weitere Arten, die Migulas etwas vager Originalbeschreibung entsprachen, aus vielen natürlichen Nischen isoliert, und viele wurden damals der Gattung zugeordnet. Inzwischen wurden jedoch viele Stämme neu klassifiziert, und zwar auf der Grundlage neuerer Methoden und unter Verwendung von Ansätzen, die Untersuchungen konservativer Makromoleküle beinhalten. ⓘ
Vor kurzem hat die 16S rRNA-Sequenzanalyse die Taxonomie vieler Bakterienarten neu definiert. Infolgedessen umfasst die Gattung Pseudomonas Stämme, die früher den Gattungen Chryseomonas und Flavimonas zugeordnet waren. Andere Stämme, die früher zur Gattung Pseudomonas gehörten, werden jetzt den Gattungen Burkholderia und Ralstonia zugeordnet. ⓘ
Im Jahr 2020 wurden im Rahmen einer phylogenomischen Analyse von 494 vollständigen Pseudomonas-Genomen zwei klar definierte Arten (P. aeruginosa und P. chlororaphis) und vier breitere phylogenetische Gruppen (P. fluorescens, P. stutzeri, P. syringae, P. putida) mit einer ausreichenden Anzahl verfügbarer Proteome identifiziert. Die vier größeren evolutionären Gruppen umfassen mehr als eine Spezies, basierend auf der Speziesdefinition anhand der durchschnittlichen Nukleotididentitätswerte. Darüber hinaus wurden bei der phylogenomischen Analyse mehrere Stämme identifiziert, die fälschlicherweise der falschen Art oder Evolutionsgruppe zugeordnet wurden. Dieses Problem der falschen Zuordnung wurde auch bei anderen Analysen festgestellt. ⓘ
Genomik
Im Jahr 2000 wurde die vollständige Genomsequenz einer Pseudomonas-Spezies bestimmt; in jüngerer Zeit wurde die Sequenz anderer Stämme bestimmt, darunter die Stämme P. aeruginosa PAO1 (2000), P. putida KT2440 (2002), P. protegens Pf-5 (2005), P. syringae pathovar tomato DC3000 (2003), P. syringae pathovar syringae B728a (2005), P. syringae pathovar phaseolica 1448A (2005), P. fluorescens Pf0-1 und P. entomophila L48. ⓘ
Bis 2016 waren mehr als 400 Pseudomonas-Stämme sequenziert worden. Die Sequenzierung der Genome von Hunderten von Stämmen ergab hochgradig divergierende Arten innerhalb der Gattung. Tatsächlich teilen viele Pseudomonas-Genome nur 50-60 % ihrer Gene, z. B. haben P. aeruginosa und P. putida nur 2971 von 5350 Proteinen (oder ~55 %) gemeinsam. ⓘ
Bis 2020 waren mehr als 500 vollständige Pseudomonas-Genome in der Genebank verfügbar. Bei einer phylogenomischen Analyse wurden 494 vollständige Proteome verwendet und 297 zentrale Orthologe identifiziert, die von allen Stämmen geteilt werden. Dieser Satz von Kernorthologen auf Gattungsebene war angereichert mit Proteinen, die am Stoffwechsel, der Translation und der Transkription beteiligt sind, und wurde zur Erstellung eines phylogenomischen Baums der gesamten Gattung verwendet, um die Beziehungen zwischen den wichtigsten evolutionären Gruppen von Pseudomonas zu beschreiben. Darüber hinaus wurden für die meisten evolutionären Gruppen gruppenspezifische Kernproteine identifiziert, was bedeutet, dass sie in allen Mitgliedern der spezifischen Gruppe vorhanden sind, aber in anderen Pseudomonaden fehlen. So wurden beispielsweise mehrere P. aeruginosa-spezifische Kernproteine identifiziert, von denen bekannt ist, dass sie eine wichtige Rolle bei der Pathogenität dieser Art spielen, wie z. B. CntL, CntM, PlcB, Acp1, MucE, SrfA, Tse1, Tsi2, Tse3 und EsrC. ⓘ
Merkmale
Die Größe variiert zwischen 0,5–1,0 × 1,5–5,0 µm. Sie können sich mit einer oder mehreren polaren Geißeln aktiv bewegen (sie sind motil). Sie reagieren im Voges-Proskauer-Test negativ, im Katalase-Test positiv. Bei den meisten Arten fällt der Oxidase-Test positiv aus, z. B. bei Pseudomonas fluorescens, diese Arten besitzen somit ein c-Typ-Cytochrom. ⓘ
Pseudomonaden sind aerob, das heißt, sie brauchen in der Regel Sauerstoff für ihren Energiestoffwechsel, um zu wachsen. Sie nutzen also einen oxidativen Energiestoffwechsel mit Sauerstoff als Elektronenakzeptor. Zucker werden dabei über den Entner-Doudoroff-Weg zur Energiegewinnung abgebaut. Nitrat kann bei den meisten Arten als alternativer Elektronenakzeptor statt Sauerstoff bei der Atmung dienen (Nitratatmung). In diesem Fall wird Nitrat (NO3−) zu elementarem, molekularem Stickstoff (N2) reduziert (Denitrifikation). Da für die Nitratatmung kein Sauerstoff nötig ist, sind einige Arten auch in der Lage, unter Ausschluss frei gelösten Sauerstoffs (anoxisch) zu wachsen, doch dies ist eher selten der Fall. Als Vertreter der nitratreduzierenden Bakterien werden die Arten Pseudomonas aeruginosa, Pseudomonas stutzeri und Pseudomonas denitrificans bei der Abfallbehandlung zur Nitratelimination eingesetzt. Eine der wenigen Arten, die nicht zur Nitratatmung fähig sind, ist Pseudomonas fluorescens. ⓘ
Fermentation wird von dieser Gattung nicht als Energiestoffwechsel genutzt, alle Arten sind sogenannte Nonfermenter (Nichtfermentierer). ⓘ
Mehrere Pseudomonas-Arten produzieren bei Eisenmangel Siderophore; mindestens 50 verschiedene Pyoverdine wurden gefunden, außerdem Ferrioxamine, die sonst nur in Actinobakterien vorkommen. ⓘ
Kulturen von P. aeruginosa zeichnen sich durch eine blau-grüne Färbung des Kulturmediums und süßlich-aromatischen Geruch aus. ⓘ
Pseudomonas ist möglicherweise der häufigste Keimbildner von Eiskristallen in Wolken und damit von größter Bedeutung für die Bildung von Schnee und Regen auf der ganzen Welt. ⓘ
Bildung von Biofilmen
Alle Arten und Stämme von Pseudomonas wurden in der Vergangenheit als strikte Aerobier eingestuft. Ausnahmen von dieser Klassifizierung wurden kürzlich in Pseudomonas-Biofilmen entdeckt. Eine beträchtliche Anzahl von Zellen kann Exopolysaccharide produzieren, die mit der Biofilmbildung verbunden sind. Die Sekretion von Exopolysacchariden wie Alginat erschwert die Phagozytose von Pseudomonaden durch weiße Blutkörperchen von Säugetieren. Die Produktion von Exopolysacchariden trägt auch zu oberflächenbesiedelnden Biofilmen bei, die sich nur schwer von Oberflächen der Lebensmittelzubereitung entfernen lassen. Das Wachstum von Pseudomonaden auf verdorbenen Lebensmitteln kann einen "fruchtigen" Geruch erzeugen. ⓘ
Antibiotikaresistenz
Die meisten Pseudomonas spp. sind von Natur aus resistent gegen Penicillin und die meisten verwandten Beta-Lactam-Antibiotika, einige sind jedoch empfindlich gegenüber Piperacillin, Imipenem, Ticarcillin oder Ciprofloxacin. Aminoglykoside wie Tobramycin, Gentamicin und Amikacin sind weitere Optionen für die Therapie. ⓘ
Diese Fähigkeit, unter rauen Bedingungen zu gedeihen, ist das Ergebnis ihrer widerstandsfähigen Zellwände, die Porine enthalten. Ihre Resistenz gegen die meisten Antibiotika wird auf Efflux-Pumpen zurückgeführt, die einige Antibiotika abpumpen, bevor sie ihre Wirkung entfalten können. ⓘ
Pseudomonas aeruginosa wird zunehmend als opportunistischer Krankheitserreger von klinischer Relevanz erkannt. Eines seiner besorgniserregendsten Merkmale ist seine geringe Antibiotika-Empfindlichkeit. Diese geringe Empfindlichkeit ist auf die konzertierte Aktion von Multidrug-Efflux-Pumpen mit chromosomal kodierten Antibiotikaresistenzgenen (z. B. mexAB-oprM, mexXY usw.) und die geringe Durchlässigkeit der bakteriellen Zellhüllen zurückzuführen. Neben der intrinsischen Resistenz entwickelt P. aeruginosa leicht eine erworbene Resistenz, entweder durch Mutation in chromosomal kodierten Genen oder durch den horizontalen Gentransfer von Antibiotikaresistenzdeterminanten. Die Entwicklung einer Multiresistenz durch P. aeruginosa-Isolate erfordert mehrere verschiedene genetische Ereignisse, zu denen der Erwerb verschiedener Mutationen und/oder der horizontale Transfer von Antibiotikaresistenzgenen gehören. Hypermutation begünstigt die Selektion von mutationsbedingter Antibiotikaresistenz in P. aeruginosa-Stämmen, die chronische Infektionen hervorrufen, während die Anhäufung mehrerer verschiedener Antibiotikaresistenzgene in Integronen den konzertierten Erwerb von Antibiotikaresistenz-Determinanten begünstigt. Einige neuere Studien haben gezeigt, dass phänotypische Resistenz mit der Biofilmbildung oder der Entstehung von Small-Colony-Varianten zusammenhängt, die für die Reaktion von P. aeruginosa-Populationen auf eine Antibiotikabehandlung von Bedeutung sein können. ⓘ
Empfindlichkeit gegenüber Gallium
Obwohl Gallium keine natürliche Funktion in der Biologie hat, interagieren Gallium-Ionen mit zellulären Prozessen in ähnlicher Weise wie Eisen(III). Wenn Galliumionen fälschlicherweise anstelle von Eisen(III) von Bakterien wie Pseudomonas aufgenommen werden, stören die Ionen die Atmung, und die Bakterien sterben. Dies geschieht, weil Eisen redox-aktiv ist und die Übertragung von Elektronen während der Atmung ermöglicht, während Gallium redox-inaktiv ist. ⓘ
Pathogenität
Die Pseudomonaden sind physiologisch hoch flexibel und können auch als opportunistische Krankheitserreger bei bereits geschwächten Pflanzen und Tieren auftreten. Von vielen Pseudomonas-Arten existieren pflanzenpathogene Stämme, so genannte Pathovare (Phytopathogene). Die Art Pseudomonas marginalis verursacht, meist an Sprossachsen, die Weichfäule. Pseudomonas syringae subsp. savastanoi pv. oleae verursacht die Tuberkelkrankheit beim Olivenbaum. Pseudomonas syringae pv. aesculi ist neben der Kastanienminiermotte und seltener Phytophthora-Arten (Oomycetes) ein bedeutender Verursacher des Kastaniensterbens, der den Baum so stark schädigt, dass er anfällig für opportunistische Pilze wird. Bei Tieren kann u. a. die Fleckenseuche der Süßwasserfische von Pseudomonas verursacht werden. Arten wie Pseudomonas aeruginosa können für Menschen mit geschwächtem Immunsystem gefährlich sein. ⓘ
Die Virulenzgene sind meistens mobil (Plasmide) und können leicht von einer Pseudomonas-Art auf eine andere, aber auch auf entfernter verwandte Bakterien übertragen werden. So besitzen die Pseudomonaden oft ein sogenanntes R-Plasmid, welches der Übertragung der Resistenzgene dient. ⓘ
Das hochgefährliche Nervengift Tetrodotoxin (TTX) wird von einigen Pseudomonas-Arten produziert. Auch anhydro-TTX, eine im molekularen Aufbau leicht abweichende Variante, wurde bei dieser Gattung nachgewiesen. Pseudomonas und auch verschiedene Vibrionen werden für die starke Giftigkeit verschiedener Kugelfische verantwortlich gemacht. Der Fisch nimmt die Bakterien vermutlich durch die Nahrung (zum Beispiel verschiedene Krebstiere, Würmer und Rotalgen) auf. Pseudomonas wurde bei dem Kugelfisch Takifugu poecilonotus (Synonym: Fugu poecilonotus) nachgewiesen. Man geht hierbei von einer Symbiose zwischen dem Kugelfisch und den Bakterien aus. Der Kugelfisch setzt das Gift für die Verteidigung bzw. Abschreckung gegen Räuber ein und liefert den Bakterien wiederum ein geeignetes Habitat. Die Fähigkeit der Bakterien, insbesondere der Vibrionen, Tetrodotoxin zu bilden, wurde allerdings wieder in Frage gestellt. ⓘ
Tierische Krankheitserreger
Zu den infektiösen Arten gehören P. aeruginosa, P. oryzihabitans und P. plecoglossicida. P. aeruginosa gedeiht in Krankenhausumgebungen und stellt in diesem Umfeld ein besonderes Problem dar, da es die zweithäufigste Infektion bei Krankenhauspatienten ist (nosokomiale Infektionen). Diese Pathogenese könnte zum Teil auf die von P. aeruginosa sezernierten Proteine zurückzuführen sein. Das Bakterium verfügt über ein breites Spektrum an Sekretionssystemen, die zahlreiche Proteine exportieren, die für die Pathogenese klinischer Stämme relevant sind. Interessanterweise sind mehrere Gene, die an der Pathogenese von P. aeruginosa beteiligt sind, wie CntL, CntM, PlcB, Acp1, MucE, SrfA, Tse1, Tsi2, Tse3 und EsrC kerngruppenspezifisch, d. h. sie werden von der überwiegenden Mehrheit der P. aeruginosa-Stämme genutzt, sind aber in anderen Pseudomonaden nicht vorhanden. ⓘ
Pflanzenpathogene
P. syringae ist ein produktiver Pflanzenpathogen. Es gibt über 50 verschiedene Pathovare, von denen viele ein hohes Maß an Wirtspflanzen-Spezifität aufweisen. Zahlreiche andere Pseudomonas-Arten können pflanzenpathogen sein, insbesondere alle anderen Mitglieder der Untergruppe P. syringae, aber P. syringae ist am weitesten verbreitet und am besten erforscht. ⓘ
P. tolaasii ist zwar kein Pflanzenpathogen im eigentlichen Sinne, kann aber in der Landwirtschaft ein großes Problem darstellen, da er die bakterielle Fleckenkrankheit bei Zuchtpilzen verursachen kann. In ähnlicher Weise kann P. agarici bei Zuchtpilzen tropfenförmige Kiemen verursachen. ⓘ
Verwendung als Biokontrollmittel
Seit Mitte der 1980er Jahre werden bestimmte Vertreter der Gattung Pseudomonas auf Getreidesamen oder direkt auf den Boden aufgebracht, um das Wachstum oder die Ansiedlung von Krankheitserregern zu verhindern. Diese Praxis wird allgemein als Biokontrolle bezeichnet. Die biologischen Bekämpfungseigenschaften von P. fluorescens und P. protegens-Stämmen (z. B. CHA0 oder Pf-5) sind derzeit am besten bekannt, obwohl nicht genau klar ist, wie die pflanzenwachstumsfördernden Eigenschaften von P. fluorescens erreicht werden. Zu den Theorien gehören: Die Bakterien könnten in der Wirtspflanze eine systemische Resistenz hervorrufen, so dass sie dem Angriff eines echten Krankheitserregers besser widerstehen kann; die Bakterien könnten andere (pathogene) Bodenmikroben ausstechen, z. B. durch Siderophore, die einen Wettbewerbsvorteil bei der Eisenaufnahme verschaffen; die Bakterien könnten Verbindungen produzieren, die anderen Bodenmikroben gegenüber antagonistisch sind, z. B. Antibiotika vom Phenazin-Typ oder Blausäure. Alle diese Theorien werden durch experimentelle Beweise gestützt. ⓘ
Weitere bemerkenswerte Pseudomonas-Arten mit Biokontroll-Eigenschaften sind P. chlororaphis, das ein Antibiotikum vom Phenazin-Typ produziert, das gegen bestimmte pilzliche Pflanzenpathogene wirkt, und die eng verwandte Art P. aurantiaca, die Di-2,4-diacetylfluoroglucylmethan produziert, eine Verbindung, die gegen grampositive Organismen antibiotisch wirkt. ⓘ
Verwendung als Bioremediationsmittel
Einige Mitglieder der Gattung sind in der Lage, chemische Schadstoffe in der Umwelt zu metabolisieren und können daher für die Bioremediation verwendet werden. Zu den bemerkenswerten Arten, die sich nachweislich für die Verwendung als Bioremediationsmittel eignen, gehören:
- P. alcaligenes, das polyzyklische aromatische Kohlenwasserstoffe abbauen kann.
- P. mendocina, die in der Lage ist, Toluol abzubauen.
- P. pseudoalcaligenes, das Cyanid als Stickstoffquelle nutzen kann.
- P. resinovorans, das Carbazol abbauen kann.
- P. aeruginosa, P. putida, P. desmolyticum und P. nitroreducens können Chlorpyrifos abbauen.
- P. veronii, das nachweislich eine Vielzahl einfacher aromatischer organischer Verbindungen abbauen kann.
- P. putida, das in der Lage ist, organische Lösungsmittel wie Toluol abzubauen. Mindestens ein Stamm dieses Bakteriums ist in der Lage, Morphin in wässriger Lösung in das stärkere und etwas teurer herzustellende Medikament Hydromorphon (Dilaudid) umzuwandeln.
- Stamm KC von P. stutzeri, der in der Lage ist, Tetrachlorkohlenstoff abzubauen. ⓘ
Nachweis von Lebensmittelverderbniserregern in Milch
Eine Möglichkeit zur Identifizierung und Kategorisierung mehrerer bakterieller Organismen in einer Probe ist die Ribotypisierung. Bei der Ribotypisierung werden unterschiedliche Längen der chromosomalen DNA aus Proben isoliert, die Bakterienarten enthalten, und in Fragmente zerlegt. Ähnliche Arten von Fragmenten aus verschiedenen Organismen werden sichtbar gemacht und ihre Längen durch Southern Blotting oder die viel schnellere Methode der Polymerase-Kettenreaktion (PCR) miteinander verglichen. Die Fragmente können dann mit Sequenzen verglichen werden, die bei Bakterienarten gefunden wurden. Die Ribotypisierung hat sich als Methode zur Isolierung von Bakterien erwiesen, die zum Verderb beitragen können. Etwa 51 % der in milchverarbeitenden Betrieben gefundenen Pseudomonas-Bakterien sind P. fluorescens, wobei 69 % dieser Isolate Proteasen, Lipasen und Lecithinasen besitzen, die zum Abbau von Milchbestandteilen und damit zum Verderb beitragen. Andere Pseudomonas-Arten können eine der Proteasen, Lipasen oder Lecithinasen oder auch gar keine besitzen. Pseudomonas desselben Ribotyps üben ähnliche enzymatische Aktivitäten aus, wobei jeder Ribotyp unterschiedliche Grade des Milchverderbs und Auswirkungen auf den Geschmack zeigt. Die Anzahl der Bakterien beeinflusst die Intensität des Verderbs, wobei nicht-enzymatische Pseudomonas-Arten in großer Zahl zum Verderb beitragen. ⓘ
Der Verderb von Lebensmitteln ist für die Lebensmittelindustrie schädlich, da die Organismen flüchtige Verbindungen produzieren, die die verschiedenen Nährstoffe in den Lebensmitteln verstoffwechseln. Verunreinigungen führen zu Gesundheitsrisiken durch die Bildung toxischer Verbindungen sowie zu unangenehmen Gerüchen und Geschmacksrichtungen. Die Technologie der elektronischen Nase ermöglicht eine schnelle und kontinuierliche Messung des mikrobiellen Verderbs von Lebensmitteln, indem sie Gerüche wahrnimmt, die durch diese flüchtigen Verbindungen entstehen. Die Technologie der elektronischen Nase kann somit eingesetzt werden, um Spuren von Pseudomonas-Milchverderb nachzuweisen und die verantwortliche Pseudomonas-Art zu isolieren. Der Gassensor besteht aus einem Nasenteil, der aus 14 modifizierbaren Polymersensoren besteht, die spezifische, von Mikroorganismen produzierte Milchabbauprodukte erkennen können. Die Sensordaten werden durch Änderungen des elektrischen Widerstands der 14 Polymere bei Kontakt mit der Zielverbindung erzeugt, wobei vier Sensorparameter zur weiteren Spezifizierung der Reaktion eingestellt werden können. Die Antworten können dann von einem neuronalen Netz vorverarbeitet werden, das dann zwischen milchverderbenden Mikroorganismen wie P. fluorescens und P. aureofaciens unterscheiden kann. ⓘ
Art
Pseudomonas umfasst die folgenden Arten, die in genomische Affinitätsgruppen eingeteilt sind: ⓘ
P. aeruginosa-Gruppe
- P. aeruginosa (Schroeter 1872) Migula 1900 (Anerkannte Listen 1980)
- P. citronellolis Seubert 1960 (Anerkannte Listen 1980)
- P. delhiensis Prakash et al. 2007
- "P. denitrificans" Bergey et al. 1961
- P. jinjuensis Kwon et al. 2003
- P. knackmussii Stolz et al. 2007
- P. nicosulfuronedens Li et al. 2021
- P. nitroreducens Iizuka und Komagata 1964 (Anerkannte Listen 1980)
- P. panipatensis Gupta et al. 2008 ⓘ
P. anguilliseptica-Gruppe
- P. anguilliseptica Wakabayashi und Egusa 1972 (Genehmigte Listen 1980)
- P. benzenivorans Lang et al. 2012
- P. borbori Vanparys et al. 2006
- P. campi Timsy et al. 2021
- P. cuatrocienegasensis Escalante et al. 2009
- P. glareae Romanenko et al. 2015
- P. guineae Bozal et al. 2007
- P. guryensis Kim et al. 2021
- P. lalucatii Busquets et al. 2021
- P. leptonychotis Nováková et al. 2020
- P. marincola Romanenko et al. 2008
- P. peli Vanparys et al. 2006
- P. segitis Park et al. 2006
- P. taeanensis Lee et al. 2010
- P. ullengensis Kim et al. 2021 ⓘ
P. fluorescens Gruppe
P. asplenii Untergruppe
- P. agarici Young 1970 (Anerkannte Listen 1980)
- P. asplenii (Ark und Tompkins 1946) Savulescu 1947 (Approved Lists 1980)
- "P. batumici" Kiprianova et al. 2011
- P. fuscovaginae (ex Tanii et al. 1976) Miyajima et al. 1983
- "P. gingeri" Cutri et al. 1984
- P. vanderleydeniana Girard et al. 2022
P. chlororaphis Untergruppe
- P. aurantiaca Nakhimovskaya 1948 (Anerkannte Listen 1980)
- P. aureofaciens Kluyver 1956 (Anerkannte Listen 1980)
- P. chlororaphis (Guignard und Sauvageau 1894) Bergey et al. 1930 (Approved Lists 1980)
- "P. piscium" (Burr et al. 2010) Chen et al. 2018
P. corrugata Untergruppe
- P. alvandae Girard et al. 2022
- P. bijieensis Liang et al. 2021
- P. brassicacearum Achouak et al. 2000
- P. canavaninivorans Hauth et al. 2022
- P. corrugata Roberts und Scarlett 1981
- P. kilonensis Sikorski et al. 2001
- P. marvdashtae Girard et al. 2022
- P. mediterranea Catara et al. 2002
- P. ogarae Garrido-Sanz et al. 2022
- P. tehranensis Girard et al. 2022
- P. thivervalensis Achouak et al. 2000
- P. viciae Zhao et al. 2020
- P. zanjanensis Girard et al. 2022
- P. zarinae Girard et al. 2022
P. fluorescens Untergruppe
- P. allii Sawada et al. 2021
- P. antarctica Reddy et al. 2004
- P. asgharzadehiana Girard et al. 2022
- P. aylmerensis corrig. Tchagang et al. 2021
- P. azadiae Girard et al. 2022
- P. azotoformans Iizuka und Komagata 1963 (Anerkannte Listen 1980)
- P. canadensis Tambong et al. 2017
- P. carnis Lick et al. 2020
- P. cedrina corrig. Dabboussi et al. 2002
- P. costantinii Munsch et al. 2002
- P. cremoris Hofmann et al. 2021
- P. cyclaminis Sawada et al. 2021
- P. edaphica Ramírez-Bahena et al. 2019
- P. extremaustralis López et al. 2010
- P. extremorientalis Ivanova et al. 2002
- P. fildesensis Pavlov et al. 2020
- P. fluorescens Migula 1895 (Anerkannte Listen 1980)
- "P. fulgida" Naureen et al. 2005
- P. grimontii Baïda et al. 2002
- P. haemolytica Hofmann et al. 2020
- P. kairouanensis Oueslati et al. 2020
- P. karstica Švec et al. 2020
- P. khavaziana Girard et al. 2022
- P. kitaguniensis Sawada et al. 2020
- P. lactis von Neubeck et al. 2017
- P. lactucae Sawada et al. 2021
- P. libanensis Dabboussi et al. 1999
- P. lurida Behrendt et al. 2007
- P. marginalis (Brown 1918) Stevens 1925 (Anerkannte Listen 1980)
- P. nabeulensis Oueslati et al. 2020
- P. orientalis Dabboussi et al. 2002
- P. palleroniana Gardan et al. 2002
- P. panacis Park et al. 2005
- P. paracarnis Lick et al. 2021
- P. paralactis von Neubeck et al. 2017
- P. pisciculturae Duman et al. 2021
- P. poae Behrendt et al. 2003
- P. rhodesiae Coroler et al. 1997
- P. salmasensis Girard et al. 2022
- P. salomonii Gardan et al. 2002
- P. simiae Vela et al. 2006
- P. sivasensis Duman et al. 2020
- P. spelaei Švec et al. 2020
- P. synxantha (Ehrenberg 1840) Holland 1920 (Anerkannte Listen 1980)
- P. tolaasii Paine 1919 (Anerkannte Listen 1980)
- P. tritici Girard et al. 2022
- P. trivialis Behrendt et al. 2003
- P. veronii Elomari et al. 1996
- P. yamanorum Arnau et al. 2015
P. fragi Untergruppe
- P. bubulae Lick et al. 2020
- P. deceptionensis Carrión et al. 2011
- P. endophytica Ramírez-Bahena et al. 2015
- P. fragi (Eichholz 1902) Gruber 1905 (Anerkannte Listen 1980)
- P. helleri von Neubeck et al. 2016
- P. lundensis Molin et al. 1986
- P. paraversuta Lick et al. 2021
- P. psychrophila Yumoto et al. 2002
- P. saxonica Hofmann et al. 2020
- P. taetrolens Haynes 1957 (Anerkannte Listen 1980)
- P. versuta See-Too et al. 2017
- P. weihenstephanensis von Neubeck et al. 2016
P. gessardii Untergruppe
- P. brenneri Baïda et al. 2002
- P. gessardii Verhille et al. 1999
- P. meridiana Reddy et al. 2004
- P. mucidolens Levine und Anderson 1932 (Anerkannte Listen 1980)
- P. proteolytica Reddy et al. 2004
- P. shahriarae Girard et al. 2022
P. jessenii Untergruppe
- P. azerbaijanoccidentalis Korrig. Girard et al. 2022
- P. azerbaijanorientalis berichtigt. Girard et al. 2022
- P. izuensis Lu et al. 2020
- P. jessenii Verhille et al. 1999
- P. laurylsulfatiphila berichtigt. Furmanczyk et al. 2019
- P. laurylsulfativorans berichtigt. Furmanczyk et al. 2019
- P. mohnii Cámara et al. 2007
- P. moorei Cámara et al. 2007
- P. reinekei Cámara et al. 2007
- P. umsongensis Kwon et al. 2003
- P. vancouverensis Mohn et al. 1999
P. koreensis Untergruppe
- P. allokribbensis Morimoto et al. 2021
- P. anatoliensis Duman et al. 2021
- P. atacamensis Poblete-Morales et al. 2021
- P. atagonensis Korrig. Morimoto et al. 2020
- P. baetica López et al. 2012
- "P. bananamidigenes" Girard et al. 2021
- "P. botevensis" Girard et al. 2021
- "P. crudilactis" Schlusselhuber et al. 2021
- P. ekonensis Girard et al. 2022
- P. glycinae Jia et al. 2021
- P. gozinkensis Morimoto et al. 2021
- P. granadensis Pascual et al. 2015
- P. hamedanensis Girard et al. 2022
- P. helmanticensis Ramírez-Bahena et al. 2014
- P. iranensis Girard et al. 2022
- P. iridis Duman et al. 2021
- P. khorasanensis Girard et al. 2022
- P. koreensis Kwon et al. 2003
- "P. kribbensis" Chang et al. 2016
- P. monsensis Girard et al. 2022
- P. moraviensis Tvrzová et al. 2006
- P. neuropathica Duman et al. 2021
- P. siliginis Girard et al. 2022
- P. tensinigenes Girard et al. 2022
- P. triticicola Girard et al. 2022
- P. zeae Girard et al. 2022
P. mandelii Untergruppe
- P. arsenicoxydans Campos et al. 2011
- P. farris Girard et al. 2022
- P. frederiksbergensis Andersen et al. 2000
- "P. gregormendelii" Kosina et al. 2016
- P. lini Delorme et al. 2002
- P. mandelii Verhille et al. 1999
- P. migulae Verhille et al. 1999
- P. mucoides Duman et al. 2021
- P. piscicola Duman et al. 2021
- P. prosekii Kosina et al. 2014
- P. silesiensis Kaminski et al. 2018
P. protegens Untergruppe
- "P. aestus" Vasconcellos et al. 2017
- P. piscis Liu et al. 2020
- P. protegens Ramette et al. 2012
- P. saponiphila Lang et al. 2012
- "P. sessilinigenes" Girard et al. 2021
incertae sedis
- "P. blatchfordae" Blatchford und Schuster 1980
- P. kielensis Gieschler et al. 2021 ⓘ
P. linyingensis Gruppe
- P. guangdongensis Yang et al. 2013
- P. linyingensis He et al. 2015
- "P. oryzae" Yu et al. 2013
- P. sagittaria Lin et al. 2013 ⓘ
P. lutea Gruppe
- P. abietaniphila Mohn et al. 1999
- P. bohemica Saati-Santamaría et al. 2018
- P. graminis Behrendt et al. 1999
- P. lutea Peix et al. 2004 ⓘ
P. massiliensis Gruppe
- "P. massiliensis" Bardet et al. 2018
- P. typographi Peral-Aranega et al. 2021 ⓘ
P. oleovorans Gruppe
- P. alcaliphila Yumoto et al. 2001
- "P. chaetocerotis" Girard et al.
- P. chengduensis Tao et al. 2014
- P. composti Gibello et al. 2011
- P. guguanensis Liu et al. 2013
- P. hydrolytica Zhou et al. 2020
- "P. indoloxydans" Manickam et al. 2008
- P. khazarica Tarhriz et al. 2020
- P. mendocina Palleroni 1970 (Anerkannte Listen 1980)
- P. oleovorans Lee und Chandler 1941 (Approved Lists 1980)
- "P. sediminis" Behera et al. 2018
- "P. sihuiensis" Wu et al. 2014
- P. toyotomiensis Hirota et al. 2011 ⓘ
P. oryzihabitans-Gruppe
- P. asuensis Reddy und Garcia-Pichel 2015
- P. duriflava Liu et al. 2008
- P. luteola Kodama et al. 1985
- P. oryzihabitans Kodama et al. 1985
- P. rhizoryzae Wang et al. 2020 ⓘ
P. pohangensis Gruppe
- P. mangrovi Ye et al. 2019
- P. pohangensis Weon et al. 2006 ⓘ
P. putida Gruppe
- P. akappageensis corrig. Morimoto et al. 2020
- P. alkylphenolica Mulet et al. 2015
- P. alloputida Keshavarz-Tohid et al. 2020
- P. anuradhapurensis Girard et al. 2022
- P. arcuscaelestis Mulet et al. 2021
- P. asiatica Tohya et al. 2019
- P. brassicae Sawada et al. 2020
- "P. capeferrum" Berendsen et al. 2015
- P. cremoricolorata Uchino et al. 2002
- P. defluvii Qin et al. 2020
- P. donghuensis Gao et al. 2015
- P. entomophila Mulet et al. 2012
- P. fakonensis Girard et al. 2022
- P. farsensis Girard et al. 2022
- P. fulva Iizuka und Komagata 1963 (Anerkannte Listen 1980)
- P. guariconensis Toro et al. 2013
- P. huaxiensis Qin et al. 2019
- "P. hunanensis" Gao et al. 2014
- "P. hutmensis" Xiang et al. 2019
- P. inefficax Keshavarz-Tohid et al. 2019
- P. japonica Pungrasmi et al. 2008
- P. juntendi Tohya et al. 2019
- P. kermanshahensis Girard et al. 2022
- P. kurunegalensis Girard et al. 2022
- P. laurentiana Wright et al. 2019
- P. maumuensis Girard et al. 2022
- P. monteilii Elomari et al. 1997
- P. mosselii Dabboussi et al. 2002
- P. muyukensis Girard et al. 2022
- P. oryzicola Girard et al. 2022
- "P. oryziphila" Yang et al. 2021
- P. palmensis Gutierrez-Albanchez et al. 2022
- P. parafulva Uchino et al. 2002
- P. peradeniyensis Girard et al. 2022
- P. persica Keshavarz-Tohid et al. 2020
- P. plecoglossicida Nishimori et al. 2000
- P. promysalinigenes Girard et al. 2022
- P. putida (Trevisan 1889) Migula 1895 (Anerkannte Listen 1980)
- "P. pyomelaminifaciens" Chakraborty et al.
- "P. qingdaonensis" Wang et al. 2019
- P. reidholzensis Frasson et al. 2017
- P. shirazensis Girard et al. 2022
- P. shirazica Keshavarz-Tohid et al. 2020
- P. sichuanensis Qin et al. 2019
- P. soli Pascual et al. 2015
- P. taiwanensis Wang et al. 2010
- P. tructae Oh et al. 2019
- P. urmiensis Girard et al. 2022
- "P. vlassakiae" Girard et al. 2021
- P. vranovensis Tvrzová et al. 2006
- P. wadenswilerensis Frasson et al. 2017
- "P. wayambapalatensis" Girard et al. 2021
- P. xantholysinigenes Girard et al. 2022
- P. xanthosomatis corrig. Girard et al. 2022 ⓘ
P. resinovorans Gruppe
- P. furukawaii Kimura et al. 2018
- P. lalkuanensis Thorat et al. 2020
- P. mangiferae Anurat et al. 2019
- P. otitidis Clark et al. 2006
- P. resinovorans Delaporte et al. 1961 (Anerkannte Listen 1980) ⓘ
P. rhizosphaerae-Gruppe
- P. baltica Gieschler et al. 2021
- P. coleopterorum Menéndez et al. 2015
- "P. eucalypticola" Liu et al. 2021
- P. rhizosphaerae Peix et al. 2003 ⓘ
P. straminea Gruppe
- P. argentinensis Peix et al. 2005
- P. daroniae Bueno-Gonzalez et al. 2019
- P. dryadis Bueno-Gonzalez et al. 2019
- P. flavescens Hildebrand et al. 1994
- P. punonensis Ramos et al. 2013
- P. seleniipraecipitans corrig. Hunter und Manter 2011
- P. straminea corrig. Iizuka und Komagata 1963 (Anerkannte Listen 1980) ⓘ
P. stutzeri Gruppe
- P. azotifigens Hatayama et al. 2005
- P. balearica Bennasar et al. 1996
- P. chloritidismutans Wolterink et al. 2002
- P. kirkiae Bueno-Gonzalez et al. 2020
- P. nitrititolerans Peng et al. 2019
- P. nosocomialis Mulet et al. 2019
- "P. saudiphocaensis" Azhar et al. 2017
- "P. songnenensis" Zhang et al. 2015
- P. stutzeri (Lehmann und Neumann 1896) Sijderius 1946 (Approved Lists 1980)
- P. urumqiensis Zou et al. 2019
- P. xanthomarina Romanenko et al. 2005
- P. zhaodongensis Zhang et al. 2015 ⓘ
P. syringae-Gruppe
- "P. alliivorans" Zhao et al. 2021
- P. amygdali Psallidas und Panagopoulos 1975 (Anerkannte Listen 1980)
- P. asturiensis González et al. 2013
- P. avellanae Janse et al. 1997
- P. cannabina (ex Šutič und Dowson 1959) Gardan et al. 1999
- P. capsici Zhao et al. 2021
- P. caricapapayae Robbs 1956 (Anerkannte Listen 1980)
- P. caspiana Busquets et al. 2017
- P. cerasi Kałuzna et al. 2017
- P. cichorii (Swingle 1925) Stapp 1928 (Approved Lists 1980)
- P. congelans Behrendt et al. 2003
- "P. coronafaciens" (Elliott 1920) Stevens 1958
- P. ficuserectae Goto 1983
- P. floridensis Timilsina et al. 2018
- "P. foliumensis" Tambong et al. 2021
- "P. helianthi" Elasri et al. 2001
- P. meliae Ogimi 1981
- P. ovata Rao et al. 2021
- P. savastanoi (Janse 1982) Gardan et al. 1992
- P. syringae van Hall 1902 (Anerkannte Listen 1980)
- "P. Tomate" Gardan et al. 1999
- P. tremae Gardan et al. 1999
- "P. triticumensis" Tambong et al. 2021
- P. viridiflava (Burkholder 1930) Dowson 1939 (Approved Lists 1980) ⓘ
incertae sedis
- "P. acephalitica" Tapia-Paniagua et al. 2014
- "P. acidophila" Imada et al. 1981
- "Ca. P. adelgestsugas" von Dohlen et al. 2013
- P. alcaligenes Monias 1928 (Approved Lists 1980)
- "P. alginovora" Boyen et al. 1990
- "P. alkanolytica" Nakao und Kuno 1972
- "P. amyloderamosa" Norrman und Wober 1975
- "P. andersonii" Han et al. 2001
- "P. bathycetes" Quigley und Colwell 1968
- "P. borealis" Wilson et al. 2006
- P. cavernae Zhu et al. 2022
- P. cavernicola Zhu et al. 2022
- "P. cellulosa" Andrews et al. 2000
- "P. clemancea" Rahman et al. 2010
- "P. coenobios" ZoBell und Upham 1944
- "P. diazotrophicus" Watanabe et al. 1987
- "P. diterpeniphila" Morgan und Wyndham 2002
- "P. elodea" Fialho et al. 1991
- "P. excibis" Steinhaus
- P. flexibilis (Hespell 1977) Shin et al. 2016
- P. fluvialis Sudan et al. 2018
- P. gelidicola Kadota 1951 (Approved Lists 1980)
- "P. guezennei" Simon-Colin et al. 2008
- "P. halodenitrificans" Alonso et al. 2001
- "P. halodurans" Cuhel et al. 1981
- "P. halosaccharolytica" Ohno et al. 1976
- "P. halosensibilis" Zou und Cai 1994
- "P. hydrogenothermophila" Goto et al. 1978
- "P. hydrogenovora" Igarashi et al. 1980
- P. indica Pandey et al. 2002
- "P. jinanensis" Cai et al. 1989
- P. kuykendallii Hunter und Manter 2012
- "P. lopnurensis" Mamtimin et al. 2021
- "P. lubricans" Rehman et al. 2010
- P. matsuisoli Lin et al. 2015
- "P. melophthora" Allen und Riker 1932
- "P. mesoacidophila" Kintaka et al. 1981
- "P. multiresinovorans" Hernandez et al. 2008
- "P. perolens" Szybalski 1950
- P. pharmacofabricae corrig. Yu et al. 2019
- "P. pratensis" Zhang et al. 2021
- P. quercus Li et al. 2021
- "P. raguenesii" Simon-Colin et al. 2009
- "P. reactans" Preece und Wong 1982
- "P. reptilivora" Caldwell und Ryerson 1940
- "P. rhizophila" Hassen et al. 2018
- "P. rhizovicinus" He et al. 2021
- "P. rubescens" Pivnick 1955
- P. schmalbachii Shelomi et al. 2021
- "P. septica" Bergey et al. 1930
- P. sesami Madhaiyan et al. 2017
- "P. siderocapsa" Falamin und Pinevich 2006
- "P. suis" Woods 1930
- "P. tamsuii" Liang et al. 2015
- P. tarimensis Anwar et al. 2017
- "P. teessidea" Rahman et al. 2010
- "P. thermocarboxydovorans" Lyons et al. 1984
- P. thermotolerans Manaia und Moore 2002
- P. tianjinensis Chen et al. 2018
- P. tohonis Yamada et al. 2021
- "P. turbinellae" Sreenivasan 1956
- P. turukhanskensis Korshunova et al. 2016
- "P. tuticorinensis" Sreenivasan 1956
- "P. wenzhouensis" Zhang et al. 2021
- P. xionganensis Zhao et al. 2020
- P. yangonensis Tohya et al. 2020 ⓘ
Früher in der Gattung klassifizierte Arten
Kürzlich hat die 16S rRNA-Sequenzanalyse die Taxonomie vieler Bakterienarten, die zuvor der Gattung Pseudomonas zugeordnet waren, neu definiert. Die aus der Gattung Pseudomonas herausgenommenen Arten sind nachstehend aufgeführt; ein Klick auf eine Art zeigt ihre neue Klassifizierung. Der Begriff "Pseudomonaden" bezieht sich nicht nur auf die Gattung Pseudomonas, sondern kann auch für frühere Mitglieder wie die Gattungen Burkholderia und Ralstonia verwendet werden. ⓘ
α-Proteobakterien: P. abikonensis, P. aminovorans, P. azotocolligans, P. carboxydohydrogena, P. carboxidovorans, P. compransoris, P. diminuta, P. echinoides, P. extorquens, P. lindneri, P. mesophilica, P. paucimobilis, P. radiora, P. rhodos, P. riboflavina, P. rosea, P. vesicularis. ⓘ
β-Proteobakterien: P. acidovorans, P. alliicola, P. antimicrobica, P. avenae, P. butanovora, P. caryophylli, P. cattleyae, P. cepacia, P. cocovenenans, P. delafieldii, P. facilis, P. flava, P. gladioli, P. glathei, P. glumae, P. huttiensis, P. indigofera, P. lanceolata, P. lemoignei, B. mallei, P. mephitica, P. mixta, P. palleronii, P. phenazinium, P. pickettii, P. plantarii, P. pseudoflava, B. pseudomallei, P. pyrrocinia, P. rubrilineans, P. rubrisubalbicans, P. saccharophila, P. solanacearum, P. spinosa, P. syzygii, P. taeniospiralis, P. terrigena, P. testosteroni. ⓘ
γ-β-Proteobakterien: P. boreopolis, P. cissicola, P. geniculata, P. hibiscicola, P. maltophilia, P. pictorum. ⓘ
γ-Proteobakterien: P. beijerinckii, P. diminuta, P. doudoroffii, P. elongata, P. flectens, P. marinus, P. halophila, P. iners, P. marina, P. nautica, P. nigrifaciens, P. pavonacea, P. piscicida, P. stanieri. ⓘ
δ Proteobakterien: P. formicans. ⓘ
Phylogenetik
Die folgenden Beziehungen zwischen den Genom-Affinitätsgruppen wurden durch phylogenetische Analyse ermittelt:
ⓘ
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Bakteriophagen
Es gibt eine Reihe von Bakteriophagen, die Pseudomonas infizieren, z. B. ⓘ
- Pseudomonas-Phagen Φ6
- Pseudomonas-Phagen ΦCTX
- Pseudomonas aeruginosa-Phagen EL
- Pseudomonas aeruginosa Phage ΦKMV (ein Phikmvvirus)
- Pseudomonas aeruginosa Phage LKD16 (ein Phikmvvirus)
- Pseudomonas aeruginosa Phage LKA1 (ein Phikmvvirus)
- Pseudomonas aeruginosa Phage LUZ19 (ein Phikmvvirus)
- Pseudomonas aeruginosa-Phagen ΦKZ
- Pseudomonas putida Phage gh-1 ⓘ
Ökologie
Pseudomonaden sind ubiquitär, also allgegenwärtig in der Umwelt („Pfützenkeim“). Man findet sie im Boden, Wasser sowie in oder auf Pflanzen und Tieren. Pseudomonaden leben oft saprotroph, also von abgestorbenem organischen Material. Man findet sie auch häufig in der Rhizosphäre, diese von den Pflanzen beeinflusste Umgebung stellt ein gutes Habitat für Pseudomonaden dar, sie sind gut an die Rhizosphäre angepasst. So bewegen sie sich unter anderem aktiv, sind chemotaxisch und resistent gegen spezielle Abwehrstoffe der Pflanzen. Obwohl einige pflanzenpathogen sind, tragen viele Arten auch zum Schutz der Pflanzen bei, indem sie innerhalb der Rhizosphäre andere Mikroorganismen verdrängen. Des Weiteren sind einige Arten wie Pseudomonas putida mit arbuskulären Mykorrhizapilzen assoziiert und fördern somit sogar das Wachstum der Pflanzen. Man spricht von plant-growth promoting rhizobacteria (PGPR), zu Deutsch pflanzenwachstumsfördernde Rhizobakterien. ⓘ
Klinische Bedeutung
Die Gattung ist klinisch besonders wichtig, weil die Mehrheit ihrer Mitglieder Resistenzen gegen Antibiotika aufweist. Außerdem sind sie fähig, bei höherer Zelldichte Biofilme (Schleime) zu bilden, die sie gegen Fresszellen und Antibiotika schützen. Während Bakterien der Gattung Pseudomonas bei Menschen mit intaktem Immunsystem selten Krankheiten verursachen, können sie bei Patienten, deren Immunsystem bereits geschwächt ist (beispielsweise in Krankenhäusern, sogenannte Hospitalismuskeime), die Infektion von Wunden, Atem- und Harnwegen, Lungenentzündung sowie Sepsis und Herzerkrankungen verursachen. Wundinfektionen durch P. aeruginosa zeichnen sich durch ihre blau-grüne Färbung und ihren süßlich-aromatischen Geruch aus. Besonders gefährdet sind Patienten mit der Erbkrankheit Cystische Fibrose (Mukoviszidose), bei denen Lungenentzündungen durch Pseudomonas die häufigste Todesursache sind. ⓘ
Zur Therapie eignen sich Acylaminopenicilline wie Piperacillin, Cephalosporine ab der dritten Generation (insbesondere Ceftazidim und Cefepim; Ceftriaxon und Cefotaxim hingegen sind nicht wirksam), Fluorchinolone, Aminoglykoside sowie Carbapeneme. ⓘ
Klinische Studien zu Pseudomonas-Impfungen werden seit einiger Zeit durchgeführt, etwa vom österreichischen Biotech-Unternehmen Intercell, das seinen Impfstoff bereits in Phase III testet. Die referenzierte Studie kam nicht zum gewünschten Ergebnis. ⓘ