Ribosom

1. Nucleolus (Kernkörperchen)
2. Zellkern (Nukleus)
3. Ribosomen
4. Vesikel
5. Raues (Granuläres) ER (Ergastoplasma)
6. Golgi-Apparat
7. Cytoskelett
8. Glattes (Agranuläres) ER
9. Mitochondrien
10. Lysosom
11. Cytoplasma (mit Cytosol und Cytoskelett)
12. Peroxisomen
13. Zentriolen
14. Zellmembran
| Übergeordnet |
| Zytoplasma Endoplasmatisches Retikulum Mitochondrium Chloroplast |
| Gene Ontology |
|---|
| QuickGO |
Ribosomen sind die makromolekularen Komplexe in Zellen, an denen Proteine hergestellt werden. Hierbei wird die Nukleotidsequenz (Basensequenz) eines Messenger-Ribonukleinsäure-Einzelstrangs (mRNA) in die Aminosäurensequenz der Polypeptidkette eines Proteins übersetzt. Diese Umwandlung der in der RNA gespeicherten Information in eine Abfolge von verknüpften Aminosäuren heißt Translation (lateinisch für Übersetzung) und ist in allen Lebewesen ein zentraler Bestandteil der Proteinbiosynthese. Die dabei wirksame Übersetzungsregel wird als Genetischer Code bezeichnet. In der Zelle geschieht die Translation, nachdem die in der Abfolge von Basenpaaren des DNA-Doppelstrangs niedergelegte Erbinformation eines Gens in die Sequenz des mRNA-Einzelstrangs umgeschrieben worden ist. ⓘ
Ribosomen sind aus Ribosomaler RNA, englisch Ribosomal ribonucleic acid (rRNA) und Proteinen (rProtein, auch r-Protein) aufgebaut und finden sich im Cytoplasma sowie in Zellorganellen, die aufgrund ihres endosymbiotischen Ursprungs eine eigene Maschinerie zur Proteinbiosynthese besitzen, wie den Mitochondrien und Chloroplasten. Auch bei einigen Viren sind in den Viruspartikeln Ribosomen enthalten, die von den Wirtszellen stammen, ohne dass sie in diesen eine Funktion erfüllen; beispielsweise sorgen diese bei den Arenaviren für das „sandfarbige“ Aussehen (lateinisch arena ‚Sand‘). ⓘ
| Zellbiologie ⓘ | |
|---|---|
| Diagramm einer tierischen Zelle | |
 Bestandteile einer typischen tierischen Zelle:
|
Ribosomen ( /ˈraɪbəˌsoʊm, -boʊ-/), auch Palade-Granula genannt (nach dem Entdecker George Palade und aufgrund ihrer körnigen Struktur), sind makromolekulare Maschinen, die in allen Zellen vorkommen und die biologische Proteinsynthese (mRNA-Übersetzung) durchführen. Ribosomen verknüpfen Aminosäuren in der durch die Codons der Boten-RNA-Moleküle (mRNA) vorgegebenen Reihenfolge, um Polypeptidketten zu bilden. Ribosomen bestehen aus zwei Hauptkomponenten: den kleinen und großen ribosomalen Untereinheiten. Jede Untereinheit besteht aus einem oder mehreren ribosomalen RNA-Molekülen (rRNA) und vielen ribosomalen Proteinen (RPs oder r-Proteine). Die Ribosomen und die zugehörigen Moleküle werden auch als Translationsapparat bezeichnet. ⓘ
Überblick
Die DNA-Sequenz, die für die Aminosäuresequenz eines Proteins kodiert, wird in eine Boten-RNA-Kette umgeschrieben. Ribosomen binden an Boten-RNAs und verwenden deren Sequenzen, um die richtige Aminosäuresequenz für die Bildung eines bestimmten Proteins zu bestimmen. Die Aminosäuren werden von Transfer-RNA-Molekülen (tRNA) ausgewählt und zum Ribosom transportiert, das sie über eine Anti-Codon-Stammschleife an die Boten-RNA-Kette bindet. Für jedes kodierende Triplett (Codon) in der Boten-RNA gibt es eine eindeutige Transfer-RNA, die genau mit dem Anti-Codon übereinstimmen muss und die richtige Aminosäure zum Einbau in eine wachsende Polypeptidkette trägt. Sobald das Protein hergestellt ist, kann es sich falten, um eine funktionelle dreidimensionale Struktur zu bilden. ⓘ
Ein Ribosom besteht aus RNA- und Proteinkomplexen und ist somit ein Ribonukleoproteinkomplex. Jedes Ribosom besteht aus kleinen (30S) und großen (50S) Komponenten, den so genannten Untereinheiten, die aneinander gebunden sind:
- (30S) hat hauptsächlich eine dekodierende Funktion und ist auch an die mRNA gebunden
- (50S) hat vor allem eine katalytische Funktion und ist auch an die aminoacylierten tRNAs gebunden. ⓘ
Die Synthese von Proteinen aus ihren Bausteinen erfolgt in vier Phasen: Initiation, Elongation, Terminierung und Recycling. Das Startcodon in allen mRNA-Molekülen hat die Sequenz AUG. Das Stoppcodon ist eines der Codons UAA, UAG oder UGA; da es keine tRNA-Moleküle gibt, die diese Codons erkennen, erkennt das Ribosom, dass die Übersetzung abgeschlossen ist. Wenn ein Ribosom das Lesen eines mRNA-Moleküls abgeschlossen hat, trennen sich die beiden Untereinheiten und werden normalerweise getrennt, können aber wiederverwendet werden. Ribosomen sind Ribozyme, denn die katalytische Peptidyltransferase-Aktivität, die Aminosäuren miteinander verbindet, wird von der ribosomalen RNA ausgeführt. ⓘ
Ribosomen sind häufig mit den intrazellulären Membranen verbunden, die das raue endoplasmatische Retikulum bilden. ⓘ
Ribosomen aus Bakterien, Archaeen und Eukaryonten im Drei-Domänen-System ähneln einander in bemerkenswerter Weise, was auf einen gemeinsamen Ursprung hindeutet. Sie unterscheiden sich in ihrer Größe, Sequenz, Struktur und dem Verhältnis von Protein zu RNA. Die Unterschiede in der Struktur ermöglichen es einigen Antibiotika, Bakterien durch Hemmung ihrer Ribosomen abzutöten, während die menschlichen Ribosomen davon unberührt bleiben. Bei allen Arten kann sich mehr als ein Ribosom gleichzeitig entlang einer einzigen mRNA-Kette bewegen (als Polysom), wobei jedes eine bestimmte Sequenz "liest" und ein entsprechendes Proteinmolekül produziert. ⓘ
Die mitochondrialen Ribosomen eukaryontischer Zellen ähneln funktionell in vielen Merkmalen denen von Bakterien, was auf den wahrscheinlichen evolutionären Ursprung der Mitochondrien hinweist. ⓘ
Entdeckung
Ribosomen wurden erstmals Mitte der 1950er Jahre von dem rumänisch-amerikanischen Zellbiologen George Emil Palade mit Hilfe eines Elektronenmikroskops als dichte Partikel oder Körnchen beobachtet. Der Begriff "Ribosom" wurde Ende 1958 von dem Wissenschaftler Haguenau vorgeschlagen:
Im Laufe des Symposiums wurde eine semantische Schwierigkeit deutlich. Für einige Teilnehmer sind mit "Mikrosomen" die Ribonukleoproteinpartikel der Mikrosomenfraktion gemeint, die durch anderes Protein- und Lipidmaterial verunreinigt sind; für andere bestehen die Mikrosomen aus Protein und Lipid, die durch Partikel verunreinigt sind. Die Formulierung "mikrosomale Partikel" erscheint nicht angemessen, und "Ribonukleoproteinpartikel der Mikrosomenfraktion" ist viel zu umständlich. In der Sitzung wurde das Wort "Ribosom" vorgeschlagen, das eine sehr befriedigende Bezeichnung und einen angenehmen Klang hat. Die derzeitige Verwirrung wäre beseitigt, wenn "Ribosom" zur Bezeichnung von Ribonukleoproteinpartikeln in Größen von 35 bis 100S verwendet würde.
- Albert, Mikrosomale Partikel und Proteinsynthese ⓘ
Albert Claude, Christian de Duve und George Emil Palade erhielten 1974 gemeinsam den Nobelpreis für Physiologie oder Medizin für die Entdeckung des Ribosoms. Der Nobelpreis für Chemie 2009 ging an Venkatraman Ramakrishnan, Thomas A. Steitz und Ada E. Yonath für die Bestimmung der detaillierten Struktur und des Mechanismus des Ribosoms. ⓘ
Struktur
Der Abbau von Ribosomen ist noch nicht vollständig verstanden. Er wird in der Regel unter Nährstoffmangel eingeleitet. Für Bakterien wie E. coli wurde vorgeschlagen, dass intakte 70S-Ribosomen zunächst in beide Untereinheiten zerfallen. Unter Mangelbedingungen wird die Translation in der Zelle heruntergefahren, so dass viele Ribosomen inaktiv sind. Die beiden Untereinheiten sind wesentlich empfindlicher gegenüber Ribonukleasen (RNasen) als ein intaktes Ribosom, da sie eine größere Angriffsfläche bieten. Danach könnten auch Exonukleasen die ribosomale RNA weiter abbauen. ⓘ
Für Backhefe, einen Eukaryot, wurde ein mit „Ribophagie“ bezeichneter Autophagieweg vorgeschlagen. Dieser lehnt an die Begriffe Mitophagie (Abbau von Mitochondrien), Pexophagie (Abbau von Peroxisomen) und Reticulophagie (Abbau des Endoplasmatischen Retikulums) an. Unter Nährstoffmangel baut Hefe Ribosomen auf einem Weg ab, der ähnlich wie bei Prokaryoten beginnt. Zunächst werden die beiden Untereinheiten getrennt. Eine Ubiquitinligase entfernt dann Ubiquitin an der 60S-Untereinheit, welche dann in einem Vesikel zur Vakuole transportiert wird. Dies erscheint zunächst paradox, da Ubiquitin ein allgemeines Abbausignal für die meisten Proteine ist. Von den Autoren wurde vorgeschlagen, dass eine Ubiquitinligase die 60S-Untereinheit zunächst für den Abbauweg markiert, der Prozess aber erst durch die Ubiquitinprotease endgültig ablaufen kann. ⓘ


Prokaryotische Ribosomen
Prokaryontische Ribosomen haben einen Durchmesser von etwa 20 nm (200 Å) und bestehen zu 65 % aus rRNA und zu 35 % aus ribosomalen Proteinen. Eukaryotische Ribosomen haben einen Durchmesser von 25 bis 30 nm (250-300 Å) und ein Verhältnis von rRNA zu Protein von nahezu 1. Kristallografische Arbeiten haben gezeigt, dass es keine ribosomalen Proteine in der Nähe der Reaktionsstelle für die Polypeptidsynthese gibt. Dies deutet darauf hin, dass die Proteinkomponenten der Ribosomen nicht direkt an der Katalyse der Peptidbindungsbildung beteiligt sind, sondern dass diese Proteine vielmehr als Gerüst fungieren, das die Fähigkeit der rRNA zur Proteinsynthese verbessern kann (siehe: Ribozym). ⓘ
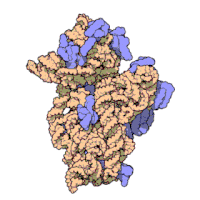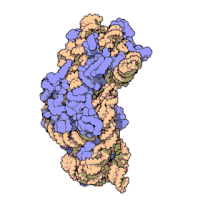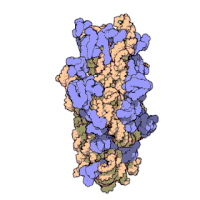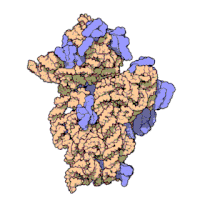
Die ribosomalen Untereinheiten von Prokaryonten und Eukaryonten sind recht ähnlich. ⓘ
Die Maßeinheit, die zur Beschreibung der ribosomalen Untereinheiten und der rRNA-Fragmente verwendet wird, ist die Svedberg-Einheit, ein Maß für die Sedimentationsrate bei der Zentrifugation und nicht für die Größe. Dies erklärt, warum die Namen der Fragmente nicht übereinstimmen: Bakterielle 70S-Ribosomen bestehen zum Beispiel aus 50S- und 30S-Untereinheiten. ⓘ
Prokaryoten haben 70S-Ribosomen, die jeweils aus einer kleinen (30S) und einer großen (50S) Untereinheit bestehen. E. coli zum Beispiel hat eine 16S-RNA-Untereinheit (bestehend aus 1540 Nukleotiden), die an 21 Proteine gebunden ist. Die große Untereinheit setzt sich aus einer 5S-RNA-Untereinheit (120 Nukleotide), einer 23S-RNA-Untereinheit (2900 Nukleotide) und 31 Proteinen zusammen. ⓘ
ⓘRibosom von E. coli (ein Bakterium) Ribosom Untereinheit rRNAs r-Proteine 70S 50S 23S (2904 nt) 31 5S (120 nt) 30S 16S (1542 nt) 21
Affinitätsmarker für die tRNA-Bindungsstellen auf dem E. coli-Ribosom ermöglichten die Identifizierung von A- und P-Stellen-Proteinen, die wahrscheinlich mit der Peptidyltransferase-Aktivität assoziiert sind; die markierten Proteine sind L27, L14, L15, L16, L2; zumindest L27 befindet sich an der Donor-Stelle, wie von E. Collatz und A.P. Czernilofsky gezeigt. Weitere Forschungen haben gezeigt, dass die Proteine S1 und S21 in Verbindung mit dem 3′-Ende der 16S ribosomalen RNA an der Initiierung der Translation beteiligt sind. ⓘ
Archäische Ribosomen
Archäische Ribosomen haben die gleichen allgemeinen Abmessungen wie die von Bakterien: ein 70S-Ribosom, das aus einer großen 50S-Untereinheit und einer kleinen 30S-Untereinheit besteht und drei rRNA-Ketten enthält. Auf Sequenzebene sind sie jedoch den eukaryotischen Ribosomen viel ähnlicher als den bakteriellen Ribosomen. Jedes zusätzliche ribosomale Protein, das Archaeen im Vergleich zu Bakterien besitzen, hat ein eukaryotisches Gegenstück, während zwischen Archaeen und Bakterien keine solche Beziehung besteht. ⓘ
Eukaryotische Ribosomen
Eukaryoten haben in ihrem Zytosol 80S-Ribosomen, die jeweils aus einer kleinen (40S) und einer großen (60S) Untereinheit bestehen. Ihre 40S-Untereinheit enthält eine 18S-RNA (1900 Nukleotide) und 33 Proteine. Die große Untereinheit setzt sich aus einer 5S-RNA (120 Nukleotide), einer 28S-RNA (4700 Nukleotide), einer 5,8S-RNA (160 Nukleotide) und 46 Proteinen zusammen. ⓘ
ⓘeukaryotische zytosolische Ribosomen (R. norvegicus) Ribosom Untereinheit rRNAs r-Proteine 80S 60S 28S (4718 nt) 49 5.8S (160 nt) 5S (120 nt) 40S 18S (1874 nt) 33
1977 veröffentlichte Czernilofsky Forschungsarbeiten, in denen er mit Hilfe von Affinitätsmarkierungen tRNA-Bindungsstellen an Rattenleber-Ribosomen identifizierte. Mehrere Proteine, darunter L32/33, L36, L21, L23, L28/29 und L13, befanden sich im oder in der Nähe des Peptidyltransferasezentrums. ⓘ
Plastoribosomen und Mitoribosomen
In Eukaryonten sind Ribosomen in Mitochondrien (manchmal Mitoribosomen genannt) und in Plastiden wie Chloroplasten (auch Plastoribosomen genannt) vorhanden. Sie bestehen ebenfalls aus großen und kleinen Untereinheiten, die mit Proteinen zu einem 70S-Partikel zusammengebunden sind. Diese Ribosomen ähneln denen von Bakterien, und man nimmt an, dass diese Organellen als symbiotische Bakterien entstanden sind. Von den beiden sind die chloroplastischen Ribosomen den bakteriellen näher als die mitochrondrialen. Viele Teile der ribosomalen RNA in den Mitochrondrien sind verkürzt und im Falle der 5S rRNA durch andere Strukturen in Tieren und Pilzen ersetzt. Insbesondere Leishmania tarentolae hat einen minimalen Satz an mitochondrialer rRNA. Im Gegensatz dazu haben pflanzliche Mitoribosomen sowohl eine erweiterte rRNA als auch zusätzliche Proteine im Vergleich zu Bakterien, insbesondere viele Pentatricopetid-Repeat-Proteine. ⓘ
Kryptomonaden und Chlorarachniophyten-Algen können einen Nukleomorphen enthalten, der einem rudimentären eukaryotischen Zellkern ähnelt. Eukaryotische 80S-Ribosomen können in dem Kompartiment vorhanden sein, das das Nukleomorph enthält. ⓘ
Die Unterschiede ausnutzen
Die Unterschiede zwischen bakteriellen und eukaryotischen Ribosomen werden von pharmazeutischen Chemikern ausgenutzt, um Antibiotika zu entwickeln, die eine bakterielle Infektion zerstören können, ohne die Zellen der infizierten Person zu schädigen. Aufgrund ihrer unterschiedlichen Strukturen sind die bakteriellen 70S-Ribosomen für diese Antibiotika anfällig, während die eukaryotischen 80S-Ribosomen dies nicht sind. Obwohl die Mitochondrien ähnliche Ribosomen wie die bakteriellen besitzen, werden sie von diesen Antibiotika nicht angegriffen, da sie von einer Doppelmembran umgeben sind, die diese Antibiotika nicht ohne weiteres in die Organelle eindringen lässt. Ein bemerkenswertes Gegenbeispiel ist jedoch das antineoplastische Antibiotikum Chloramphenicol, das bakterielle 50S- und eukaryotische mitochondriale 50S-Ribosomen erfolgreich hemmt. Was für die Mitochondrien gilt, kann nicht für die Chloroplasten gesagt werden, wo die Antibiotikaresistenz der ribosomalen Proteine ein Merkmal ist, das als Marker in die Gentechnik eingeführt werden soll. ⓘ
Gemeinsame Eigenschaften
Die verschiedenen Ribosomen haben eine gemeinsame Kernstruktur, die trotz der großen Größenunterschiede recht ähnlich ist. Ein großer Teil der RNA ist hochgradig in verschiedenen tertiären Strukturmotiven organisiert, z. B. Pseudoknoten, die eine koaxiale Stapelung aufweisen. Die zusätzliche RNA in den größeren Ribosomen befindet sich in mehreren langen kontinuierlichen Einschüben, so dass sie Schleifen außerhalb der Kernstruktur bilden, ohne diese zu unterbrechen oder zu verändern. Die gesamte katalytische Aktivität des Ribosoms wird von der RNA ausgeführt; die Proteine befinden sich an der Oberfläche und scheinen die Struktur zu stabilisieren. ⓘ
Hochauflösende Struktur

Die allgemeine molekulare Struktur des Ribosoms ist seit den frühen 1970er Jahren bekannt. Anfang der 2000er Jahre wurde die Struktur mit einer hohen Auflösung in der Größenordnung von einigen ångströmen erreicht. ⓘ
Die ersten Arbeiten, die die Struktur des Ribosoms in atomarer Auflösung zeigten, wurden Ende 2000 fast gleichzeitig veröffentlicht. Die 50S-Untereinheit (große prokaryotische Untereinheit) wurde aus dem Archäon Haloarcula marismortui und dem Bakterium Deinococcus radiodurans bestimmt, und die Struktur der 30S-Untereinheit wurde aus Thermus thermophilus bestimmt. Diese Strukturstudien wurden 2009 mit dem Nobelpreis für Chemie ausgezeichnet. Im Mai 2001 wurden diese Koordinaten verwendet, um das gesamte 70S-Partikel von T. thermophilus mit einer Auflösung von 5,5 Å zu rekonstruieren. ⓘ
Im November 2005 wurden zwei Arbeiten mit Strukturen des 70S-Ribosoms von Escherichia coli veröffentlicht. Die Strukturen eines leeren Ribosoms wurden bei einer Auflösung von 3,5 Å mittels Röntgenkristallographie bestimmt. Zwei Wochen später wurde eine auf der Kryo-Elektronenmikroskopie basierende Struktur veröffentlicht, die das Ribosom mit einer Auflösung von 11-15 Å beim Einschleusen eines neu synthetisierten Proteinstrangs in den proteinleitenden Kanal zeigt. ⓘ
Die ersten atomaren Strukturen des Ribosoms im Komplex mit tRNA- und mRNA-Molekülen wurden von zwei Gruppen unabhängig voneinander mit Hilfe der Röntgenkristallographie bei 2,8 Å und 3,7 Å gelöst. Diese Strukturen ermöglichen es, die Einzelheiten der Wechselwirkungen des Ribosoms von Thermus thermophilus mit mRNA und mit tRNAs, die an klassischen ribosomalen Stellen gebunden sind, zu erkennen. Die Wechselwirkungen des Ribosoms mit langen mRNAs, die Shine-Dalgarno-Sequenzen enthalten, wurden kurz darauf mit einer Auflösung von 4,5-5,5 Å sichtbar gemacht. ⓘ
Im Jahr 2011 wurde die erste vollständige atomare Struktur des eukaryotischen 80S-Ribosoms aus der Hefe Saccharomyces cerevisiae durch Kristallographie erhalten. Das Modell offenbart die Architektur der eukaryontenspezifischen Elemente und ihre Interaktion mit dem universell konservierten Kern. Gleichzeitig wurde das vollständige Modell einer eukaryotischen 40S-Ribosomenstruktur in Tetrahymena thermophila veröffentlicht, das die Struktur der 40S-Untereinheit und vieles über die Interaktion der 40S-Untereinheit mit eIF1 während der Translationsinitiierung beschreibt. In ähnlicher Weise wurde auch die Struktur der eukaryotischen 60S-Untereinheit im Komplex mit eIF6 aus Tetrahymena thermophila bestimmt. ⓘ
Funktion
Ribosomen sind winzige Partikel, die aus RNA und assoziierten Proteinen bestehen und für die Synthese von Proteinen zuständig sind. Proteine werden für viele zelluläre Funktionen benötigt, z. B. für die Reparatur von Schäden oder die Steuerung von chemischen Prozessen. Ribosomen können im Zytoplasma schwimmen oder an das endoplasmatische Retikulum gebunden sein. Ihre Hauptaufgabe besteht darin, den genetischen Code in eine Aminosäuresequenz umzuwandeln und Proteinpolymere aus Aminosäuremonomeren aufzubauen. ⓘ
Ribosomen wirken als Katalysatoren in zwei äußerst wichtigen biologischen Prozessen, dem Peptidyltransfer und der Peptidylhydrolyse. Das PT-Zentrum ist für die Herstellung von Proteinbindungen während der Proteinverlängerung verantwortlich. ⓘ
Zusammenfassend lässt sich sagen, dass Ribosomen zwei Hauptfunktionen haben: die Entschlüsselung der Botschaft und die Bildung von Peptidbindungen. Diese beiden Funktionen sind in den ribosomalen Untereinheiten untergebracht. Jede Untereinheit besteht aus einer oder mehreren rRNAs und vielen r-Proteinen. Die kleine Untereinheit (30S in Bakterien und Archaeen, 40S in Eukaryonten) hat die Entschlüsselungsfunktion, während die große Untereinheit (50S in Bakterien und Archaeen, 60S in Eukaryonten) die Bildung von Peptidbindungen katalysiert, was als Peptidyl-Transferase-Aktivität bezeichnet wird. Die kleine Untereinheit der Bakterien (und Archaeen) enthält die 16S rRNA und 21 r-Proteine (Escherichia coli), während die kleine Untereinheit der Eukaryoten die 18S rRNA und 32 r-Proteine enthält (Saccharomyces cerevisiae; die Anzahl variiert jedoch von Art zu Art). Die große bakterielle Untereinheit enthält die 5S- und 23S-rRNAs und 34 r-Proteine (E. coli), während die große eukaryotische Untereinheit die 5S-, 5,8S- und 25S/28S-rRNAs und 46 r-Proteine enthält (S. cerevisiae; auch hier variieren die genauen Zahlen zwischen den Arten). ⓘ
Übersetzung
Ribosomen sind der Arbeitsplatz der Proteinbiosynthese, also des Prozesses der Übersetzung von mRNA in Protein. Die mRNA enthält eine Reihe von Codons, die vom Ribosom entschlüsselt werden, um das Protein herzustellen. Unter Verwendung der mRNA als Vorlage durchläuft das Ribosom jedes Codon (3 Nukleotide) der mRNA und paart es mit der entsprechenden Aminosäure, die von einer Aminoacyl-tRNA bereitgestellt wird. Die Aminoacyl-tRNA enthält an einem Ende ein komplementäres Anticodon und am anderen Ende die entsprechende Aminosäure. Um die passende tRNA schnell und genau zu erkennen, nimmt das Ribosom große Konformationsänderungen vor (konformationelles Korrekturlesen). Die kleine ribosomale Untereinheit, die normalerweise an eine Aminoacyl-tRNA mit der ersten Aminosäure Methionin gebunden ist, bindet an ein AUG-Codon auf der mRNA und rekrutiert die große ribosomale Untereinheit. Das Ribosom enthält drei RNA-Bindungsstellen, die mit A, P und E bezeichnet werden. Die A-Stelle bindet eine Aminoacyl-tRNA oder Terminationsfreisetzungsfaktoren; die P-Stelle bindet eine Peptidyl-tRNA (eine an die Polypeptidkette gebundene tRNA); und die E-Stelle (Ausgang) bindet eine freie tRNA. Die Proteinsynthese beginnt an einem Startcodon AUG in der Nähe des 5'-Endes der mRNA. Die mRNA bindet zuerst an die P-Stelle des Ribosoms. Das Ribosom erkennt das Startcodon anhand der Shine-Dalgarno-Sequenz der mRNA in Prokaryonten und der Kozak-Box in Eukaryonten. ⓘ
An der Katalyse der Peptidbindung ist zwar das C2-Hydroxyl des P-site-Adenosins der RNA in einem Protonen-Shuttle-Mechanismus beteiligt, andere Schritte der Proteinsynthese (wie die Translokation) werden jedoch durch Veränderungen der Proteinkonformationen verursacht. Da ihr katalytischer Kern aus RNA besteht, werden Ribosomen als "Ribozyme" eingestuft, und es wird vermutet, dass sie Überbleibsel der RNA-Welt sind. ⓘ

In Abbildung 5 setzen sich die beiden ribosomalen Untereinheiten (klein und groß) am Startcodon (am 5'-Ende der mRNA) zusammen. Das Ribosom verwendet die tRNA, die dem aktuellen Codon (Triplett) auf der mRNA entspricht, um eine Aminosäure an die Polypeptidkette anzuhängen. Dies geschieht für jedes Triplett auf der mRNA, während sich das Ribosom in Richtung des 3'-Endes der mRNA bewegt. Normalerweise arbeiten in Bakterienzellen mehrere Ribosomen parallel an einer einzigen mRNA und bilden ein so genanntes Polyribosom oder Polysom. ⓘ


Die beiden Hauptzustände des Ribosoms (prä- und posttranslational) sind durch eine hohe Aktivierungsenergie-Barriere voneinander getrennt. Die zentrale Rolle der beiden Elongationsfaktoren besteht darin, diese Energiebarriere zu erniedrigen und so das Ribosom in den jeweils anderen Zustand zu versetzen. ⓘ
Nachdem ein Peptid im Ribosom verknüpft wurde, durchwandert es einen ribosomalen Tunnel. Dieser besteht größtenteils aus rRNA und tritt aus der großen ribosomalen Untereinheit aus. Er hat eine Länge von ca. 100 Å (10 nm) und einen durchschnittlichen Durchmesser von 15 Å (1,5 nm). An dessen engster Stelle wird der Kanal durch zwei konservierte ribosomale Proteine begrenzt, L4e und L22. ⓘ
Cotranslationale Faltung
Es ist bekannt, dass das Ribosom aktiv an der Proteinfaltung beteiligt ist. Die auf diese Weise erhaltenen Strukturen sind in der Regel identisch mit denen, die bei der chemischen Rückfaltung von Proteinen entstehen; die Wege, die zum Endprodukt führen, können jedoch unterschiedlich sein. In einigen Fällen ist das Ribosom entscheidend für die Erlangung der funktionellen Proteinform. Einer der möglichen Mechanismen für die Faltung tief verknoteter Proteine beruht zum Beispiel darauf, dass das Ribosom die Kette durch die angehängte Schleife schiebt. ⓘ
Hinzufügung von übersetzungsunabhängigen Aminosäuren
Das Vorhandensein des Ribosomen-Qualitätskontrollproteins Rqc2 ist mit einer mRNA-unabhängigen Proteinverlängerung verbunden. Diese Dehnung ist das Ergebnis der ribosomalen Addition von CAT-Schwänzen (über tRNAs, die von Rqc2 eingebracht werden): Ribosomen verlängern den C-Terminus eines blockierten Proteins mit zufälligen, translationsunabhängigen Sequenzen von Alaninen und Threoninen. ⓘ
Ribosomen-Standorte
Ribosomen werden entweder als "frei" oder als "membrangebunden" klassifiziert. ⓘ

Freie und membrangebundene Ribosomen unterscheiden sich nur in ihrer räumlichen Verteilung; sie sind in ihrer Struktur identisch. Ob das Ribosom in einem freien oder membrangebundenen Zustand vorliegt, hängt vom Vorhandensein einer ER-targeting Signalsequenz auf dem zu synthetisierenden Protein ab, so dass ein einzelnes Ribosom membrangebunden sein kann, wenn es ein Protein herstellt, aber frei im Zytosol, wenn es ein anderes Protein herstellt. ⓘ
Ribosomen werden manchmal als Organellen bezeichnet, aber die Verwendung des Begriffs Organelle beschränkt sich häufig auf die Beschreibung von subzellulären Komponenten, die eine Phospholipidmembran enthalten, was bei Ribosomen nicht der Fall ist, da sie vollständig aus Partikeln bestehen. Aus diesem Grund werden Ribosomen manchmal auch als "nicht-membranöse Organellen" bezeichnet. ⓘ
Freie Ribosomen
Freie Ribosomen können sich überall im Zytosol bewegen, sind aber vom Zellkern und anderen Organellen ausgeschlossen. Proteine, die von freien Ribosomen gebildet werden, werden in das Zytosol abgegeben und innerhalb der Zelle verwendet. Da das Cytosol hohe Konzentrationen an Glutathion enthält und somit eine reduzierende Umgebung darstellt, können Proteine, die Disulfidbindungen enthalten, die aus oxidierten Cysteinresten gebildet werden, dort nicht hergestellt werden. ⓘ
Membrangebundene Ribosomen
Wenn ein Ribosom beginnt, Proteine zu synthetisieren, die in bestimmten Organellen benötigt werden, kann das Ribosom, das dieses Protein herstellt, "membrangebunden" werden. In eukaryotischen Zellen geschieht dies in einer Region des endoplasmatischen Retikulums (ER), dem so genannten "rauen ER". Die neu produzierten Polypeptidketten werden durch das Ribosom, das die vektorielle Synthese vornimmt, direkt in das ER eingefügt und dann über den sekretorischen Weg an ihren Bestimmungsort transportiert. Gebundene Ribosomen produzieren in der Regel Proteine, die innerhalb der Plasmamembran verwendet oder durch Exozytose aus der Zelle ausgestoßen werden. ⓘ
Biogenese
In Bakterienzellen werden die Ribosomen im Zytoplasma durch die Transkription mehrerer Ribosomengen-Operons synthetisiert. Bei Eukaryonten findet der Prozess sowohl im Zytoplasma der Zelle als auch im Nukleolus, einem Bereich innerhalb des Zellkerns, statt. Der Zusammenbauprozess umfasst die koordinierte Funktion von über 200 Proteinen bei der Synthese und Verarbeitung der vier rRNAs sowie den Zusammenbau dieser rRNAs mit den ribosomalen Proteinen. ⓘ
Ursprung
Der Ursprung der Ribosomen wird in der RNA-Welt vermutet, in der ein selbstreplizierender Komplex erst später die Fähigkeit zur Proteinsynthese entwickelte, als dafür ausreichend Aminosäuren zur Verfügung standen. Die katalytischen Fähigkeiten der RNA (Ribozym) sind ein zentraler Bestandteil der RNA-Welt-Hypothese. Untersuchungen legen nahe, dass diese Ribosomen-Vorläufer, die ausschließlich aus rRNA aufgebaut waren, die Fähigkeit zur Bildung von Peptidbindungen entwickelt haben könnten. Darüber hinaus gibt es eine starke Evidenz, dass ursprüngliche Ribosomen selbstreplizierende Komplexe waren, in denen die rRNA informationelle, strukturelle und katalytische Zwecke hatte, da sie tRNA und Proteine für die ribosomale Selbstreplikation codiert haben könnte. Die hypothetischen DNA-freien zellulären Organismen, die mit solcher selbstreplizierender RNA ausgestattet waren, nennt man Ribozyten. ⓘ
Als sich in der RNA-Welt unter noch präbiotischen Bedingungen allmählich Aminosäuren anreicherten, könnte ihre Wechselwirkung mit der katalytischen RNA sowohl deren Wirkungsbereich als auch ihre Effizienz erhöht haben. Der selektive Druck, Proteine in die selbstreplizierenden Mechanismen der Ribosomen einzubauen, könnte die treibende Kraft für die Evolution der Ribosomen von einer ursprünglich selbstreplizierenden Maschine in ihre heutige Form als Translationsmaschine gewesen sein, da dies die Kapazität der Selbstreplikation erhöht hätte. ⓘ
Heterogene Ribosomen
Die Zusammensetzung der Ribosomen ist von Spezies zu Spezies und sogar innerhalb ein und derselben Zelle heterogen, wie die Existenz von zytoplasmatischen und mitochondrialen Ribosomen in ein und derselben eukaryontischen Zelle zeigt. Einige Forscher haben vorgeschlagen, dass die Heterogenität in der Zusammensetzung der ribosomalen Proteine bei Säugetieren für die Genregulation wichtig ist, d. h. die Hypothese der spezialisierten Ribosomen. Diese Hypothese ist jedoch umstritten und Gegenstand laufender Forschungen. ⓘ
Vince Mauro und Gerald Edelman schlugen erstmals vor, dass die Heterogenität der Ribosomenzusammensetzung an der Translationskontrolle der Proteinsynthese beteiligt ist. Sie schlugen die Ribosomenfilter-Hypothese vor, um die regulatorischen Funktionen der Ribosomen zu erklären. Es gibt Hinweise darauf, dass spezialisierte Ribosomen, die für verschiedene Zellpopulationen spezifisch sind, die Art und Weise der Translation von Genen beeinflussen können. Einige ribosomale Proteine werden aus dem zusammengebauten Komplex mit zytosolischen Kopien ausgetauscht, was darauf hindeutet, dass die Struktur des In-vivo-Ribosoms verändert werden kann, ohne dass ein völlig neues Ribosom synthetisiert werden muss. ⓘ
Bestimmte ribosomale Proteine sind für das zelluläre Leben absolut entscheidend, andere dagegen nicht. In der Hefe sind 14/78 ribosomale Proteine nicht essentiell für das Wachstum, während dies beim Menschen von der untersuchten Zelle abhängt. Andere Formen der Heterogenität sind posttranslationale Modifikationen an ribosomalen Proteinen wie Acetylierung, Methylierung und Phosphorylierung. Arabidopsis, Virale interne Ribosomen-Eintrittsstellen (IRESs) können die Übersetzung durch unterschiedlich zusammengesetzte Ribosomen vermitteln. So sind beispielsweise 40S-Ribosomeneinheiten ohne eS25 in Hefe- und Säugetierzellen nicht in der Lage, die CrPV IGR IRES zu rekrutieren. ⓘ
Die Heterogenität der ribosomalen RNA-Modifikationen spielt eine wichtige Rolle bei der Strukturerhaltung und/oder Funktion, und die meisten mRNA-Modifikationen befinden sich in hochkonservierten Regionen. Die häufigsten rRNA-Modifikationen sind die Pseudouridylierung und die 2'-O-Methylierung der Ribose. ⓘ
Aufbau und Arten
Eukaryotische Ribosomen
In eukaryotischen Zellen befinden sich die Ribosomen im Cytoplasma (nicht im Karyoplasma des Zellkerns). Daneben kommen spezielle Ribosomen auch in einigen Organellen vor, wenn diese über eigene DNA verfügen:
- in den Mitochondrien (oder ersatzweise in Hydrogenosomen, wenn mit DNA), sowie
- in (fast allen) Chloroplasten und anderen Plastiden, falls – wie bei den Pflanzen – vorhanden. ⓘ
Mitoribosomen und Plastoribosomen
Die Ribosomen aus Mitochondrien und Chloroplasten sind den prokaryotischen Ribosomen ähnlich, was die Endosymbiontenhypothese stützt. Die mitochondrialen Ribosomen des Menschen und anderen Säugern bestehen aus vielen Proteinen, von denen 21 nur in Mitochondrien vorkommen, und erzeugen nur mitochondriale Membranproteine. ⓘ
80S-Ribosomen in komplexen Plastiden
Im Gegensatz dazu können die komplexen Plastiden etwa der Chlorarachniophyten mit einem zusätzlichen Zellkern (Nucleomorph) eigene eukaryotische 80S-Ribosomen enthalten. Die komplexen Plastiden werden als Resultat einer sekundären Endosymbiose gedeutet (sekundäre Plastiden). ⓘ
