Nukleotide
| Teil einer Serie über ⓘ |
| Genetik |
|---|
 |
|

Nukleotide sind organische Moleküle, die aus einem Nukleosid und einem Phosphat bestehen. Sie dienen als monomere Einheiten der Nukleinsäurepolymere Desoxyribonukleinsäure (DNA) und Ribonukleinsäure (RNA), die beide wesentliche Biomoleküle in allen Lebensformen auf der Erde sind. Nukleotide werden mit der Nahrung aufgenommen und auch von der Leber aus gängigen Nährstoffen synthetisiert. ⓘ
Nukleotide bestehen aus drei Untereinheiten: einer Nukleobase, einem Fünf-Kohlenstoff-Zucker (Ribose oder Desoxyribose) und einer Phosphatgruppe, die aus ein bis drei Phosphaten besteht. Die vier Nukleobasen in der DNA sind Guanin, Adenin, Cytosin und Thymin; in der RNA wird Uracil anstelle von Thymin verwendet. ⓘ
Nukleotide spielen auch eine zentrale Rolle im Stoffwechsel auf einer grundlegenden, zellulären Ebene. Sie liefern chemische Energie - in Form der Nukleosidtriphosphate Adenosintriphosphat (ATP), Guanosintriphosphat (GTP), Cytidintriphosphat (CTP) und Uridintriphosphat (UTP) - in der gesamten Zelle für die zahlreichen Zellfunktionen, die Energie benötigen, wie z. B. die Aminosäure-, Protein- und Zellmembransynthese, die Bewegung der Zelle und von Zellteilen (sowohl intern als auch interzellulär), die Zellteilung usw. Darüber hinaus sind Nucleotide an der Zellsignalisierung beteiligt (zyklisches Guanosinmonophosphat oder cGMP und zyklisches Adenosinmonophosphat oder cAMP) und werden in wichtige Cofaktoren enzymatischer Reaktionen eingebaut (z. B. Coenzym A, FAD, FMN, NAD und NADP+). ⓘ
In der experimentellen Biochemie können Nukleotide mit Hilfe von Radionukliden radiomarkiert werden, um Radionukleotide zu erhalten. ⓘ
5-Nukleotide werden auch in Geschmacksverstärkern als Lebensmittelzusatzstoff verwendet, um den Umami-Geschmack zu verstärken, häufig in Form eines Hefeextrakts. ⓘ
Als Nukleotide, auch Nucleotide (abgekürzt nt), werden die Bausteine von Nukleinsäuren sowohl in Strängen der Ribonukleinsäure (RNA bzw. deutsch RNS) wie auch der Desoxyribonukleinsäure (DNA bzw. deutsch DNS) bezeichnet. Ein Nukleotid setzt sich aus einem Basen-, einem Zucker- und einem Phosphatanteil zusammen. ⓘ

Ein Nukleosid ist die Verbindung einer Nukleinbase (Base) mit einem Einfachzucker, einer Pentose. Deren 2'-Rest (R) ist im Falle der Ribose eine Hydroxygruppe (OH-), im Falle der Desoxyribose hingegen Wasserstoff (H-).
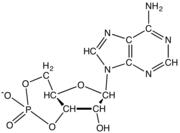
Bei einem Nukleotid ist die 5'-OH-Gruppe der Pentose eines Nukleosids mit einem Phosphatrest verestert. Ein Nukleosidtriphosphat (NTP) weist drei Phosphatgruppen auf, die untereinander Säureanhydridbindungen ausbilden. Mit Adenin als Base und Ribose als Saccharid liegt das Adenosintriphosphat (ATP) vor.
Bei artifiziellen Didesoxynukleotiden ist auch die 3'-OH-Gruppe durch ein H-Atom ersetzt. ⓘ
Nukleotide treten nicht nur als Monophosphate (NMP) verknüpft in den informationstragenden Makromolekülen von Nukleinsäuren auf. Sie tragen auch weitere Funktionen für die Regulation von Lebensvorgängen in Zellen. So spielen Triphosphate (NTP) wie beispielsweise Adenosintriphosphat (ATP) eine zentrale Rolle beim Energietransfer zwischen Stoffwechselwegen, als Cofaktor für die Aktivität von Enzymen, für den Transport durch Motorproteine oder die Kontraktion von Muskelzellen. Guanosintriphosphat (GTP) bindende (G-)Proteine übermitteln Signale von Membranrezeptoren, das cyclische Adenosinmonophosphat (cAMP) ist ein wichtiger intrazellulärer Botenstoff. ⓘ
Struktur
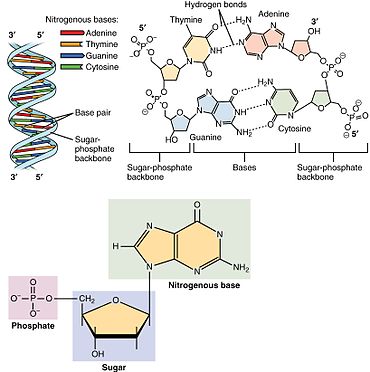
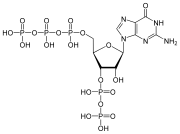
Ein Nukleotid besteht aus drei verschiedenen chemischen Untereinheiten: einem Zuckermolekül mit fünf Kohlenstoffatomen, einer Nukleobase - beide zusammen werden als Nukleosid bezeichnet - und einer Phosphatgruppe. Wenn alle drei miteinander verbunden sind, wird ein Nukleotid auch als "Nukleosidmonophosphat", "Nukleosiddiphosphat" oder "Nukleosidtriphosphat" bezeichnet, je nachdem, aus wie vielen Phosphaten die Phosphatgruppe besteht. ⓘ
In Nukleinsäuren enthalten Nukleotide entweder eine Purin- oder eine Pyrimidinbase - d. h. das Nukleobasenmolekül, das auch als stickstoffhaltige Base bezeichnet wird - und werden als Ribonukleotide bezeichnet, wenn der Zucker Ribose ist, oder als Desoxyribonukleotide, wenn der Zucker Desoxyribose ist. Einzelne Phosphatmoleküle verbinden wiederholt die Zuckerringmoleküle in zwei benachbarten Nukleotidmonomeren und verbinden so die Nukleotidmonomere einer Nukleinsäure Ende an Ende zu einer langen Kette. Diese Kettenverbindungen von Zucker- und Phosphatmolekülen bilden das "Rückgrat" eines Strangs für eine Einfach- oder Doppelhelix. In einem beliebigen Strang verläuft die chemische Ausrichtung (Direktionalität) der Kettenverbindungen vom 5'-Ende zum 3'-Ende (lies: 5 prime-end to 3 prime-end), was sich auf die fünf Kohlenstoffstellen auf Zuckermolekülen in benachbarten Nukleotiden bezieht. In einer Doppelhelix sind die beiden Stränge in entgegengesetzte Richtungen ausgerichtet, was eine Basenpaarung und Komplementarität zwischen den Basenpaaren ermöglicht, was für die Replikation oder Transkription der in der DNA kodierten Informationen unerlässlich ist. ⓘ
Nukleinsäuren sind also polymere Makromoleküle, die aus Nukleotiden, den Monomereinheiten der Nukleinsäuren, aufgebaut sind. Die Purinbasen Adenin und Guanin sowie die Pyrimidinbase Cytosin kommen sowohl in der DNA als auch in der RNA vor, während die Pyrimidinbasen Thymin (in der DNA) und Uracil (in der RNA) nur in einem der beiden vorkommen. Adenin bildet mit Thymin ein Basenpaar mit zwei Wasserstoffbrücken, während Guanin mit Cytosin ein Paar mit drei Wasserstoffbrücken bildet. ⓘ
Einzelne Nukleotide sind nicht nur Bausteine für den Aufbau von Nukleinsäurepolymeren, sondern spielen auch eine Rolle bei der zellulären Energiespeicherung und -bereitstellung, bei der zellulären Signalübertragung, als Quelle von Phosphatgruppen, die zur Modulation der Aktivität von Proteinen und anderen Signalmolekülen verwendet werden, und als enzymatische Cofaktoren, die häufig Redoxreaktionen durchführen. Zyklische Signalnukleotide werden gebildet, indem die Phosphatgruppe zweimal an dasselbe Zuckermolekül gebunden wird und die 5'- und 3'-Hydroxylgruppen des Zuckers überbrückt werden. Einige Signalnukleotide weichen von der Standardkonfiguration mit nur einer Phosphatgruppe ab, da sie mehrere Phosphatgruppen an verschiedenen Positionen des Zuckers aufweisen. Zu den Nukleotid-Cofaktoren gehört ein breiteres Spektrum chemischer Gruppen, die über die glykosidische Bindung an den Zucker gebunden sind, darunter Nikotinamid und Flavin, und im letzteren Fall ist der Ribosezucker linear und bildet nicht den Ring wie bei anderen Nukleotiden. ⓘ

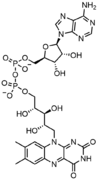
FAD, ein enzymatischer Dinukleotid-Cofaktor, bei dem einer der Ribosezucker eine lineare Konfiguration statt eines Rings annimmt. ⓘ
Synthese
Nukleotide können auf verschiedene Weise sowohl in vitro als auch in vivo synthetisiert werden. ⓘ
In vitro können bei der Laborherstellung von Nukleotiden Schutzgruppen verwendet werden. Ein gereinigtes Nukleosid wird geschützt, um ein Phosphoramidit zu bilden, das dann verwendet werden kann, um in der Natur nicht vorkommende Analoga zu erhalten und/oder ein Oligonukleotid zu synthetisieren. ⓘ
In vivo können Nukleotide de novo synthetisiert oder durch Rückgewinnungswege recycelt werden. Die bei der De-novo-Synthese von Nukleotiden verwendeten Komponenten stammen aus biosynthetischen Vorläufern des Kohlenhydrat- und Aminosäurenstoffwechsels sowie aus Ammoniak und Kohlendioxid. Kürzlich wurde auch gezeigt, dass der zelluläre Bikarbonat-Stoffwechsel durch mTORC1-Signale reguliert werden kann. Die Leber ist das wichtigste Organ für die De-novo-Synthese aller vier Nukleotide. Die De-novo-Synthese von Pyrimidinen und Purinen erfolgt auf zwei verschiedenen Wegen. Pyrimidine werden zunächst aus Aspartat und Carbamoylphosphat im Zytoplasma zu der gemeinsamen Vorläufer-Ringstruktur Orotsäure synthetisiert, an die eine phosphorylierte Ribosyleinheit kovalent gebunden wird. Purine hingegen werden zunächst aus der Zuckervorlage synthetisiert, an der die Ringsynthese stattfindet. Die Synthese der Purin- und Pyrimidin-Nukleotide wird von mehreren Enzymen im Zytoplasma der Zelle durchgeführt, nicht in einer bestimmten Organelle. Die Nukleotide werden abgebaut, so dass nützliche Teile in Synthesereaktionen wiederverwendet werden können, um neue Nukleotide herzustellen. ⓘ
Pyrimidin-Ribonukleotid-Synthese
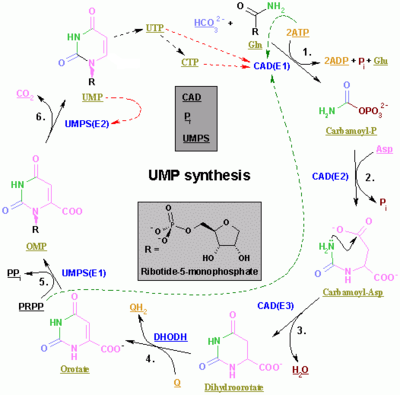
Die Synthese der Pyrimidine CTP und UTP erfolgt im Zytoplasma und beginnt mit der Bildung von Carbamoylphosphat aus Glutamin und CO2. Anschließend katalysiert die Aspartat-Carbamoyltransferase eine Kondensationsreaktion zwischen Aspartat und Carbamoylphosphat zur Bildung von Carbamoyl-Asparaginsäure, die durch Dihydroorotase zu 4,5-Dihydroorotinsäure cyclisiert wird. Letztere wird durch Dihydroorotat-Oxidase in Orotat umgewandelt. Die Nettoreaktion ist:
- (S)-Dihydroorotat + O2 → Orotat + H2O2 ⓘ
Orotat ist kovalent mit einer phosphorylierten Ribosyl-Einheit verbunden. Die kovalente Bindung zwischen der Ribose und dem Pyrimidin erfolgt an der Position C1 der Riboseeinheit, die ein Pyrophosphat enthält, und N1 des Pyrimidinrings. Die Orotat-Phosphoribosyltransferase (PRPP-Transferase) katalysiert die Nettoreaktion unter Bildung von Orotidinmonophosphat (OMP):
- Orotat + 5-Phospho-α-D-ribose-1-diphosphat (PRPP) → Orotidin-5'-phosphat + Pyrophosphat ⓘ
Orotidin-5'-monophosphat wird von der Orotidin-5'-phosphat-Decarboxylase unter Bildung von Uridinmonophosphat (UMP) decarboxyliert. Die PRPP-Transferase katalysiert sowohl die Ribosylierungs- als auch die Decarboxylierungsreaktion und bildet UMP aus Orotsäure in Gegenwart von PRPP. Aus UMP werden andere Pyrimidin-Nukleotide abgeleitet. UMP wird von zwei Kinasen über zwei aufeinander folgende Reaktionen mit ATP zu Uridintriphosphat (UTP) phosphoryliert. Zunächst wird das Diphosphat aus UDP hergestellt, das wiederum zu UTP phosphoryliert wird. Beide Schritte werden durch ATP-Hydrolyse angekurbelt:
- ATP + UMP → ADP + UDP ⓘ
- UDP + ATP → UTP + ADP ⓘ
CTP wird anschließend durch Aminierung von UTP mit Hilfe der katalytischen Aktivität der CTP-Synthetase gebildet. Glutamin ist der NH3-Donor, und die Reaktion wird auch durch ATP-Hydrolyse angeheizt:
- UTP + Glutamin + ATP + H2O → CTP + ADP + Pi ⓘ
Cytidinmonophosphat (CMP) wird aus Cytidintriphosphat (CTP) unter Verlust von zwei Phosphaten gebildet. ⓘ
Synthese von Purin-Ribonukleotiden
Die Atome, die zum Aufbau der Purinnukleotide verwendet werden, stammen aus verschiedenen Quellen: ⓘ

 |
Die biosynthetischen Ursprünge der Purinringatome N1 entsteht aus der Amingruppe von Asp C2 und C8 stammen von Formiat N3 und N9 werden von der Amidgruppe von Gln beigesteuert C4, C5 und N7 werden von Gly abgeleitet C6 stammt von HCO3- (CO2) ⓘ |
Die De-novo-Synthese der Purinnukleotide, bei der diese Vorstufen in den Purinring eingebaut werden, verläuft über einen 10-stufigen Weg bis zum Verzweigungs-Zwischenprodukt IMP, dem Nukleotid der Base Hypoxanthin. AMP und GMP werden anschließend über separate, zweistufige Wege aus diesem Zwischenprodukt synthetisiert. Die Purineinheiten werden also zunächst als Teil der Ribonukleotide und nicht als freie Basen gebildet. ⓘ
An der IMP-Synthese sind sechs Enzyme beteiligt. Drei von ihnen sind multifunktionell:
- GART (Reaktionen 2, 3 und 5)
- PAICS (Reaktionen 6, und 7)
- ATIC (Reaktionen 9 und 10) ⓘ
Der Stoffwechselweg beginnt mit der Bildung von PRPP. PRPS1 ist das Enzym, das R5P, das hauptsächlich über den Pentosephosphatweg gebildet wird, durch Reaktion mit ATP zu PRPP aktiviert. Die Reaktion ist insofern ungewöhnlich, als eine Pyrophosphorylgruppe direkt von ATP auf C1 von R5P übertragen wird und das Produkt die α-Konfiguration um C1 hat. Diese Reaktion wird auch bei den Synthesewegen von Trp, His und den Pyrimidinnukleotiden durchgeführt. Da sich diese Reaktion an einem wichtigen metabolischen Knotenpunkt befindet und viel Energie benötigt, ist sie stark reguliert. ⓘ
In der ersten Reaktion, die nur in der Purinnukleotidbiosynthese vorkommt, katalysiert PPAT die Verdrängung der Pyrophosphatgruppe (PPi) von PRPP durch einen Amidstickstoff, der entweder von Glutamin (N), Glycin (N&C), Aspartat (N), Folsäure (C1) oder CO2 stammt. Dies ist der entscheidende Schritt in der Purinsynthese. Die Reaktion erfolgt mit der Umkehrung der Konfiguration um Ribose C1, wodurch β-5-Phosphorybosylamin (5-PRA) gebildet wird und die anomere Form des zukünftigen Nukleotids entsteht. ⓘ
Anschließend wird durch ATP-Hydrolyse ein Glycin eingebaut, und die Carboxylgruppe bildet eine Aminbindung mit dem zuvor eingeführten NH2. Anschließend wird eine Ein-Kohlenstoff-Einheit des Folsäure-Coenzyms N10-Formyl-THF an die Aminogruppe des substituierten Glycins angehängt und der Imidazolring geschlossen. Anschließend wird eine zweite NH2-Gruppe von Glutamin auf den ersten Kohlenstoff der Glycineinheit übertragen. Gleichzeitig wird eine Carboxylierung des zweiten Kohlenstoffs der Glycineinheit hinzugefügt. Dieser neue Kohlenstoff wird durch die Hinzufügung einer dritten NH2-Einheit modifiziert, die diesmal von einem Aspartatrest übertragen wird. Schließlich wird eine zweite Ein-Kohlenstoff-Einheit aus Formyl-THF an die Stickstoffgruppe angehängt, und der Ring wird kovalent geschlossen, um die gemeinsame Purin-Vorstufe Inosinmonophosphat (IMP) zu bilden. ⓘ
Inosinmonophosphat wird in zwei Schritten in Adenosinmonophosphat umgewandelt. Zunächst wird durch die GTP-Hydrolyse die Addition von Aspartat an IMP durch die Adenylosuccinat-Synthase angekurbelt, wobei der Carbonylsauerstoff durch einen Stickstoff ersetzt wird und das Zwischenprodukt Adenylosuccinat entsteht. Anschließend wird Fumarat unter Bildung von Adenosinmonophosphat abgespalten. Dieser Schritt wird von der Adenylosuccinat-Lyase katalysiert. ⓘ
Inosinmonophosphat wird durch Oxidation von IMP unter Bildung von Xanthylat in Guanosinmonophosphat umgewandelt, gefolgt von der Einfügung einer Aminogruppe an C2. NAD+ ist der Elektronenakzeptor bei dieser Oxidationsreaktion. Die Übertragung der Amidgruppe von Glutamin wird durch ATP-Hydrolyse angekurbelt. ⓘ
Abbau von Pyrimidinen und Purinen
Beim Menschen können die Pyrimidinringe (C, T, U) vollständig zu CO2 und NH3 abgebaut werden (Harnstoffausscheidung). Die Purinringe (G, A) können dies jedoch nicht. Stattdessen werden sie zu der stoffwechselmäßig trägen Harnsäure abgebaut, die dann aus dem Körper ausgeschieden wird. Harnsäure wird gebildet, wenn GMP in die Basen Guanin und Ribose gespalten wird. Guanin wird zu Xanthin desaminiert, das wiederum zu Harnsäure oxidiert wird. Diese letzte Reaktion ist irreversibel. In ähnlicher Weise kann Harnsäure gebildet werden, wenn AMP zu IMP desaminiert wird, von dem die Riboseeinheit entfernt wird, um Hypoxanthin zu bilden. Hypoxanthin wird zu Xanthin und schließlich zu Harnsäure oxidiert. Anstelle der Harnsäureausscheidung können Guanin und IMP in Gegenwart von PRPP und Aspartat (NH3-Donor) für Recyclingzwecke und die Nukleinsäuresynthese verwendet werden. ⓘ
Präbiotische Synthese von Nukleotiden
Theorien über den Ursprung des Lebens erfordern die Kenntnis der chemischen Wege, die die Bildung der wichtigsten Bausteine des Lebens unter plausiblen präbiotischen Bedingungen ermöglichen. Die RNA-Welt-Hypothese geht davon aus, dass in der Ursuppe frei schwimmende Ribonukleotide existierten, die grundlegenden Moleküle, die sich in Reihe zu RNA verbinden. Komplexe Moleküle wie die RNA müssen aus kleinen Molekülen entstanden sein, deren Reaktivität durch physikalisch-chemische Prozesse gesteuert wurde. Die RNA besteht aus Purin- und Pyrimidin-Nukleotiden, die beide für eine zuverlässige Informationsübertragung und damit für die darwinistische Evolution notwendig sind. Becker et al. zeigten, wie Pyrimidin-Nukleoside aus kleinen Molekülen und Ribose synthetisiert werden können, und zwar ausschließlich durch Nass-Trocken-Zyklen. Purinnukleoside können über einen ähnlichen Weg synthetisiert werden. 5'-Mono- und Di-Phosphate werden ebenfalls selektiv aus phosphathaltigen Mineralien gebildet, was die gleichzeitige Bildung von Polyribonukleotiden mit Purin- und Pyrimidinbasen ermöglicht. So kann ein Reaktionsnetzwerk zu den Purin- und Pyrimidin-RNA-Bausteinen ausgehend von einfachen atmosphärischen oder vulkanischen Molekülen aufgebaut werden. ⓘ
Unnatürliches Basenpaar (UBP)
Ein unnatürliches Basenpaar (UBP) ist eine konstruierte Untereinheit (oder Nukleobase) der DNA, die in einem Labor hergestellt wird und in der Natur nicht vorkommt. Beispiele hierfür sind d5SICS und dNaM. Diese künstlichen Nukleotide, die hydrophobe Nukleobasen tragen, weisen zwei verschmolzene aromatische Ringe auf, die in der DNA einen (d5SICS-dNaM)-Komplex oder ein Basenpaar bilden. E. coli wurde dazu veranlasst, ein Plasmid, das UBPs enthält, über mehrere Generationen zu replizieren. Dies ist das erste bekannte Beispiel dafür, dass ein lebender Organismus einen erweiterten genetischen Code an nachfolgende Generationen weitergibt. ⓘ
Medizinische Anwendungen von synthetischen Nukleotiden
Mehrere Nukleotidderivate wurden als antivirale Mittel gegen Hepatitis und HIV eingesetzt. Tenofovir Disoproxil, Tenofovir Alafenamid und Sofosbuvir sind Beispiele für NRTI, die gegen Hepatitis eingesetzt werden. Bestimmte Arzneimittel wie z. B. Mericitabin, Lamivudin, Entecavir und Telbivudin sind zwar Nukleoside, werden aber durch Phosphorylierung in ihre bioaktiven Nukleotidformen umgewandelt. ⓘ
Längeneinheit
Nukleotid (abgekürzt "nt") ist eine gängige Längeneinheit für einzelsträngige Nukleinsäuren, ähnlich wie das Basenpaar eine Längeneinheit für doppelsträngige Nukleinsäuren ist. ⓘ
Notation von Nukleotiden
Für die Notation der Basen von Nukleotiden in Nukleinsäuresequenzen werden Buchstabensymbole verwendet. Um auch Mehrdeutigkeiten (engl.: ambiguity) unvollständig spezifizierter Nukleobasen berücksichtigen zu können, wurde vom Nomenklaturkomitee der International Union of Biochemistry and Molecular Biology der Ambiguity Code vorgeschlagen:
| Symbol | Bedeutung | Herleitung |
|---|---|---|
| G | G | Guanin |
| A | A | Adenin |
| C | C | Cytosin |
| T | T | Thymin |
| U | U | Uracil |
| R | G oder A | Purine |
| Y | C oder T | Pyrimidine |
| W | A oder T | schwach (engl. weak) mit 2 Wasserstoffbrücken gepaart |
| S | G oder C | stark (engl. strong) mit 3 Wasserstoffbrücken gepaart |
| M | A oder C | Aminogruppe |
| K | G oder T | Ketogruppe |
| H | A, C, oder T (U) | nicht G, im Alphabet folgt H auf G |
| B | G, C, oder T (U) | nicht A, im Alphabet folgt B auf A |
| V | G, A, oder C | nicht T (U), im Alphabet folgt V auf U |
| D | G, A, oder T (U) | nicht C, im Alphabet folgt D auf C |
| N | G, A, C oder T (U) | irgendeine (engl. any) der Basen |
Man beachte den Unterschied zwischen den obigen generischen Symbolen W, S,… und den so genannten Wobble-Basen. Modifikationen der obigen Symbole und auch zusätzliche Symbole gibt es für Nicht-Standard-Basen und modifizierte Basen, etwa den griechischen Buchstaben Psi, Ψ für Pseudouridin (eine Wobble-Base). ⓘ
Etliche der obigen Symbole finden allerdings auch alternativ für synthetische Basen Verwendung, siehe DNA §Synthetische Basen. Eine Auswahl:
- P – 2-Amino-imidazo[1,2-a]-1,3,5-triazin-4(8H)-on und Z – 6-Amino-5-nitro-2(1H)-pyridon
- X – NaM, Y – 5SICS und Y' – TPT3! Weitere Basen aus dieser Serie: FEMO und MMO2
- P – 5-Aza-7-deazaguanin, B – Isoguanin, rS – Isocytosin, dS – 1-Methylcytosin und Z – 6-Amino-5-nitropyridin-2-on, siehe Hachimoji-DNA
- xA, xT, xC, xG (analog mit Präfix xx, y und yy), siehe xDNA ⓘ
Die IUPAC hat die Symbole für Nukleotide festgelegt. Neben den fünf Basen (A, G, C, T/U) werden häufig degenerierte Basen verwendet, insbesondere für den Entwurf von PCR-Primern. Diese Nukleotidcodes sind hier aufgeführt. Einige Primersequenzen können auch das Zeichen "I" enthalten, das für das nicht standardisierte Nukleotid Inosin steht. Inosin kommt in tRNAs vor und kann sich mit Adenin, Cytosin oder Thymin paaren. Dieses Zeichen erscheint jedoch nicht in der folgenden Tabelle, da es keine Entartung darstellt. Inosin kann zwar eine ähnliche Funktion wie die Entartung "D" erfüllen, ist aber ein tatsächliches Nukleotid und keine Darstellung einer Nukleotidmischung, die alle erforderlichen Paarungsmöglichkeiten abdeckt. ⓘ
| Symbol | Beschreibung | Dargestellte Basen ⓘ | ||||
|---|---|---|---|---|---|---|
| A | Adenin | A | 1 | |||
| C | Cytosin | C | ||||
| G | Guanin | G | ||||
| T | Thymin | T | ||||
| U | Uracil | U | ||||
| W | schwach | A | T | 2 | ||
| S | stark | C | G | |||
| M | Amino | A | C | |||
| K | Keto | G | T | |||
| R | Purin | A | G | |||
| Y | Pyrimidin | C | T | |||
| B | nicht A (B kommt nach A) | C | G | T | 3 | |
| D | nicht C (D kommt nach C) | A | G | T | ||
| H | nicht G (H kommt nach G) | A | C | T | ||
| V | nicht T (V kommt nach T und U) | A | C | G | ||
| N | beliebige Basis (keine Lücke) | A | C | G | T | 4 |
Ribose als Zucker
| Nukleobase | Nukleosid | Nukleotid | Strukturformel ⓘ | ||
|---|---|---|---|---|---|
| Adenin | A | Adenosin | Adenosinmonophosphat | AMP | 
|
| Adenosindiphosphat | ADP | 
| |||
| Adenosintriphosphat | ATP | 
| |||
| Guanin | G | Guanosin | Guanosinmonophosphat | GMP | 
|
| Guanosindiphosphat | GDP | 
| |||
| Guanosintriphosphat | GTP | 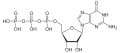
| |||
| Cytosin | C | Cytidin | Cytidinmonophosphat | CMP | 
|
| Cytidindiphosphat | CDP | 
| |||
| Cytidintriphosphat | CTP | 
| |||
| Uracil | U | Uridin | Uridinmonophosphat | UMP | 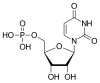
|
| Uridindiphosphat | UDP | 
| |||
| Uridintriphosphat | UTP | 
| |||
Desoxyribose als Zucker
| Nukleobase | Nukleosid | Nukleotid | Strukturformel ⓘ | ||
|---|---|---|---|---|---|
| Adenin | A | Desoxyadenosin | Desoxyadenosinmonophosphat | dAMP | 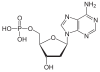
|
| Desoxyadenosindiphosphat | dADP | 
| |||
| Desoxyadenosintriphosphat | dATP | 
| |||
| Guanin | G | Desoxyguanosin | Desoxyguanosinmonophosphat | dGMP | 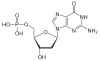
|
| Desoxyguanosindiphosphat | dGDP | 
| |||
| Desoxyguanosintriphosphat | dGTP | 
| |||
| Cytosin | C | Desoxycytidin | Desoxycytidinmonophosphat | dCMP | 
|
| Desoxycytidindiphosphat | dCDP | 
| |||
| Desoxycytidintriphosphat | dCTP | 
| |||
| Thymin | T | Desoxythymidin | Desoxythymidinmonophosphat | dTMP | 
|
| Desoxythymidindiphosphat | dTDP | 
| |||
| Desoxythymidintriphosphat | dTTP | 
| |||
Didesoxyribose als Zucker
Artifizielle Didesoxyribonukleosidtriphosphate (ddNTPs) finden beispielsweise Verwendung bei der DNA-Sequenzierung nach Sanger. ⓘ
Funktionen von Nukleotiden
Neben der Funktion der Nukleotide als Grundbausteine im Polymer von Nukleinsäuren der DNA und RNA, erfüllen Nukleotide weitere Funktionen als einzelne Moleküle, monomer, und spielen so bei der Regulation von Lebensvorgängen eine wichtige Rolle. Beispiele hierfür finden sich zahlreich im Energietransfer zwischen Stoffwechselwegen der Zelle. Monomere Nukleotide treten auch als Cofaktoren von Enzymen auf, so etwa im Coenzym A. ⓘ
Nukleotide lassen sich energetisch nach der Anzahl der Phosphatgruppen unterscheiden als Monophosphate (NMP), Diphosphate (NDP) oder Trisphosphate (NTP) der jeweiligen Nukleoside. Beispielsweise entsteht aus Adenosintriphosphat (ATP) durch Abspaltung eines Phosphatrestes Adenosindiphosphat (ADP), oder durch Abspaltung von Pyrophosphat das energetisch minderwertigere Adenosinmonophosphat (AMP). Das cyclische AMP (cAMP) spielt daneben eine bedeutende Rolle bei der Signaltransduktion in einer Zelle, als sogenannter sekundärer Botenstoff (englisch second messenger). ⓘ